Nanoparticules de silice pour l'administration de protéines intracellulaires :une nouvelle approche de synthèse utilisant la protéine fluorescente verte
Résumé
Dans cette étude, une nouvelle approche pour la préparation de nanoparticules de silice dopées à la protéine fluorescente verte (GFP) avec une distribution de taille étroite est présentée. La GFP a été choisie comme protéine modèle en raison de son autofluorescence. Les nanoparticules dopées aux protéines ont un potentiel d'application élevé dans le domaine de l'administration de protéines intracellulaires. De plus, des particules marquées par fluorescence peuvent être utilisées pour la bio-imagerie. La taille de ces nanoparticules dopées aux protéines a été ajustée de 15 à 35 nm à l'aide d'un processus de synthèse en plusieurs étapes, comprenant la synthèse du cœur des particules suivie des étapes de repousse de la coquille. La GFP a été sélectivement incorporée dans la matrice de silice du noyau ou de l'enveloppe ou des deux par une réaction en un seul pot. Les nanoparticules obtenues ont été caractérisées par la détermination de la taille des particules, du diamètre hydrodynamique, du potentiel , de la fluorescence et du rendement quantique. Les mesures ont montré que la fluorescence de la GFP était maintenue pendant la synthèse des particules. Des expériences d'absorption cellulaire ont démontré que les nanoparticules dopées GFP peuvent être utilisées comme sondes fluorescentes stables et efficaces. L'étude révèle le potentiel de l'approche choisie pour l'incorporation de macromolécules biologiques fonctionnelles dans des nanoparticules de silice, ce qui ouvre de nouveaux domaines d'application comme la livraison de protéines intracellulaires.
Contexte
Ces dernières années, l'encapsulation de protéines dans des micro- et nanoparticules a suscité une grande attention en raison du large potentiel d'application de matériaux tels que les biocapteurs [1] ou les bioréacteurs [2], et plus loin dans les domaines de la livraison contrôlée de protéines [3], livraison de protéines intracellulaires [4] et ingénierie tissulaire [5]. Dans nombre de ces applications, l'activité catalytique des enzymes encapsulées est une fonction de base de ces matériaux. En revanche, les protéines pharmaceutiquement pertinentes, les hormones peptidiques ou les anticorps en tant que cargaison potentielle de tels nanomatériaux exercent leur fonction par liaison spécifique de cibles dans les tissus ou les cellules. Par conséquent, une condition préalable à toutes ces applications est le maintien d'une conformation et d'une fonctionnalité intactes des protéines de cargaison. Les systèmes nanostructurés sont devenus l'un des domaines qui se développent le plus rapidement dans la recherche biomédicale, en raison de leur petite taille, de leur grande surface spécifique et d'autres propriétés uniques [6]. Par conséquent, le développement de nouveaux porteurs de particules pour améliorer la fonctionnalité et la stabilité des systèmes conçus est un sujet important dans le domaine [7]. La matrice de supports nanoparticulaires peut être basée sur des composants biomacromoléculaires ou organiques tels que des glucides, des lipides ou des polymères, formant des systèmes tels que des nanoparticules solides-lipidiques, des liposomes ou des dendrimères. De plus, les systèmes nanostructurés peuvent également être basés sur des matériaux inorganiques comme des métaux ou des oxydes [8]. Tous ces systèmes de matériaux doivent répondre à diverses exigences communes et spécifiques. Tout d'abord, les matériaux de la matrice doivent être biocompatibles afin de faciliter des applications sûres [9]. Deuxièmement, ils doivent être suffisamment stables pour remplir leur fonction de supports tout au long du cycle de vie des systèmes. En outre, ils doivent fournir une capacité de charge et de rétention protéique significative ainsi qu'une libération contrôlée de protéines [10].
Outre la fixation des protéines à la surface des nano-objets par adsorption ou liaison covalente [11], les protéines peuvent être piégées dans des nanostructures, améliorant ainsi leur stabilité et leur activité enzymatique [2]. Le nanopiégeage peut être réalisé par hydrolyse et condensation d'un précurseur de silice via un traitement sol-gel [12] ou via des approches de microémulsion eau-dans-huile, provoquant la polymérisation de l'enzyme entourant la coque à l'interface eau-huile [13]. Dans ces méthodes, le piégeage des protéines peut se produire par deux approches chimiques différentes, en utilisant des processus de liaison covalents ou non covalents [14]. En particulier, le dioxyde de silicium amorphe est un matériau porteur prometteur pour les protéines, en raison de sa biocompatibilité élevée, de son inertie et de sa stabilité mécanique [15]. Diverses voies, en particulier des approches biomimétiques pour l'encapsulation d'enzymes dans le dioxyde de silicium ont été suivies [2, 16], le profil de libération des enzymes étant contrôlé par des réactions chimiques du lieur ou la dégradation de la matrice de silice. Des matériaux mésoporeux ont également été utilisés comme matrice pour immobiliser des enzymes dans des pores de 2 à 50 nm [13, 17]. La libération de la cargaison des nanoparticules mésoporeuses peut être ajustée en utilisant la stratégie « gatekeeper » ou en modifiant la surface interne des pores pour contrôler l'affinité de liaison avec les médicaments [10b]. Néanmoins, la taille des pores peut limiter le chargement d'enzymes dans les échafaudages de silice mésoporeux réalisés [18], c'est pourquoi de nouvelles stratégies sont récemment à l'étude pour l'administration de protéines.
Les nanoparticules de silice étant largement utilisées pour la bio-imagerie [19], l'incorporation de protéines fluorescentes constitue une option pour la génération de sondes fluorescentes biocompatibles. Par exemple, l'incorporation de protéine fluorescente verte (GFP) dans des nanoparticules de silice via des techniques d'émulsion inverse a été décrite dans la littérature [20]. Ces études indiquent que l'incorporation de GFP dans la matrice de particules de silice améliore non seulement l'intensité de fluorescence de la protéine mais également sa stabilité thermique, sa stabilité contre la dénaturation chimique et le traitement par la protéase. Néanmoins, la méthode est moins adaptée à la synthèse de nanoparticules de silice bien définies dans la gamme nanométrique inférieure avec une distribution de taille étroite. De plus, les conditions de synthèse incluent le contact avec des tensioactifs, des alcools ou des bases hautement alcalines ainsi que des températures élevées qui peuvent toutes ne pas être compatibles avec l'incorporation de protéines sensibles [20, 21].
Par conséquent, nous rapportons une nouvelle approche pour la préparation de nanoparticules de silice dopées aux protéines, en utilisant la GFP comme protéine modèle. À cette fin, nous avons utilisé une synthèse en un seul pot dans des conditions de synthèse douces (température ambiante, faible salinité) suivie d'étapes de dialyse pour la purification. L'approche est caractérisée par son potentiel à préparer des nanoparticules de silice piégées dans des protéines présentant une distribution de taille étroite dans le régime de taille inférieur à 50 nm.
Méthodes
Matériaux
Tous les produits chimiques ont été utilisés tels qu'ils ont été achetés auprès de Sigma-Aldrich (Taufkirchen, Allemagne) et sans autre purification. Pour toutes les étapes de synthèse et de purification, de l'eau ultrapure (18,2 MΩ, système de purification d'eau Milli-Q de type ELIX 20, Millipore Corp., États-Unis) a été utilisée.
Préparation de GFP
La GFP a été obtenue par expression protéique et purification ultérieure comme décrit ailleurs [22]. En bref, la GFP comprenant une étiquette His6 N-terminale a été exprimée en utilisant un vecteur d'expression bactérien de haut niveau basé sur le système de vecteur pQE (Qiagen, Hilden, Allemagne) dans E. coli XL1-Blue et purifié par chromatographie d'affinité chargée Ni (Qiagen, Hilden, Allemagne). Par la suite, la protéine a été transférée dans un dispositif concentrateur (membrane de coupure de poids moléculaire (MWCO) de 3 kDa, Pall, Dreieich, Allemagne) pour un échange de tampon. La GFP a été lavée trois fois par addition de 15 mL de solution de -arginine et de bicarbonate de sodium, respectivement, puis récupérée dans 3 mL de la solution de -arginine/bicarbonate de sodium. Après cela, la suspension GFP a été filtrée dans des tubes stériles à travers des filtres stériles en acétate de cellulose de 0,22 um (Carl Roth, Karlsruhe, Allemagne). Avant utilisation, la concentration en protéines a été ajustée à 1 mg mL −1 soit dans 7,2 mmol L −1 ʟ-arginine (pH = 10,3) ou 10,0 mmol L −1 NaHCO3 (pH = 9.2) solution.
Synthèses et purification de nanoparticules
Les nanoparticules de silice ont été préparées selon un protocole modifié décrit précédemment [23]. Brièvement, le tétraéthoxysilane (TEOS), utilisé comme précurseur non polaire, a été hydrolysé dans un système biphasique eau/cyclohexane médié par la catalyse ʟ-arginine.
Préparation des particules de base
Dans un ballon à fond rond à trois cols, 91 mg (0,52 mmol) de ʟ-arginine ont été dissous dans 69 mL d'eau, avant que 4,5 mL de cyclohexane ne soient ajoutés comme couche supérieure. Le mélange réactionnel a été chauffé à 40°C sous agitation. Après addition de 5,5 mL (24,63 mmol) de TEOS, le mélange a été maintenu dans ces conditions pendant 20 h supplémentaires.
Couches de coquille de silice
Pour les étapes de croissance de la coquille ultérieures, soit les particules de noyau, soit les particules résultant de la première étape de croissance de la coquille ont été utilisées. Pour la croissance de la coquille, 14 mg (0,08 mmol) de ʟ-arginine ont été dissous dans 36 ml d'eau et 10 ml de dispersions de particules préalablement préparées ont été ajoutés. Après addition de 5 ml de cyclohexane, le mélange a été chauffé à 40°C. Après addition de 3,52 mL (15,8 mmol) de TEOS, le mélange a été agité pendant 20 h supplémentaires.
Préparation de nanoparticules dopées GFP. Pour la préparation de nanoparticules dopées GFP, 30 min après l'ajout de TEOS, 200 μg (6,9 nmol) de GFP ont été ajoutés.
Purification des particules
Les nanoparticules ont été purifiées par dialyse ultérieure contre de l'eau (4 L, échange d'eau après 30, 90 et 180 min) pendant 4 h à l'aide d'une membrane en hydrate de cellulose (tube de dialyse Nadir, MWCO 10 kDa, Carl Roth, Karlsruhe, Allemagne). Enfin, les nanoparticules ont été filtrées dans des flacons stériles à l'aide de membranes filtrantes stériles en acétate de cellulose de 0,22 µm (Carl Roth, Karlsruhe, Allemagne).
Microscopie électronique à transmission (MET)
La morphologie et le diamètre moyen des particules ont été déterminés à l'aide d'un microscope JEM-2100F (JEOL, Freising, Allemagne). La distribution granulométrique a été déterminée sur un échantillon aléatoire de 50 nanoparticules à l'aide du logiciel X-ImageJ (Version :1.45 s, winPenPack X-ImageJ Launcher du National Institute of Health (http://rsb.info.nih.gov/ij /).
Diamètre hydrodynamique
Le diamètre hydrodynamique des nanoparticules a été enregistré à l'aide d'un Zetasizer Nano ZSP (Malvern Instruments, Herrenberg, Allemagne). Avant les mesures, les dispersions de particules ont été diluées à 1:10 dans l'eau. Les mesures ont été effectuées à 25°C. Chaque échantillon a été mesuré 3 x 15 fois. Le diamètre a été déterminé par le calcul de la distribution de volume. Ceci a été converti à partir de la distribution de la taille de l'intensité en utilisant la théorie de Mie.
ζ-potentiel
Le potentiel ζ a été mesuré à l'aide du même instrument dans les conditions décrites ci-dessus, sauf que les échantillons ont été dilués dans 0,01 M de KCl (9 :1).
Ultracentrifugation analytique (AUC)
Pour mesurer la vitesse de sédimentation, un Beckman-Coulter XL-80 K modifié avec un rotor AnTi60. Pour les expériences, la température a été réglée sur 20 °C, la vitesse a été réglée sur 10 000 rpm et 21 scans ont été effectués. Les longueurs d'onde ont été réglées sur 261 nm pour la silice et 488 nm pour la détection GFP.
Spectroscopie de fluorescence
Les spectres de fluorescence des nanoparticules, de la GFP pure et des filtrats issus d'expériences de lixiviation ont été enregistrés à l'aide du spectrofluoromètre Fluoromax-3 (Spex, Horiba Scientific, Oberursel, Allemagne). Pour les mesures, la GFP pure, les dispersions de particules et le filtrat ont été dilués à 1:10 dans de l'eau. La longueur d'onde d'excitation a été réglée sur 488 nm et le spectre a été enregistré dans une plage spectrale de 498 à 800 nm.
Rendements quantiques de fluorescence
Les rendements quantiques des nanoparticules obtenues et de la GFP pure ont été déterminés en utilisant la méthode relative de Williamson et al. [24]. Comme référence pour la GFP, la rhodamine 6G et l'Atto488 ont été utilisées. Des mesures comparatives ont été réalisées en utilisant des nanoparticules non dopées qui ont été mélangées avec le colorant de référence. Les spectres de fluorescence ont été enregistrés en utilisant une longueur d'onde d'excitation de 450 nm. Des mesures UV/vis supplémentaires ont été prises à l'aide d'un Varian Cary 300 Scan UV (Agilent Technologies, Darmstadt, Allemagne).
Pour le calcul du rendement quantique, l'Eq. 2 a été utilisé.
$$ {\varPhi}_P={\varPhi}_S\bullet \frac{{\mathrm{slope}}_S}{{\mathrm{slope}}_P}\bullet {\left(\frac{n_P}{n_S }\droit)}^2 $$ (2)Ici, φ P est le rendement quantique du produit, φ S le rendement quantique de la référence. Les termes penteS et penteP représentent les pentes dérivées des tracés de l'intensité de fluorescence intégrée en fonction de l'absorbance de la référence et du produit, respectivement. n P et n S correspondent à l'indice de réfraction du solvant utilisé [25].
Fuite de protéines
Pour les expériences de lixiviation, les dispersions de particules non diluées ont été ultrafiltrées à travers des membranes de polyéther sulfone modifiées (MWCO = 100 kDa ou 300 kDa, Pall, Dreieich, Allemagne) par centrifugation (16 000 g, 5 min).
Stabilité thermique
Pour l'analyse de la stabilité thermique, les nanoparticules et la GFP pure ont été conservées pendant 0 et 24 h à 20 ou 60 °C. Les nanoparticules et la GFP pure ont été diluées comme décrit ci-dessus.
Photoblanchiment
Pour étudier la stabilité des nanoparticules dopées à la GFP et de la GFP pure contre le photoblanchiment, les solutions ont été exposées à la lumière émise par sept LED vertes sur une période allant jusqu'à 20 min. L'intensité de fluorescence des échantillons prélevés à t = 0, 2 et 20 min ont été mesurés.
Stabilité contre la dégradation des protéines
Pour déterminer la stabilité de la GFP contre la protéinase K, la GFP pure, des nanoparticules de silice non marquées (CU S1U S2U ) avec du GFP supplémentaire et des nanoparticules de silice marquées trois fois (CF S1F S2F ) ont été utilisés dans les mêmes concentrations de GFP et la même quantité de particules. Tous les échantillons ont été dilués au 1:100. Pour la quantité de 10 molécules de GFP, une molécule de protéinase K a été choisie. Avant l'ajout de l'enzyme, un échantillon a été mesuré avec les conditions ci-dessus. Après ajout, les mesures ont été effectuées après t = 0, 15, 30, 45, 60 et 90 min.
Expériences d'absorption cellulaire
Pour déterminer l'internalisation des nanoparticules et de la GFP par les cellules, des expériences d'absorption cellulaire ont été réalisées en utilisant la lignée cellulaire de carcinome pulmonaire A549 (ACC-107).
Culture de cellules
Des cellules A549 (DSMZ, Braunschweig, Allemagne) ont été cultivées dans des flacons T75 (Greiner bio-one, Frickenhausen, Allemagne) en utilisant le milieu Eagle modifié de Dulbecco (DMEM, Thermo-Fisher-Scientific, Waltham, MA, USA) contenant 10 % de fœtus sérum de veau (SVF). 2 × 10 4 cm −2 Les cellules A549 ont été ensemencées sur des lamelles dans des plaques à 12 puits et ont été cultivées pendant 24 h. Les cellules ont ensuite été traitées avec des nanoparticules dopées à la GFP et une solution de GFP dans 1 mL de milieu pendant 24 h. Le SiO2 la concentration des nanoparticules était de 37 μg mL −1 tandis que la concentration de GFP était de 5 μg mL −1 pour les nanoparticules et la GFP pure. Après le traitement, les cellules ont été lavées deux fois avec une solution saline tamponnée au phosphate (PBS).
Préparation des échantillons et imagerie confocale
Les cellules ont été fixées avec du paraformaldéhyde à 4 % dans du PBS pendant 20 min à température ambiante. Pour la coloration de la membrane cellulaire, WGA conjugué à la tétraméthylrhodamine (agglutinine de germe de blé (2 μg mL −1 (en PBS), W849, Thermo-Fisher-Scientific (Invitrogen), Waltham, MA, USA) a été ajouté et incubé pendant 10 min à température ambiante. Après trois étapes de lavage avec du PBS, les cellules ont été lavées trois fois avec du PBS et montées sur des lames de verre avec Mowiol/DABCO (Carl Roth, Karlsruhe, Allemagne).
Les images confocales ont été prises sur un système TCS SP5 (Leica, Wetzlar, Allemagne). Pour l'imagerie, un objectif 63× à immersion dans l'huile (n = 1,518) a été utilisé. Des scans séquentiels ont été effectués à l'aide de la ligne laser argon-ion à λ = 488 nm (25 %) pour l'excitation de la GFP et un laser à semi-conducteur pompé par diode à λ = 561 nm (25 %) pour l'excitation de la tétraméthylrhodamine.
Résultats et discussion
Cette étude vise à fonctionnaliser des nanoparticules de silice avec de la GFP dans des conditions appropriées qui maintiennent les caractéristiques biochimiques et la fonctionnalité de la protéine. Dans des travaux antérieurs, nous avons synthétisé des nanoparticules de silice fluorescente monodisperse dopées par colorant proche IR dans la plage de taille comprise entre 15 et 80 nm, en utilisant une hydrolyse contrôlée par la ʟ-arginine du tétraéthoxysilane (TEOS) dans un système biphasique cyclohexane/eau [26]. Ici, nous avons adopté cette procédure de synthèse pour intégrer la GFP, en tant que protéine modèle, dans la matrice de silice. Dans le schéma 1, la procédure pour la synthèse des particules est représentée schématiquement. Les structures dopées et non dopées GFP (cœur/coquilles) sont respectivement surlignées en vert et en gris. Dans un premier temps, des particules de cœur de silice dopées GFP (CF ) ont été obtenus. Étapes de repousse ultérieures (CF S1 et CF S1 S2 ) a permis la synthèse de plus grandes tailles de particules. Lors de la première étape de repousse, la coque a été modifiée soit avec (CF S1F ) ou sans (CF S1U ) incorporation de protéines. De même, dans la deuxième étape de repousse, soit un étiqueté (CF S1F S2F , CF S1U S2F ) ou un non étiqueté (CF S1F S2U , CF S1U S2U ) shell a été ajouté. Ces variations permettent un excellent contrôle sur la quantité de protéine incorporée et son agencement sur mesure dans les coques désignées ou le noyau de la particule. De plus, des nanoparticules de silice pure sans GFP embarqué (CU , CU S1U , et CU S1U S2U ) ont été synthétisés pour étudier une influence potentielle de l'enrobage des protéines sur les propriétés des particules. De plus, pour toutes ces étapes, la GFP a été dissoute dans deux systèmes tampons différents (ʟ-arginine et NaHCO3 ) de différentes valeurs de pH, pour déterminer l'influence du solvant protéique sur les synthèses de particules, la morphologie, l'intensité de fluorescence, la longueur d'onde d'émission et le potentiel ζ.
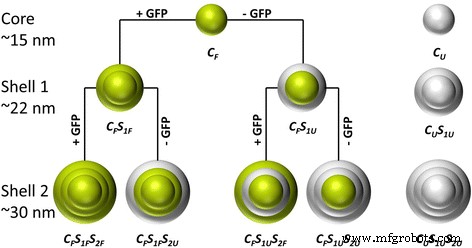
Aperçu des particules synthétisées et de leur structure particulaire. La couleur verte indique l'intégration de la GFP dans le noyau ou les coques, respectivement. La couleur grise représente les coques sans aucun GFP (CF = noyau fluorescent, CU = noyau sans étiquette, SF = coquille fluo, SU = coque sans étiquette, S1 = première couche de coque, S2 = deuxième couche de coque)
Caractérisation des nanoparticules
Détermination des attributs physiques des particules
Pour décrire la taille des particules et la morphologie après incorporation de GFP et afin de déterminer l'influence des deux systèmes tampons différents sur ces propriétés, des images MET ont été enregistrées (Fig. 1). D'autres images TEM de GFP(NaHCO3 ) modifiées, GFP(ʟ-arginine) modifiées et des nanoparticules non étiquetées sont présentées dans le SI (Fichier supplémentaire 1 :Figure S1, Fichier supplémentaire 2 :Figure S2, Fichier supplémentaire 3 :Figure S3, Fichier supplémentaire 4 :Figure S4). Suite à la procédure de synthèse avec deux étapes de repousse, trois tailles de particules différentes ont été obtenues. Les particules de noyau avaient une taille d'environ 15 nm, les particules après la première étape de repousse d'environ 22 nm et les particules après la deuxième étape d'environ 32 nm. En résumé, toutes les nanoparticules étaient approximativement sphériques et présentaient une distribution de taille étroite (p < 10 %). Les trois générations de nanoparticules de GFP(ʟ-arginine) entièrement colorées (CF , CF S1F , et CF S1F S2F ) et le GFP(NaHCO3 ) (CF ) des nanoparticules centrales ont été choisies comme modèle.

Images MET de trois générations de nanoparticules modifiées par GFP-ʟ-arginine et de particules centrales de GFP(NaHCO3 ) nanoparticules modifiées. Dans a , c et d , les trois générations de GFP(ʟ-arginine) sont indiquées :CF particules de noyau (a , dTEM = 15,5 ± 1,1 nm); CF S1F nanoparticules après la première étape de repousse (noyau + coque 1) (c , dTEM = 23,5 ± 2,0 nm) et CF S1F S2F après la deuxième étape de repousse (noyau + coque 1 + coque 2) (d , dTEM = 35,3 ± 2,0 nm). En b , le GFP(NaHCO3 nanoparticules de noyau marquées par ) (dTEM = 15,2 ± 1,2 nm) sont affichés
En comparant les tailles des différentes nanoparticules dopées et non marquées à la GFP (tableau 1), il est à noter que le même nombre d'étapes de repousse a entraîné la même taille de particule moyenne, indépendamment de la présence de protéine ou de la solution tampon utilisée. Les particules non marquées avaient également des tailles similaires (CU :dTEM = 13,4 ± 0,4 nm, dDLS = 10 ± 3 nm ; CU S1U :dTEM = 20,9 ± 1,3 nm, dDLS = 20 ± 6 nm ; CU S1U S2U :dTEM = 33,2 ± 1,0 nm, dDLS = 38 ± 10 nm).
En conclusion, il a été démontré que l'incorporation de protéines dans la matrice de silice et la solution tampon, dans laquelle la protéine était fournie, n'avait pas d'influence significative sur la taille et la morphologie des particules résultantes.
À notre connaissance, il n'y a pas d'autres nanoparticules de silice intégrées à la GFP décrites dans la littérature, présentant des petites tailles similaires ainsi que des distributions de tailles également étroites (< 10%) [20, 27]. De telles petites nanoparticules présentent un potentiel d'application prometteur dans le domaine de l'administration de protéines intracellulaires ainsi que dans le diagnostic et le traitement du cancer [28].
ζ-Potentiel
Le potentiel de toutes les nanoparticules a été déterminé par des calculs utilisant leur mobilité électrophorétique. Tous les types de nanoparticules dopées présentaient un potentiel ζ négatif avec des valeurs absolues allant de - 28 à - 36 mV (Fig. 2). En comparaison, le potentiel des particules non marquées indique des valeurs très similaires avec − 35,5 ± 2,0 mV pour les particules centrales, − 34,0 ± 3,7 mV après la première étape de repousse et − 34,5 ± 1,2 mV après la deuxième étape de repousse. Ces valeurs de potentiel ζ très négatives (< − 28 mV) indiquent une stabilité élevée des nanoparticules contre l'agglomération due à la répulsion électrostatique. Par rapport au potentiel des nanoparticules non marquées, les données indiquent que ni la taille des particules résultantes ni l'incorporation de GFP dans la matrice de particules du noyau ou de l'enveloppe des particules n'ont eu d'influence significative sur la charge des particules.
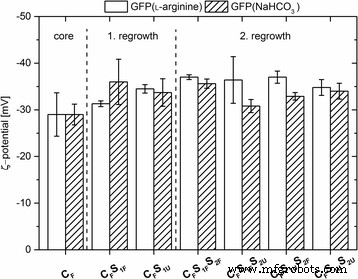
Potentiel [mV] des nanoparticules marquées. Les nanoparticules ont été préparées à partir de GFP dissous dans 7,2 mM de ʟ-arginine ou 10 mM de NaHCO3 . Barres d'erreur indiquer l'écart type dérivé de trois mesures
Études de spectroscopie
Spectroscopie de fluorescence
Toutes les nanoparticules de silice dopées GFP présentaient un maximum d'émission similaire (λ les = 508 nm), ce qui était également comparable au maximum d'émission de GFP libre (SI, Fichier supplémentaire 5 :Figure S5). Pour comparer l'intensité de fluorescence des différentes nanoparticules marquées, la concentration en nanoparticules a été normalisée (calculs en SI 5.). Comme prévu, l'ajout progressif de coques marquées a provoqué une augmentation de la fluorescence des nanoparticules (Fig. 3).
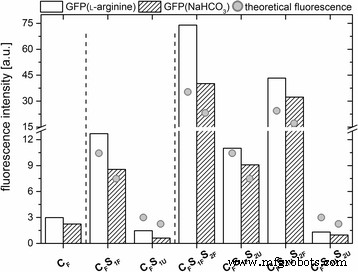
Intensité de fluorescence normalisée du maximum d'émission à 508 nm, pour chacun des différents systèmes de particules. De plus, l'intensité de fluorescence théorique (points gris ) des particules par rapport à l'augmentation du volume des particules est affiché
Les nanoparticules avec un noyau marqué uniquement, mais avec des coques non dopées, présentaient la fluorescence la plus faible. Les nanoparticules avec une coque marquée supplémentaire ont présenté une fluorescence intermédiaire, et les nanoparticules avec deux coques marquées ont montré la fluorescence la plus forte (Fig. 3). Remarquablement, l'ajout d'une coque externe non dopée semble réduire légèrement la fluorescence des nanoparticules par rapport aux nanoparticules possédant une couche externe dopée. Cet effet pourrait être provoqué par les effets de protection de la coque de silice non marquée. En résumé, l'ajout de coques dopées GFP aux particules centrales a provoqué une augmentation de l'intensité de fluorescence des nanoparticules résultantes qui semblait être corrélée au changement de volume accompagnant la croissance des nanoparticules.
L'inclusion de la GFP initialement dissoute dans la ʟ-arginine après purification a entraîné une intensité de fluorescence 1,3 fois plus élevée des nanoparticules résultantes par rapport aux nanoparticules obtenues via le processus d'inclusion analogique à partir de la GFP dissoute dans NaHCO3 . De même, la GFP diluée dans la ʟ-arginine présentait une intensité de fluorescence plus élevée par rapport à la GFP diluée dans NaHCO3 (Fichier supplémentaire 5 :Figure S5). L'effet pourrait s'expliquer par les différentes valeurs de pH des tampons (pHʟ-arginine = 10,3, pH\( _{{\mathrm{NaHCO}}_3} \) = 9.2).
Pour cette raison, la fluorescence de la GFP pure a été systématiquement mesurée en fonction de la valeur du pH (SI, Fiche complémentaire 6 :Figure S6). Les données ont montré une augmentation de forme hyperbolique de la fluorescence avec l'augmentation du pH dans la plage de pH 5,5 - 10,5. Les résultats sont cohérents avec d'autres rapports sur la fluorescence dépendante du pH de la GFP. Pour la GFP de type sauvage, il a été rapporté que la fluorescence n'est pas altérée dans la plage de pH 6 - 10 mais diminue à un pH inférieur et augmente à des valeurs de pH> 10 [29]. De plus, la sensibilité au pH de la GFP pourrait être modifiée par l'introduction de mutations ponctuelles [30]. La GFP utilisée dans cette étude possède des mutations en trois points par rapport à Aequorea protéine de type sauvage, à savoir S2A, F64L, S65T. Parmi ceux-ci, il a été démontré que la substitution de la sérine en position 65 contre la thréonine augmente l'intensité de fluorescence de la protéine, lorsqu'elle est excitée à 480 nm, car cet acide aminé est impliqué dans la formation du chromophore. De plus, le variant S65T/F64L présente une fluorescence pH-dépendante [30]. Les nanoparticules dopées GFP (CF ) présentait une fluorescence pH-dépendante comparable (Fig. 3), indiquant que le mécanisme de dépendance au pH n'était pas affecté par le processus d'inclusion.
Rendement quantique de fluorescence
Afin de caractériser davantage les propriétés des nanoparticules fluorescentes, leurs rendements quantiques ont été déterminés. Ceci a été réalisé en traçant l'intensité de fluorescence intégrée par rapport à l'absorbance à 488 nm (Fig. 4). Par la suite, les rendements quantiques ont été calculés en utilisant l'équation. 2. En utilisant la rhodamine 6G comme référence, les rendements quantiques des nanoparticules dopées GFP CF S1F et la GFP pure ont été déterminées comme étant φ\( _{{\mathrm{C}}_{\mathrm{F}}{\mathrm{S}}_{1\mathrm{F}}} \) = 0.62 et φpur GFP = 0,38, respectivement. Les résultats ont été confirmés en utilisant Atto488 comme deuxième référence (SI, fichier supplémentaire 7 :figure S7). Le rendement quantique plus élevé des nanoparticules dopées à la GFP par rapport à la GFP pure semble être causé par l'encapsulation de la GFP dans la matrice de silice et pourrait être lié soit à l'immobilisation spatiale de la protéine, soit à l'environnement chimique altéré fourni par la matrice de silice. .
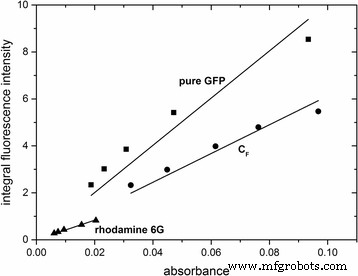
Graphiques de l'intensité de fluorescence intégrée des particules dopées à la GFP et de la GFP pure en fonction de l'absorbance à 488 nm. La rhodamine 6G a été utilisée comme référence. La corrélation linéaire a été ajustée par les lignes droites . Les équations linéaires correspondantes sont les suivantes :y pur GFP = 1.00554 × 10 10 × x , R 2 = 0.97712 ; y\( _{C_F{S}_{1F}} \) = 6.12332 × 10 9 × x , R 2 = 0.99331; y rhodamin6G = 4.1772 × 10 9 × x , R 2 = 0.99678
Stabilité des particules
Fuite de protéines
Des expériences de lixiviation ont été réalisées pour prouver la stabilité de liaison des nanoparticules dopées GFP. Après ultrafiltration à travers des membranes avec un MWCO qui permet le passage de la GFP (MW~27 kDa) mais retient les nanoparticules, aucune fluorescence n'a pu être détectée dans le filtrat, indiquant un couplage permanent de la GFP à la matrice de silice.
Ultracentrifugation analytique
Pour étayer les résultats obtenus et déterminer le type de liaison GFP à la matrice de particules, une ultracentrifugation analytique a été réalisée. A cet effet, étiqueté CF S1F S2F particules et CU non étiquetés S1U S2U particles mixed with GFP were measured at the same particle and GFP concentrations. The results (Additional file 8:Figure S8 in the SI) indicate that most of the GFP molecules are embedded into the silica matrix during the synthesis.
Thermal Stability
To determine their thermal stability, the fluorescence signals of CF in comparison to pure GFP were measured after incubation at room temperature and 60 °C respectively (Fig. 5). After 24 h at room temperature, no decrease in the fluorescence of both samples was detectable, indicating no influence on the protein stability. However, after 24 h at elevated temperature of 60 °C, only 20% of the initial fluorescence intensity of CF could be observed, whereas no fluorescence signal of pure GFP reverted. This strongly indicates a higher thermal stability of GFP-embedded silica compared to pure GFP. Since an elevated temperature leads to a significant increase in the thermal motions of the protein molecule, which can disrupt its structure, it is hypothesised that the surrounding silica matrix protected the GFP against external influences by spatial constraints.
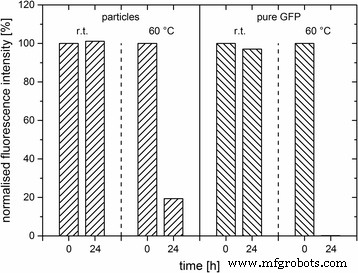
Influence of temperature (r.t., 60 °C) on the fluorescence of GFP-doped particles (CF , ʟ-arginine) and pure GFP. The normalised fluorescence intensity [%] of the emission maximum at 508 nm versus time [h] is shown
Photostability
Furthermore, the photostability of the samples was tested. For measurements, the nanoparticle stock suspension (CF , ʟ-arginine) was diluted tenfold. Pure GFP was diluted in ʟ-arginine according to the calculated concentration of GFP in the nanoparticle suspension. After exposure of the samples to light of a green LED array over a period of time up to 20 min, the fluorescence intensity was measured (Fig. 6). Within 20 min, the fluorescence intensity of the nanoparticle suspension decreased only slightly. After 20 min, 89% of the initial fluorescence (100%) of the nanoparticles was preserved. In comparison, the pure GFP seemed to be more affected by light exposure. After 20 min, only 81% of the initial fluorescence of pure GFP remained. This result indicated, that GFP, when embedded into silica nanoparticles, was better protected from photochemical alterations induced by the LED light than the pure protein.
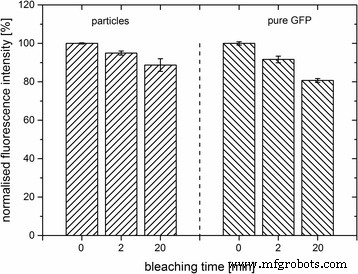
Photostability of GFP-doped nanoparticles (CF ) and pure GFP in ʟ-arginine. The normalised fluorescence intensity [%] of the emission maximum at 508 nm was measured after exposure to LED light for the given times. Data are mean values. Error bars indicate the standard deviation
Stability Against Protein Degradation
As a further characterisation step, the degradation of GFP in the presence of proteinase K was tested. Therefore, three different systems were used (pure GFP, unlabelled CU S1U S2U mixed with GFP and labelled CF S1F S2F ). For all systems, equal amounts of GFP and particles were used. After 90 min of incubation, the fluorescence intensity of pure GFP and unlabelled particles with added GFP decreased to 5 - 7% of the initial fluorescence intensity, whereas the one of the labelled particles decreased to 52% (Fig. 7). This result indicates that the GFP is protected by the silica matrix and is degraded slower than free GFP in presence of proteolytic enzymes.
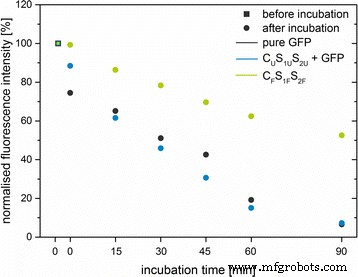
Stability against protein degradation of pure GFP (grey ), unlabelled particles mixed with GFP (CU S1U S2U , blue ), and GFP-doped silica nanoparticles (CF S1F S2F , green ). The normalised fluorescence intensity [%] of the emission maximum at 508 nm was plotted against the incubation time [min] with proteinase K
To conclude, the encapsulation of GFP into silica matrix appeared to bring about significant advantages:The stability of the protein was increased not only against elevated temperatures and light-induced photobleaching but also against the degradation through enzymes. Therefore, the silica matrix seems to protect the embedded GFP as compared to the free GFP.
Cellular Uptake Experiments
In order to determine, if the GFP-doped nanoparticles are able to deliver their cargo into cells, uptake experiments were performed (Fig. 8). A549 cells were exposed to GFP-doped nanoparticles and for comparison to the pure protein. In order to optimise the GFP load of the particles for imaging, a higher amount of GFP as compared to the nanoparticles described before was embedded into the particles. More specifically, a 20-fold amount of GFP in ʟ-arginine was used to label the second shell of the CF S1F S2F particules. These nanoparticles were diluted to a final concentration of 37 μg SiO2 per millilitre in cell culture medium and incubated for 24 h with the cells. The amount of GFP in both samples (nanoparticles and pure GFP) was 5 μg mL −1 .
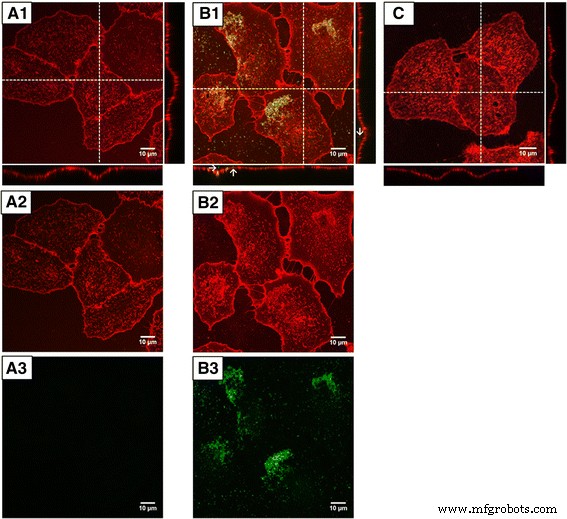
Confocal microscopy images of A549 cells after 24 h exposure to GFP dissolved in ʟ-arginine (A1 –A3 ) and GFP-doped nanoparticles CF S1F S2F (B1 –B3 ), and control cells (C ). Top (1):merge-images; middle (2):Cell membrane (WGA):red; bottom (3):GFP, green . Arrows indicate internalised nanoparticles. Contrast and brightness were enhanced by using the ImageJ software
In order to visualise the cells, the cell membrane was labelled, using tetramethylrhodamine-coupled WGA (wheat germ agglutinin). Confocal imaging was used to localise the GFP-doped nanoparticles and the pure GFP in the cells. After exposure of cells to GFP, no signal related to GFP was observed inside the cell bodies (Fig. 8a). Compared to the control cells, no difference in signal intensity of both channels could be observed (Fig. 8c).
In contrast, after exposure of the cells to the GFP-loaded nanoparticles, bright fluorescence signals were detected in the perinuclear region, indicating internalisation of the loaded nanoparticles through endocytosis. The GFP-loaded nanoparticles appeared to be excluded from the nuclear compartment. A second fraction of agglomerated nanoparticles was detected on top of the cell membrane (Fig. 8b).
In conclusion, the GFP-doped nanoparticles are internalised by the cells and are able to transport their cargo into the cells. After exposure of the cells to GFP, fluorescence signals were not detected inside the cell body. This finding is in line with the results of Pesce et al. [31], who did not observe cell-associated fluorescence after incubation of A549 cells with GFP for 24 h. The lack of cell-associated GFP signals might be due to the fact that GFP is not internalised by the cells. Alternatively, GFP fluorescence might be quenched by the low pH value present in endocytic vesicles or lysosomes or degraded by proteolytic enzymes. Therefore, the fluorescence signals of the nanoparticles might indicate a protective effect of the silica nanoparticle matrix against lysosomal degradation.
Conclusions
In this study, a novel approach is presented for synthesis of monodisperse GFP-doped silica nanoparticles with a mean particle-core size of 15 nm. By subsequent growth steps, the particle size and the amount of embedded GFP can be varied. At the end of this procedure, the fluorescence properties of GFP are kept. Incorporation of GFP into additional outer shells results in an increase in the nanoparticle fluorescence. Coverage of the nanoparticles by non-doped shells seems to slightly decrease their fluorescence. These properties indicate the potential to incorporate cargo molecules into specific particle shells. The GFP-doped nanoparticles exhibit a higher quantum yield as compared to the pure GFP. The incorporation into the silica matrix appeared to be durable, as no leaching of protein was detected by ultrafiltration. The silica matrix also seems to improve the thermal properties and photostability of the protein. Furthermore, it is possible to encapsulate different proteins in the different shells, in order to prepare multifunctional nano-carriers. Finally, the nanoparticles are applicable for intracellular delivery of their cargo. The incorporation of proteins into the particle matrix seems to increase delivery and reduce lysosomal degradation of the cargo. Therefore, the protein-doped silica nanoparticles constitute a promising novel tool for biomedical applications of nanoparticles, especially in the field of intracellular delivery of macromolecules.
Abréviations
- AUC:
-
Analytical ultracentrifuge
- DLS :
-
Diffusion dynamique de la lumière
- FCS:
-
Foetal calf serum
- GFP:
-
Green fluorescent protein
- MW:
-
Molecular weight
- MWCO:
-
Molecular weight cut off membrane
- PBS :
-
Phosphate buffered saline
- r.t.:
-
Room temperature
- TEM :
-
Microscopie électronique à transmission
- TEOS :
-
Tetraethoxysilane
Nanomatériaux
- Avancées et défis des nanomatériaux fluorescents pour la synthèse et les applications biomédicales
- 131I-Tracé PLGA-Lipid Nanoparticles as Drug Delivery Carriers for the Targeted Chemotherapeutic Treatment of Melanoma
- Synthèse facile de nanoparticules SiO2@C ancrées sur MWNT en tant que matériaux anodiques hautes performances pour batteries Li-ion
- Nouveaux nanoparticules Au Nanostars@PEG biocompatibles pour l'imagerie CT in vivo et les propriétés de clairance rénale
- Méthode de post-traitement pour la synthèse de nanoparticules binaires monodisperses FePt-Fe3O4
- Une approche simple pour la synthèse de points quantiques de carbone fluorescent à partir d'eaux usées de tofu
- Synthèse et performances in vitro de nanoparticules de fer-platine revêtues de polypyrrole pour la thérapie photothermique et l'imagerie photoacoustique
- Aptitude verte dans la synthèse et la stabilisation des nanoparticules de cuivre :activités catalytiques, antibactériennes, cytotoxiques et antioxydantes
- Nouveau double mitochondrial et récepteur CD44 ciblant des nanoparticules pour une libération déclenchée par des stimuli redox



