Études d'établissement et d'application d'un indicateur de pH de lysosome précis basé sur des nanoparticules autodécomposables
Résumé
Le pH acide des lysosomes est étroitement lié à l'autophagie; ainsi, bien connu des lysosomes précis, les changements de pH donneront plus d'informations sur le processus et l'état de l'autophagie. Jusqu'à présent, cependant, seuls des changements de pH dans une plage relativement large ont pu être indiqués, la détection exacte du pH des lysosomes n'est jamais arrivée. Dans notre étude, nous avons établi un indicateur de pH endo/lysosome basé sur le SiO2 autodécomposable système de nanoparticules avec des paramètres de synthèse spécifiques. Le bleu de méthylène (MB) central concentré dans les nanoparticules structurelles creuses centrales a présenté une libération sensible en fonction des valeurs de pH de pH 4,0 à 4,8, ce qui correspond exactement à la plage de pH des lysosomes. La corrélation linéaire des valeurs de densité optique (DO) et des valeurs de pH a été établie, qui a été utilisée pour la détection du pH des lysosomes dans 6 lignées cellulaires différentes. De plus, grâce à ce système, nous avons réussi à détecter avec précision les changements de pH moyen des lysosomes avant et après l'endocytose du silicium mésoporeux noir (BPSi) NP, clarifiant le mécanisme de la terminaison de l'autophagie après l'endocytose du BPSi. Ainsi, l'indicateur de pH luminal autodécomposable à base de nanoparticules peut fournir une nouvelle méthodologie et une nouvelle stratégie pour mieux connaître le pH des lysosomes, puis indiquer plus de détails sur le processus d'autophagie ou d'autres signaux importants sur les métabolismes.
Introduction
Les lysosomes servent de destination finale aux macromolécules, où ces macromolécules sont dégradées par des enzymes hydrolytiques activées par un faible pH [1]. Le pH acide des lysosomes maintenu par la H + −ATPase de type vacuolaire (v-ATPase) [2] qui pompe les protons du cytoplasme dans la lumière lysosomale devait maintenir l'activité d'environ 60 types d'enzymes hydrolytiques [3]. De plus, des rapports récents de la littérature ont révélé que le pH acide des lysosomes est étroitement lié à l'autophagie [4], de sorte qu'une connaissance précise des changements de pH des lysosomes donnera plus d'informations sur le processus et l'état de l'autophagie. Sur la base de nos études et de la revue de la littérature, l'endocytose des nanoparticules chargées positives aux amines augmentera probablement le changement de pH dans les endo/lysosomes, tels que les nanoparticules décorées de PEG aux amines primaires et secondaires ou certaines décorations hydrophile sur la surface des particules [5, 6].
L'augmentation du pH induite par l'endocytose des nanoparticules d'amine augmentera considérablement la localisation nucléaire du facteur de transcription EB (TFEB) [7], entraînant non seulement une régulation à la hausse de la transcription de la voie, mais également un dysfonctionnement lysosomal, entraînant finalement un blocage du flux autophagique [ 7,8,9]. Comme le TFEB régule l'autophagie, et par conséquent, sa surexpression conduit à une augmentation significative de la production d'autophagosome dans les cellules en culture.
Ainsi, afin de prédire le processus d'autophagie et les détails de l'autophagie, le pH précis du lysosome et sa mesure de changement sont très cruciaux. Jusqu'à présent, à partir de la valeur du pH de l'endo/lysosome indiquant les revues de la littérature [10] et des produits commerciaux pour détecter les valeurs de pH de l'endo/lysosome, seuls les changements de pH dans une plage relativement large pouvaient être indiqués, et la détection exacte du pH du lysosome n'est jamais arrivée. . Ainsi, pour connaître les détails de l'autophagie, l'établissement d'une méthode précise de détection de changement de pH luminal est une approche importante.
Basé sur nos expériences précédentes sur le SiO2 autodécomposable nanoparticules, dans cette étude, nous avons établi un indicateur de pH précis qui pourrait réaliser la détection des changements de pH luminal. SiO2 les nanoparticules présentent de bons avantages en termes de taille ajustable et de biocompatibilité [11]. En définissant des paramètres de synthèse spécifiques, le SiO2 auto-décomposable établi L'indicateur de pH pourrait libérer de manière sensible la charge utile de bleu de méthylène (MB) à un pH 4,0 à 4,8, ce qui correspond exactement à la plage de pH des lysosomes. De plus, la libération de MB a présenté une corrélation linéaire avec les changements de valeur de pH (schéma 1). Ensuite, nous avons testé la faisabilité de l'indicateur de pH sur les niveaux cellulaires en introduisant 6 lignées cellulaires différentes, avons réussi à déterminer les changements de pH moyens des lysosomes avant et après l'endocytose des NPs de silicium mésoporeux noir (BPSi), clarifiant le mécanisme de la terminaison de l'autophagie après BPSi endocytose. Ainsi, l'indicateur de pH luminal autodécomposable à base de nanoparticules peut fournir une nouvelle méthodologie et une nouvelle stratégie pour mieux connaître le pH des lysosomes, puis indiquer plus de détails sur le processus d'autophagie ou d'autres signaux importants sur le métabolisme.
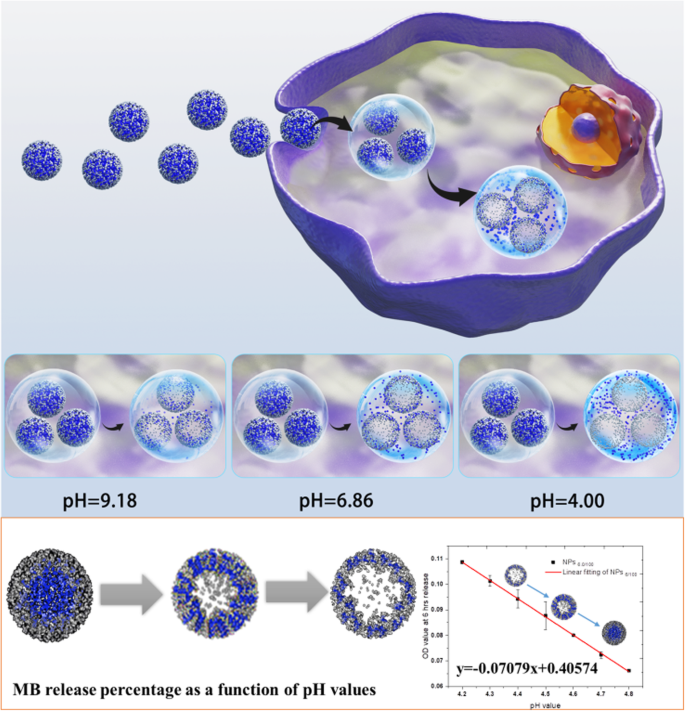
Illustration schématique de MB@SiO2 mesure du pH du lysosome dans les cellules vivantes
Matériaux et méthodes
Section Matériel
Des plaquettes de siliciure de sodium (NaSi) et de Si (diamètre 20 cm, p + (100), 0,01–0,02 Ω cm) ont été fournies par SiGNa Chemistry Inc. et Ocmetic Inc., respectivement. Bromure d'ammonium (NH4 Br, 99 %), bromure de sodium (NaBr, 99 %), toluène (anhydre, 99,8 %), acide chlorhydrique (HCl, 37 %), MB et orthosilicate de tétraéthyle (TEOS) ont été achetés auprès de Sigma-Aldrich. 0,5 kDa de méthoxy-PEG-silane et 2 kDa de méthoxy-PEG-silane ont été achetés séparément auprès de Fluorochem Ltd. et Laysan Bio Inc.. Le support RPMI 1640 a été fourni par Life Technologies. Le sérum bovin fœtal (FBS) a été acheté auprès de la technologie biologique TianHang. Bicarbonate de sodium, sulfate de streptomycine, pénicilline G, HEPES, solution de lysozyme, CellLight Early-endosomes-GFP, LysoTracker™ Red DND-99, Pierce® BCA Protein Assay Kit, chimiluminescence améliorée, conjugué pHrodo™ Red Transferrine, solution d'imagerie de cellules vivantes et Le réactif Trizol a été acheté auprès de Thermo Fisher Scientific. L'éthanol et l'eau ammoniacale étaient fournis par la Sinopharm. Le kit BioRT Master HiSensi cDNA First-Strand Synthesis a été acheté auprès de Hangzhou Bioer Technology Co., Ltd. Le lysat RIPA a été acheté auprès de Heart Biological Technology Co., Ltd. P62, TFEB et l'anticorps β-actine ont été achetés auprès de Proteintech Group , Inc. L'anticorps LC 3B a été fourni par Abcam. Le 2-(4-pyridyl)-5-((4-(2-diméthylaminoéthyl-laminocarbamoyl)méthoxy)phényl)oxazole (PDMPO) a été fourni par Yeasen Biotech Co., Ltd.
Objectif, conception et cadre de l'étude
Le but de cette étude est de (1) explorer l'influence des nanoparticules de BPSi sur l'autophagie dans les cellules HepG2, (2) découvrir le mécanisme sous-jacent des changements de valeur du pH des lysosomes affectant l'autophagie, (3) établir un indicateur précis du pH des lysosomes qui pourrait mesurer le pH des lysosomes précisément, et enfin, (4) indiquent l'influence de la fluctuation du pH sur l'autophagie. Afin de réaliser l'objectif de l'étude ci-dessus, nous avons utilisé l'expérience de séquençage du transcriptome pour explorer les modifications du gène du transcriptome dans les cellules HepG2 après avoir alimenté des nanoparticules BPSi et l'avons vérifié par RT-qPCR et expériences occidentales. Des colorants fluorescents tels que le PDMPO ont été utilisés pour mesurer le changement de pH lysosomal dans les cellules HepG2 après avoir nourri BPSi. Pour mesurer précisément le pH des lysosomes, nous avons développé MB@SiO2 nanoparticules avec 10 paramètres et testé les caractérisations de ces 10 types de nanoparticules à travers des expériences telles que DLS et HR-TEM. L'efficacité de chargement MB et l'étude de la cinétique de libération de systèmes de nanoparticules auto-décomposables de 10 séries ont été testées dans différentes solutions de pH et cellules HepG2. Pour vérifier l'emplacement intracellulaire des nanoparticules après avoir pénétré dans la cellule, nous avons effectué une expérience TEM cellulaire et une microscopie confocale de cellules vivantes. Enfin, nous avons mesuré les changements de pH lysosomal dans 6 types de cellules après avoir nourri BPSi pour vérifier l'universalité de MB@SiO2 nanoparticules pour mesurer les changements de pH lysosomal.
Synthèse de nanoparticules BPSi
Les nanoparticules BPSi ont été préparées par notre méthode précédente [12] et fournies par notre collaborateur (Wujun Xu, Department of Applied Physics, University of Eastern Finland). La préparation de BPSi, NaSi, sel d'ammonium et NaBr (NaSi :NH4 Br :NaBr de 1:4:4, w/w/w) a été broyé dans une boîte à gants avec une atmosphère d'Ar. Ils ont été autorisés à réagir dans un four tubulaire sous un N2 atmosphère à 240°C pendant 5 h (Eq. 1). Après avoir été refroidies à température ambiante, les microparticules obtenues ont été purifiées par rinçage avec des solutions de HCl 0,5 µM et de HF 1,0 µM séparément. Les microparticules ont été broyées dans de l'éthanol à 1000 rpm pendant 15 min, et les nanoparticules de BPSi avec le diamètre souhaité ont été collectées en ajustant la vitesse de centrifugation.
$$ \mathrm{NaSi}+{\mathrm{NH}}_4\mathrm{Br}\to \mathrm{NaBr}+{\mathrm{NH}}_3+\mathrm{Si}/\mathrm{H}+{ \mathrm{H}}_2 $$ (1)Grâce à l'expérience de diffusion dynamique de la lumière, la distribution du diamètre et la charge de surface de la nanoparticule ont été étudiées. Toutes les NP ont été dispersées dans le milieu après stérilisation par ultrasons légers (5 s pour les rendre uniformément dispersées dans la solution, nettoyeur à ultrasons SB-5200DT, Ningbo Scientz Biotechnology Co., Ltd.) juste avant leur introduction dans les cellules.
Établissement d'un système de nanoparticules autodécomposables de la série 10
Les nanoparticules autodécomposables de la série 10 ont été synthétisées par les méthodologies que nous avons décrites précédemment [13,14,15,16], avec des paramètres modifiés. Dans une procédure typique, une certaine quantité de MB a d'abord été ajoutée à un mélange d'éthanol (75 mL) avec une solution ammoniacale-eau (25 %, 3,4 mL), après quoi une certaine quantité de TEOS a été ajoutée. La série auto-décomposable MB@SiO2 Les NP ont été obtenues après 24h d'agitation et lavées 3 fois avant d'être séchées. Les quantités de MB et de TEOS ajoutées dans les protocoles étaient telles que décrites dans le tableau 1. La signification de 1,0/100 dans les NP1,0/100 représentait l'inventaire de MB et de TEOS lorsque nous avons synthétisé les nanoparticules, avec 1,0 mg de MB et 100 μL de TEOS. Et les significations de 1.5/100 dans les NPs1.5/100 et d'autres sont cohérents avec celui de 1,0/100.
Culture cellulaire
Afin de tester l'efficacité et l'universalité de l'indicateur de pH basé sur des nanoparticules autodécomposables, nous avons essayé de le tester sur des lignées cellulaires cancéreuses spécifiques dérivées de tumeurs. Ainsi, nous avons sélectionné le cancer du foie, le cancer du poumon, le cancer du côlon et les lignées cellulaires de mélanocytome comme objets de recherche. Les lignées cellulaires de cellules cancéreuses du côlon humain HCT116, HCT8 et HCT15 ; cellules cancéreuses du foie humain HepG-2; cellules humaines de cancer du poumon A549; et les cellules de mélanome de souris B16 ont été maintenues dans du milieu RPMI 1640 (Life Technologies) complété avec 10 % de FBS inactivé par la chaleur, 2,0 g/L de bicarbonate de sodium, 0,1 g/L de sulfate de streptomycine, 0,06 g/L de pénicilline G et 5,958 g/L HEPES. Les cellules ont été maintenues dans un incubateur de culture cellulaire standard à 37°C dans une atmosphère humidifiée avec 5% de CO2 .
Caractérisations des systèmes de nanoparticules autodécomposables de la série 10
La morphologie de toutes les séries de nanoparticules a été caractérisée par HR-TEM avec mode STEM, et la cartographie Si a été étudiée par cartographie d'éléments EDS. L'analyse de la distribution de la taille des nanoparticules a été réalisée par le logiciel ImageJ en calculant les diamètres des nanoparticules dans des images STEM sélectionnées au hasard. Le potentiel zêta des nanoparticules et l'indice de polydispersité (PDI) ont été mesurés par l'étude de diffusion dynamique de la lumière (DLS), dans des tampons en série avec des valeurs de pH spécifiques. Les données ont été analysées par SPSS15.0 et les résultats statistiques ont été présentés sous forme de moyenne ± S.D.
Étude de l'efficacité de chargement MB et de la cinétique de libération des systèmes de nanoparticules autodécomposables de la série 10
Afin d'étudier l'efficacité de chargement du MB et la cinétique de libération, la courbe standard de MB en concentrations en série a d'abord été établie. L'absorption du MB a été réalisée par spectre UV-Vis avec l'absorbance à 660 nm, qui est le λ max du monomère MB. L'efficacité de chargement MB a été calculée par l'équation ci-dessous, l'efficacité de chargement MB (%) = la quantité de MB encapsulé/(quantité totale d'entrée MB).
La libération de MB des nanoparticules de la série 10 a été étudiée dans de l'eau pure et des tampons de pH avec différentes valeurs de pH (pH 4,0, pH 6,86 et pH 9,18) et une solution de lysozyme (Thermo Scientific™ # 90082). De plus, la cinétique de libération de MB après une durée spécifique dans différents tampons de pH a également été étudiée. Les valeurs de DO à 660 nm et les pourcentages de libération de MB en fonction du temps ont ensuite été étudiés.
Plus en détail, les études de libération de MB ont été réalisées selon les protocoles ci-dessous ; nanoparticules dissoutes de la série 10 dans un tampon standard de 15 mL de pH 4,0, 6,86 et 9,18 avec une solution de lysosome, respectivement ; et effectué la libération de MB dans un mélangeur Hula à 37°C. Au cours des 15 jours suivants, 1 mL de chaque échantillon a été collecté, puis centrifugé à 12 000 rpm pendant 10 min. Le surnageant et le précipité ont été mesurés pour leurs spectres d'absorption à 200-800 nm.
De plus, la libération de MB dans des tampons de pH précis avec une solution de lysozyme dans la plage de pH de 4,1 à 5,5 a également été étudiée avec les mêmes protocoles ci-dessus. Des durées spécifiques (6 h, 12 h et 24 h) ont été placées comme points de temps d'observation. Les absorptions à 660 nm ont été enregistrées dans chaque échantillon. La relation linéaire d'absorption de chaque solution de pH et la somme des carrés des résidus ont été comptées à chaque instant, respectivement.
Pour détecter les profils de libération de MB dans les cellules, les cellules HepG-2 ont été cultivées dans un 75 cm 2 flacon de culture et alimenté en NPs (300µg/mL) lorsque les cellules ont proliféré à 70 % du flacon de culture. Toutes les 30 min plus tard, les cellules ont été collectées. Les cellules ont été congelées et décongelées à plusieurs reprises pour libérer complètement le MB dans les cellules. Les lysats cellulaires ont été centrifugés à 12000 r/min pendant 10 min. Le surnageant a été obtenu et mesuré son absorbance à 660 nm pour calculer la quantité totale de MB libérée. Dans cette étude, les cellules HepG2 ont été choisies comme objets de recherche, en raison de leur prolifération cellulaire rapide qui pourrait minimiser la variance entre les 10 groupes testés.
Colocalisation cellulaire des nanoparticules de la série 10 et performance de libération dans 6 lignées cellulaires différentes
Cell TEM a été utilisé pour étudier la colocalisation de la nanoparticule dans les endo/lysosomes suivant les protocoles standard de TEM cellulaire. Les cellules ont été ensemencées à l'intensité de 1 × 10 6 cellules/flacon et incubé pendant 24 h, permettant la fixation des cellules. Dix séries de nanoparticules dans un milieu avec la même concentration (100 μg/mL) ont été incubées avec les cellules pendant 12 et 24 h, respectivement. Les cellules ont ensuite été lavées au PBS 3 fois pour éliminer les nanoparticules en excès, puis fixées dans une solution de glutaraldéhyde à 2,5% pendant plus d'un jour. Les cellules fixées ont ensuite été lavées et colorées par du tétroxyde d'osmium, 1% dans de l'eau déminéralisée pendant 1hh, suivi d'un lavage avec du PBS 3 fois et de l'eau DI 2 fois. Le protocole MET cellulaire classique [17, 18] a ensuite été réalisé et des sections d'une épaisseur de 90 nm ont été collectées pour l'observation MET. La libération de MB en fonction des valeurs de pH a été étudiée dans 6 lignées cellulaires avec les deux NP6/100 et NPs7.5/80 . En outre, les valeurs OD de la libération du MB et les pourcentages de libération de MB ont été enregistrées pour l'analyse des données.
Enquête sur l'absorption intracellulaire de MB@SiO2 Nanoparticules
La microscopie confocale à cellules vivantes a été utilisée pour évaluer l'absorption cellulaire et le devenir intracellulaire du MB@SiO2 nanoparticules. Les endosomes précoces des cellules HepG-2 ont été colorés (CellLight Early-endosomes-GFP, BacMam 2.0 ThermoFisher Scientific C10586, avec excitation/émission ~ 488/510 nm) pendant 16 h. Et puis les cellules ont été incubées avec des NPs6/100 (excitation/émission MB :640/650-700 nm) à la concentration de nanoparticules de 100 μg/mL à des intervalles de temps spécifiques (2 h, 2,5 h, 3 h, 5 h et 6 h). Avant que les images ne soient prises, le lysotracker a été coloré avec le LysoTracker™ Red DND-99 (Thermo Fisher Scientific L7528, excitation/émission :577/590 nm) pendant 40 min. Après cela, retirez la solution de coloration et lavez les cellules 2 à 3 fois dans du PBS. Les images ont été prises à l'aide d'un microscope confocal Nikon A1R.
Séquençage du transcriptome pour évaluer le changement d'expression génique après l'alimentation BPSi
L'extraction de l'ARN total du groupe témoin et du groupe traité par BPSi a été réalisée en utilisant le réactif Trizol en suivant les procédures opératoires standard. La qualité de l'échantillon d'ARN total initial pour l'expérience de séquençage a été détectée à l'aide d'un spectrophotomètre NanoDrop ND-2000. L'ARN total qui a passé le contrôle de qualité a été utilisé dans des expériences de séquençage ultérieures. Une comparaison de l'expression des gènes a été réalisée par séquençage de nouvelle génération. Tous les programmes de séquençage ont été réalisés par BGI-Shenzhen Corporation (Shenzhen, Chine) en utilisant la plate-forme BGISEQ-500. Les données brutes obtenues par séquençage sont soumises à un contrôle qualité pour déterminer si les données de séquençage sont adaptées à une analyse ultérieure. En cas de réussite, effectuez une analyse quantitative des gènes basée sur les niveaux d'expression génique et effectuez une analyse d'enrichissement significatif des fonctions d'ontologie génique (GO) sur les gènes exprimés de manière différentielle entre les échantillons sélectionnés.
Test de réaction en chaîne par polymérase quantitative par transcription inverse (RT-qPCR) pour confirmer l'activation du réseau de gènes TFEB-CLEAR
L'ARN total a été extrait des cellules HepG-2 cultivées des groupes témoins et traités par BPSi à l'aide du réactif Trizol et transcrit à l'envers en ADNc à l'aide du kit de synthèse BioRT Master HiSensi cDNA First-Strand (Hangzhou Bioer Technology Co., Ltd. .) avec des amorces aléatoires. L'ADNc a été utilisé pour amplifier le réseau de gènes TFEB-CLEAR par PCR quantitative avec le système de PCR en temps réel Applied Biosystems™ 7500 (Applied Biosystems, Life Technologies, Carlsbad, CA) avec l'actine comme contrôle de référence. Les amorces utilisées pour la RT-PCR quantitative étaient répertoriées dans le tableau S4.
Dosage Western Blot pour confirmer que l'autophagie est activée après l'alimentation BPSi
Les protéines cellulaires du groupe témoin et des groupes traités au BPSi à différentes concentrations ont été extraites par le lysat RIPA (Heart Biological Technology Co., Ltd.). L'inhibiteur de protéase a été ajouté au lysat de RIPA et pré-refroidi sur de la glace. Lavé les cellules 3 fois par PBS pré-refroidi. J'ai complètement vidé le liquide et j'ai placé le plat dans de la glace pendant 2 min. Quatre cent microlitres de lysat RIPA ont été ajoutés à la surface de la boîte entière, pipetés plusieurs fois avec une pipette et incubés sur de la glace pendant 30 min, pendant lesquels la boîte a été agitée plusieurs fois pour lyser complètement les cellules. Le fluide cellulaire lysé a été transféré dans des tubes Eppendorf de 1 ml et centrifugé à 13 000 rpm pendant 10 min, 4 °C. Le surnageant obtenu a été bouilli dans de l'eau pendant 10 min et placé à – 20 °C pour une utilisation ultérieure. La concentration en protéines a été quantifiée à l'aide du Pierce® BCA Protein Assay Kit (Thermo scientifique).
Des extraits cellulaires contenant 25 µg de protéine totale ont été directement soumis à une SDS-PAGE et transférés. Les membranes ont été bloquées avec 5% de lait écrémé et sondées avec des anticorps primaires qui reconnaissent P62 (Proteintech # 18420–1-AP), TFEB (Proteintech # 13372-1-AP), LC 3B (Abcam # ab192890), et la -actine (Proteintech # 20536-1-AP). Les anticorps secondaires ont été choisis en fonction de l'espèce d'origine des anticorps primaires et détectés par chimiluminescence renforcée (Pierce) ou en utilisant le système d'imagerie Bio-Rad ChemiDoc XRS + Gel (Bio-Rad, USA). L'intensité de bande normalisée de P62, TFEB et LC 3B par rapport à la -actine a été quantifiée par densitométrie à l'aide du logiciel ImageJ dans les groupes BPSi, et les données sont la moyenne ± S.D. à partir de trois expériences indépendantes.
Mesure du pH cellulaire par PDMPO et conjugué pHrodo™ Red Transferrine par microscopie confocale
Étude PDMPO
1 × 10 5 Les cellules HepG-2 ont été cultivées sur des plaques confocales stériles pendant une nuit et les nanoparticules de BPSi ont été nourries avec une concentration de 100 μg/mL. Le lendemain, avant la coloration par immunofluorescence, les lames ont été lavées trois fois avec 0,01 M de solution saline tamponnée au phosphate (PBS), pH 7,4, puis ajoutées 1 M de colorant PDMPO (Ex/Em = 329/440). Après avoir été lavées trois fois avec du PBS, les cellules ont été incubées avec du milieu de culture RPMI-1640 frais et observées au microscope à fluorescence (Nikon A1R, Japon) avec une caméra CCD et ont pris des photos dans les 5 min, et le rapport d'intensité de fluorescence bleu et vert dans les lysosomes a ensuite été calculé selon la procédure de Chen et al. [19].
Étude sur le conjugué de transferrine rouge pHrodo™
Les cellules HepG-2 ont été étalées dans des plaques confocales de la même manière pour la fixation des cellules pendant 24 h, puis les plaques ont été conservées sur de la glace pendant 10 min. Cellules lavées avec une solution d'imagerie cellulaire vivante froide contenant 20 mM de glucose et 1% de BSA. Ajouter le conjugué pHrodo™ Red Transferrine (Ex/Em = 560/585 nm) à 25 μg/mL dans une solution d'imagerie cellulaire vivante et incuber à 37 °C pendant 20 min, puis laver les cellules dans une solution d'imagerie cellulaire vivante. L'observation a également été réalisée par microscopie confocale. L'analyse quantitative de l'intensité des images de microscopie a été réalisée par le logiciel ImageJ.
Détection du pH lysosomal des cellules
Les cellules A549, HepG-2, HCT8, HCT15, HCT116 et B16 ont été cultivées dans un 75 cm 2 flacon de culture et alimenté avec des NP lorsque les cellules ont proliféré à 70 % du flacon de culture, et 6 h plus tard, les cellules ont été collectées. Les cellules ont été congelées et décongelées à plusieurs reprises pour libérer complètement le MB dans les cellules. Les lysats cellulaires ont été centrifugés à 12000 r/min pendant 10 min. Le surnageant a été obtenu et mesuré son absorbance à 660 nm pour calculer la quantité totale de MB libérée. L'absorbance des NPs6.0/100 à un pH standard a été comparé pour obtenir la valeur du pH de chaque cellule.
Analyse statistique
L'analyse statistique a été effectuée avec le logiciel SPSS15.0 en utilisant une analyse de variance à deux voies (ANOVA) pour les groupes indépendants et en utilisant la méthode Tukey HSD pour le test de comparaisons multiples. La signification statistique était basée sur une valeur de P < 0,05.
Résultats et discussion
Nous avons d'abord détecté l'expression différentielle des gènes lorsque les cellules ont été nourries avec les doubles nanoparticules de silice poreuse noire fonctionnelle PEG (BPSi NP) (proposées par notre laboratoire collaborateur [12]). Le changement du potentiel zêta de − 18,5 à + 2,8 mV a également indiqué que la double-PEGylation de surface était réussie (Fig. S1a). Le diamètre moyen des nanoparticules de BPSi était de 156 nm (Fig. S1b). Sur la base de la carte thermique de cluster de l'expression génique différentielle (Fig. S2), nous avons sélectionné plus de 2 fois les expressions géniques différentielles pour une enquête plus approfondie. Go et KEGG ont été introduits pour analyser les gènes différentiels. À partir des cartes à bulles d'enrichissement de Go (Fig. 1a), les gènes métaboliques et associés aux lysosomes, y compris l'assemblage des phagolysosomes, la phagocytose et le processus métabolique xénobiotique, ont été sélectionnés pour une analyse plus approfondie. Notamment, les expressions géniques associées à TFEB-CLEAR [17] étaient significativement augmentées. Les résultats de la RT-PCR (Fig. 1b) ont également vérifié les résultats du séquençage des gènes, les gènes sur TFEB-l'expression lysosomale coordonnée et la voie de régulation (CLEAR) ont considérablement augmenté, tels que CTSD, CTSF, TFEB, MFN1, LAMP2 et TPP1. . Ces gènes ont été marqués sur la voie des lysosomes, comme le montre la figure S3. Leur expression est supérieure à celle du groupe témoin et a une signification statistique (P Contrôle BPSi VS <0,05). Et le TFEB régule positivement l'expression des gènes lysosomal, contrôle la population de lysosome et favorise la dégradation cellulaire des substrats lysosomal.
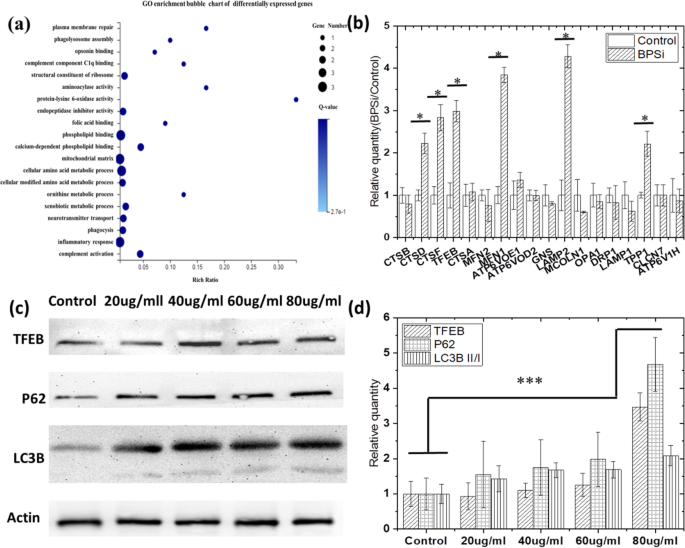
L'autophagie est activée dans les cellules HepG-2 après avoir nourri BPSi. un Carte à bulles d'enrichissement GO des gènes différentiellement exprimés trouvés par séquençage du transcriptome. b Vérification des modifications génétiques dans le réseau de gènes TFEB-CLEAR après BPSi traité par des expériences RT-qPCT. c Expression des protéines P62, TFEB, LC3B II/I après traitement par BPSi. d Intensité de gris de bande normalisée de la protéine P62, TFEB, LC3B II/I dans les groupes traités par BPSi selon le groupe témoin. Les données ont été présentées sous forme de moyenne ± S.D.
De plus, le TFEB régule l'autophagie et sa surexpression entraîne une augmentation significative de la production d'autophagosome dans les cellules en culture du fait que la fonction principale du gène TFEB est d'induire la biosynthèse du lysosome et de favoriser l'apparition de l'autophagie [20]. L'analyse Western blot a également été utilisée pour attester si l'autophagie s'est produite lorsque les cellules ont été nourries avec des NP BPSi. Le but du test de Western était de confirmer davantage que l'expression de TFEB était augmentée et l'apparition d'autophagie cellulaire après l'alimentation de BPSi. Les protéines LC3B et P62 sont toutes deux des marqueurs de l'autophagie. Lorsque l'autophagie se produit, l'expression de la protéine associée aux microtubules 1A/1B-chaîne légère 3B (LC3B) II/I augmente. p62 est un récepteur pour les vésicules qui seront dégradées par l'autophagie et est également un récepteur pour les agrégats de protéines ubiquitinées à éliminer, et son expression diminue lorsque l'autophagie se produit. Nous mesurons donc l'expression de ces protéines dans l'expérience occidentale.
D'après les résultats du Western blot illustrés à la Fig. 1c, d, TFEB (P 80 μg/ml VS contrôle = 0,000008), LC3B II/I (P 80 μg/ml VS contrôle = 0,000297), et p62 (P 80 μg/ml VS contrôle = 0,000016) les protéines sont toutes régulées positivement de manière significative. Comme la régulation à la hausse des protéines TFEB et LC3B II/I indiquant l'activation de l'autophagie [18], nous avons suspecté que l'endocytose BPSi favorise la survenue de l'autophagie. Cependant, la protéine p62 est supposée être régulée à la baisse au cours du processus d'autophagie, en raison de la nature de la protéine porteuse qui a amené les endosomes aux lysosomes et s'est finalement dégradée. Dans notre étude, la régulation à la hausse significative de p62 a indiqué la fin de la dégradation au cours du processus de fusion endo-lysosome [21], qui est probablement causée par l'augmentation du pH dans les vésicules endo-lysosome. Ainsi, l'endocytose BPSi peut d'abord induire l'apparition de l'autophagie, puis inhiber le processus d'autophagie en augmentant les valeurs de pH endo/lysosome, du fait de son alcalinité amide.
Afin de témoigner des caractéristiques d'augmentation du pH dans les endo/lysosomes par endocytose BPSi, deux sondes fluorescentes de pH commerciales ont été utilisées dans notre étude, le pHrodo™ Red Transferrin Conjugué (Thermo Fisher # P35376) et RatioWorks™ PDMPO.
Le pHrodo™ Red, en tant qu'indicateur de pH intracellulaire commercial, présente généralement une fluorescence faible à pH neutre, mais une fluorescence croissante lorsque le pH baisse. Il était censé quantifier le pH cytosolique cellulaire dans la plage de 9 à 4 avec un pKa de ~ 6,5 avec une excitation/émission de 560/585 nm. Nous avons pu obtenir une conclusion d'analyse qualitative à partir de 6 déterminations de lignées cellulaires selon lesquelles l'endocytose des NP BPSi a la capacité d'augmenter les valeurs de pH dans les endo/lysosomes, en raison de l'affaiblissement des signaux fluorescents rouges (Fig. S4 et S5). Cependant, après avoir répété les expériences plusieurs fois en suivant les protocoles de fonctionnement du produit, nous avons à peine analysé quantitativement la valeur de pH exacte qui a diminué parmi les différentes lignées cellulaires avant ou après l'alimentation avec des NP BPSi en raison de l'absence de corrélation entre l'intensité et les valeurs de pH établies.
Le PDMPO a ensuite été utilisé comme une meilleure solution pour indiquer les changements de valeur du pH après l'endocytose BPSi, qui introduisent des techniques d'imagerie de rapport dans la mesure quantitative du pH. Le PDMPO [2-(4-pyridyl)-5-((4-(2-diméthylaminoéthyl-laminocarbamoyl) méthoxy) phényl) oxazole] est caractérisé comme une sonde de pH acidotrope à double excitation et à double émission. Il émet une fluorescence verte intense à un pH inférieur et donne une fluorescence bleue intense à un pH plus élevé. Cette fluorescence unique dépendante du pH fait du PDMPO une sonde de pH idéale pour les organites acides avec un pKa =4,47. Le PDMPO marque sélectivement les organites acides (tels que les lysosomes) des cellules vivantes et les deux pics d'émission distincts peuvent être utilisés pour surveiller les fluctuations de pH des cellules vivantes dans les mesures de rapport. Cependant, nous n'avons toujours pas réussi à mesurer les valeurs de pH dans 6 lignées cellulaires avant et après l'alimentation BPSi. Comme les résultats le montrent sur la figure S6, aucune différence significative n'a été observée dans les 6 lignées cellulaires avant et après l'alimentation par BPSi. Bien qu'une corrélation ait été établie entre le rapport bleu/vert et les valeurs de pH (Fig. S7), la corrélation non linéaire de pH 4–5 fait que la méthode PDMPO a échoué dans l'analyse quantitative des endo/lysosomes avant et après les tétées BPSi.
À partir des données de deux indicateurs de pH commerciaux ci-dessus, nous avons d'abord démontré notre soupçon que des nanoparticules décorées de PEG avec de l'amide sur la chaîne PEG pourraient faire augmenter le pH de l'endo/lysosome en raison de la nature alcaline de l'amide. Cependant, sans l'analyse quantitative des changements de pH précis (plage de pH 0,1), nous ne pouvons toujours pas établir les corrélations entre l'état de l'autophagie et les valeurs de pH de l'endo/lysosome, ce qui a échoué dans la prédiction de l'autophagie.
Sur la base de notre étude précédente sur les nanoparticules autodécomposables [13, 14] [15, 22], nous avons conservé la même concentration d'hydroxyde d'ammonium dans 75 % d'éthanol, mais ajusté les concentrations de MB et de TEOS. Deux séries de quantité de TEOS ont été fixées à 100 μL et 80 μL, afin d'obtenir différentes épaisseurs de coque et tailles de pores. Dix séries de quantités de MB ont été définies pour obtenir différentes tailles de structure creuse centrale et d'efficacité de chargement de MB.
Les quantités de MB et de TEOS ajoutées dans les protocoles étaient telles que décrites dans le tableau 1 ci-dessous.
Comme le montrent les figures 2a, b, la taille des nanoparticules augmente avec l'augmentation de la quantité de MB, dans les deux concentrations de TEOS (100 μL et 80 μL). A la même concentration de MB, la taille des particules a augmenté avec l'augmentation de la quantité de TEOS. Moreover, with the increase of the TEOS amount, the shell thickness grew up, which has been proved by the element mapping (shown as Fig. 2c). The polydispersity index (PDI) and surface charge of the nanoparticles are shown in Fig. S8 and Table S1. The morphology studies predicted that with the increase of MB amount, the loading efficiency will grow up, leading to the faster release profile, while with the increase of TEOS amount, the release will slow down. And we need to find out the appropriate MB and TEOS concentration, with which we could obtain the optimized nanoparticle systems, that we may be able to make the MB release profile linear correlated with the pH changes.

Morphology characterization of 10 different self-decomposable nanoparticles with specific MB or TEOS amount. un STEM figures. b Nanoparticle size distribution analysis. c Si mapping of 10 self-decomposable nanoparticles. Scale bars in all figures are 100 nm. The size distribution analysis was performed by randomly chosen 100 nanoparticles from STEM figures and measured by ImageJ software. Data was presented as mean ± S.D.
The MB loading efficiency was determined by UV-Vis spectrum. The standard curve (Fig. S9) of MB was firstly drawn using series concentrations of MB solution (from 6.25 to 46.88 μg/mL), with the equation as y = 67.63x + 0.10919, R 2 = 0.9987. As calculated with the equation above, we obtain MB loading efficiency of 10 self-decomposable nanoparticles with specific parameters, detailed data shown in Fig. S10.
Before the study of the MB release profiles in different pH solutions, the release profiles in pure water have been studied. As shown in Fig. S11 and Fig. S12, all the nanoparticles with TEOS amount of 80 μL presented increased MB release along with the duration increase, which was reflected by the UV–Vis absorption. Moreover, with the MB encapsulated amount increase, the growth trend of MB release becomes more significant. Also, the release velocity grows faster. However, as the TEOS amount increase to 100 μL, the particle surface became more densed and the release becomes slower when the MB amount below 3.0 mg; almost no increase trend could be observed in the MB release in water during 14 days of release. As long as the MB amount increases to above 4.0 mg, an obvious increase trend of MB release could be observed. One thing to be noticed is that the nanoparticle parameter of both NPs7.5/80 and NPs6.0/100 presented solid growth as the time prolongs, almost showed a linear increase trend during the first 7 days, and then reached the platform.
Then, we focused on the MB release behavior in different pH buffers to figure out whether self-decomposable nanoparticles with specific parameters could have the linear pH-dependent MB release.
Firstly, we carried out the MB release experiments at pH 4.0 buffer solution. From Fig. S13, we could easily reach the conclusion that with the same TEOS amount of 100 μL, the MB release velocity presented a similar trend was observed in the 5 nanoparticle systems of TEOS at 80 μL (Fig. S14), the center positive correlation with the MB encapsulated amount.
Concentrated MB diffuses into the surrounding solution via diffusion due to concentration difference. The bigger concentration gradient makes the faster MB release. Compared with the MB release in pure water, we found that the acidic environment speeded up the release of MB (Fig. S13 and S14 compared with Fig. S11 and S12), indicating that the MB release is not only driven by diffusion; however, in acidic solutions, electrostatic repulsion is also an important driven force due to the positive charge nature of MB. We then calculated the release percentage of each nanoparticle parameter according to the MB loading efficiency, MB standard curve, and the dilution ratio at measurements. The release percentage reflected the release speed of MB in pH 4.0 acidic solution, and the results (Fig. 3) showed that only the release percentage of NPs7.5/80 presented linear release in pH 4.0 solution. Other nanoparticle systems with specific MB and TEOS parameters showed similar release trends, and the release percentage did not have a linear growth. One exception is NPs6/100 , and the MB release reached the platform in only 72 h; thus, it was hard to tell whether the MB release could grow linear before that duration at this stage.
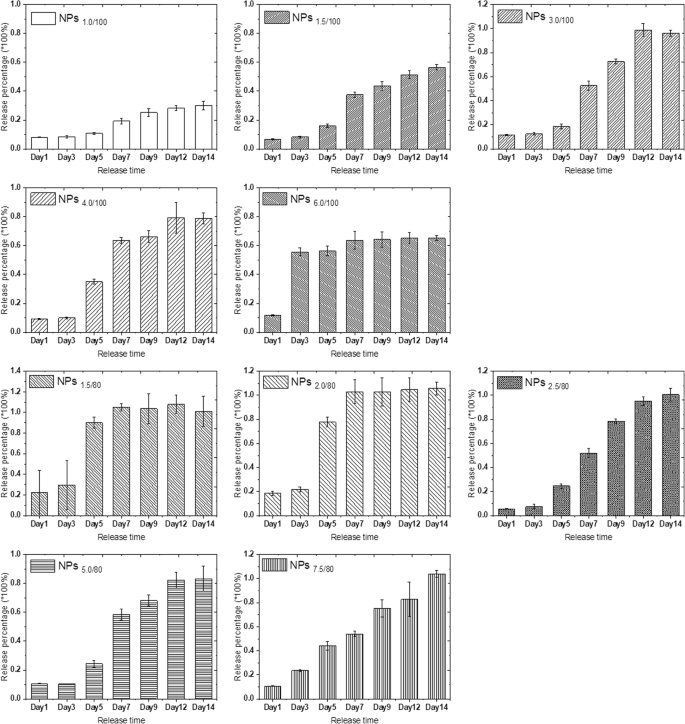
MB release percentage of 10 series self-decomposable nanoparticles after a specific duration in pH 4.0 buffer. All experiments were triple repeated, and the data were shown as mean ± S.D.
Meanwhile, we tested the MB release profiles in near-neutral and alkali buffers (pH 6.86 and pH 9.18). The results in both Fig. S15, S16, Fig. S17, and S18 demonstrated that the MB release slowed down with the solution pH increase to 6.86; moreover, with the central MB concentration increased, the MB release percentage decreased. At pH 9.18, all nanoparticles with 10 specific parameters presented a very slow MB release (Fig. S19 and S20); no matter in UV–Vis absorption or the release percentage, the trend was similar with the one in pH 6.86 buffer, but with even lower release percentage. So, it was clear that the self-decomposable nanoparticles only presented MB release linear growth in acidic solutions. We thought back to the endo/lysosomes pH, from 4 to 5, which is exactly the pH range of MB linear growth as a function of time in a specific MB/TEOS parameter. Thus, we get more confident that the self-decomposable nanoparticle system may be an accurate measuring tool for quantitative determining the endo/lysosome average pH, then provide evidence on the exact pH value of autophagy status.
The precondition of using the specific self-decomposable nanoparticles as an endo/lysosome pH indicator is that the nanoparticles stay stable in the endo/lysosome during the whole measurement process. Secondly, the MB release in endo/lysosome should occur smoothly when the measurement carried out.
The colocalization of the nanoparticle in the endo/lysosomes by cell TEM study and the MB release in 6 different cell lines were studied. From the cell TEM results, all of the 10 series nanoparticles stayed in the endo/lysosomes without escaping, after 24-h incubation with the HepG-2 cells (Fig. 4). Since the diameter of HepG2 cells used in Fig. 4 is about 10–20 μm and the diameter of MB@SiO2 nanoparticles is between 75 and 200 nm, it will be very difficult to clarify the nanoparticle morphologies using the images with low magnification (as shown in Fig. S21). We also investigated the intracellular location of the nanoparticles in the other 5 cell lines, and 4 nanoparticles were randomly selected to demonstrate the nanoparticles were trapped in the endo/lysosomes (Fig. S22). The nanoparticles with all parameters showed a central hollow structure in all other 5 cell lines after 24-h incubation, indicating the MB release. Moreover, under more precise observation, we noticed the MB release may be different due to different hollow sizes, pointing to the fact that (1) the endo/lysosome pH in different cells is different and (2) the MB release from the nanoparticles is very sensitive to the endo/lysosomes pH, especially for the NPs6/100 and NPs7.5/80 . From Fig. S23, we can find that nanoparticles have already realized the endocytosis and stayed in the vesicles 2 h after nanoparticle feeding, then nanoparticles gradually accumulated in lysosomes.
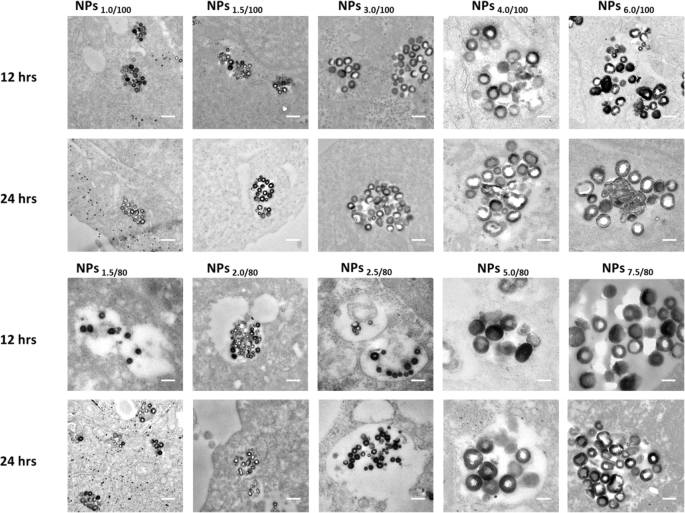
The colocalization of the nanoparticle in the endo/lysosomes by cell TEM study after 12 and 24 h incubation with the 10 series nanoparticles. The scale bar is 200 nm in the TEM images
We then evaluated the correlation between the pH values and the OD values in pH 4.0–4.8. From the results in Fig. 5 and Fig. S24, for NPs6/100 and NPs7.5/80 nanoparticle systems, the MB release presented a linear decrease as a function of pH in the pH range from 4.0 to 4.8.
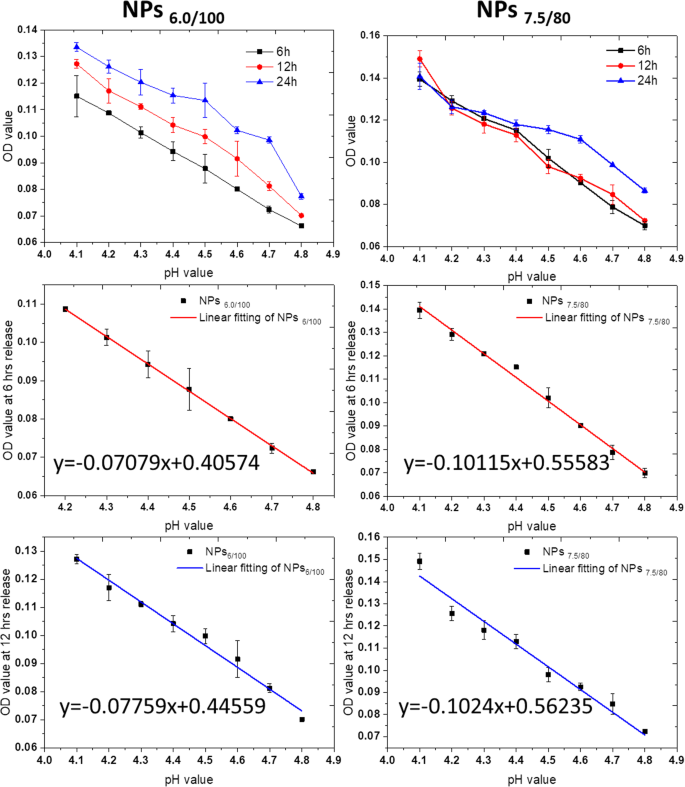
MB release as a function of pH values in NPs6/100 and NPs7.5/80 after specific incubation duration, 6 h, 12 h, and 24 h. The linear correlation equations were also calculated for 6 h and 12 h for MB release from both NPs6/100 and NPs 7.5/80 as a function of pH values. All experiments were carried out triplicated, and the data were shown as mean ± S.D.
We then converted the OD value to the MB release percentage according to the MB loading efficiency and feeding amount. As shown in Fig. S25, in the first 6 h, the MB release percentage in NPs6/100 and NPs7.5/80 nanoparticle systems also presented as a function of pH values. We then calculated the residual sum of squares and Pearson’s related coefficient at 6 h and 12 h release duration, respectively, as the residual sum of squares present a negative correlation with closeness of linear fitting, while the closer the absolute value of Pearson’s related coefficient to 1, the more linear it is. As shown in Table S2 and S3, the highest degree of linearity is the fitting of NPs6.0/100 nanoparticle systems, followed by the one of NPs7.5/80 at 6 h release.
Till then, we were so excited by the results that the method for precisely monitoring the pH values has been established, especially with the accuracy less than or equal to 0.1 pH value interval. That means, we have great possibilities to figure out the correlation between endo/lysosome pH values and the autophagy status, which is of great significance for better studying the autophagy mechanism and predicting the autophagy process. As we can see in Fig. S26, the MB release in HepG-2 cells have already reached the plateau after incubation for 4 h. Thus, we chose 6 h as the observation time point.
We then carefully investigated the MB release of NPs6.0/100 in 6 cell lines in the nanoparticle cell interaction duration of 6 h, including liver cancer HepG-2 cell line, colon cancer HCT8, HCT 15, and HCT 116 cell lines, lung cancer A549 cell line, and myomelanocytic cancer B16 cell line.
As shown in Table 2, we clearly differentiate the endo/lysosomes in 6 cancer cell lines, with the accuracy at 0.01 pH values, which is impossible to be done with the commercial intraocular pH indicator kits.
Moreover, we re-evaluated pH values in endo/lysosomes of the HepG2 cells before and after cultured with BPSi nanoparticles. We reached the conclusion that BPSi uptake significantly increases the endo/lysosome pH values, from 4.70 ± 0.09 to 5.59 ± 0.05, perfectly illustrating the reason for BPsi uptaken induced the autophagy initially then terminated the autophagy flux. The intracellular uptake of BPSi makes the quantities of the endo/lysosomes increased, which was consistent with the results of gene sequencing, that autophagy-related genes (TFEB-CLEAR) were activated. Meanwhile, the autophagy termination by the increased pH values in endo/lysosomes also coincide with the results of p62 proteins upregulation in Western blot study.
Discussion
Nanoparticles can generally cause autophagy in cells [23], and studies have shown that the autophagic response to nanoparticles presenting a neutral or anionic surface involves enhanced clearance of autophagic cargo. Cell exposure to nanoparticles presenting a cationic surface, on the other hand, results in transcriptional upregulation of the TFEB pathway, but also causes lysosomal dysfunction, ultimately resulting in blockage of autophagic flux [7]. And our results are in consistent with these previous conclusions. In our study, we found that the expression of autophagy-related genes and proteins in HepG2 cells has been increased after feeding of BPSi nanoparticles through transcriptome sequencing, RT-qPCR, and Western experiments. However, the expression level of autophagy-related P62 protein does not decrease as the autophagy is activated. We suspect that the PEG-amine on the surface of BPSi nanoparticles raises the pH value of the lysosome, resulting in inhibition of P62 degradation. Existing lysosomal pH indicators cannot verify our guess. To accurately measure the lysosomal pH of living cells, we established a new method for endo/lysosomes pH qualitative determination based on self-decomposable nanoparticle systems. Ten nanoparticle systems with specific MB/TEOS parameters were employed for obtaining optimized pH sensitively responsive measurement method. The radial MB concentration gradient from inner out served as a major driving force for MB release. The drug release proceeded with simultaneously carrier decomposition, which was driven by a diffusion-controlled mechanism. Moreover, as the pH value decreases, the hydrogen ion concentration increases, and the enhanced electrostatic interaction promotes inner MB to release faster than in neutral solution [24]. The optimized central hollow nanoparticle system could release the central concentrated MB as a linear function of precise pH values in the range of pH 4.0–4.8, which is exactly the pH of lysosomes. Finally, by this qualitative pH indicator based on self-decomposable nanoparticles, we have succeeded in the detection of the average pH values of lysosomes in 6 cell lines. Moreover, by this system, we can qualitatively differentiate the pH changes of lysosomes before and after BPSi nanoparticle endocytosis by HepG-2 cells, clarifying the mechanism of the autophagy occurrence and then termination after BPSi endocytosis. The self-decomposable nanoparticle systems pave a brand new way for studying the luminal pH values, providing new tools to know better of the cell signaling and metabolism, and then providing new ways and methods for the treatment of cancer [25, 26].
Conclusion
In this study, we found that BPSi can promote cell autophagy through transcriptome sequencing, but the amino groups on the surface of the nanoparticles can increase the pH of the lysosome and inhibit the degradation of autophagic flow. Thus, the lysosome pH significantly influences the autophagy stages. And precisely acquiring the information of lysosome pH will promote the perceiving of autophagy. However, the existing fluorescent lysosomal pH indicators could only determine a wide range of lysosomal pH; thus, we established a precise lysosomal pH indicator based on the self-dissociation system. By adjusting the synthesis parameters of MB@SiO2 , the release of MB loaded on the nanoparticles was linearly and negatively correlated with pH. And the nanoparticles mainly stay in the lysosome after entering the cell. By measuring the amount of MB released in the cells, the pH value of the lysosome can be calculated exactly according to the linear function. The established precise pH indicator provided a brand new tool and methodology to precisely study the lysosome pH values and further acquire more information on autophagy.
Disponibilité des données et des matériaux
Toutes les données générées ou analysées au cours de cette étude sont incluses dans cet article publié et ses fichiers d'informations supplémentaires.
Abréviations
- BPSi:
-
Black mesoporous silicon
- FBS :
-
Sérum fœtal bovin
- GO :
-
Gene ontology
- HCl :
-
Acide chlorhydrique
- KEGG:
-
Kyoto Encyclopedia of Genes and Genomes
- Mo :
-
Bleu de méthylène
- NaBr:
-
Sodium bromide
- NaSi :
-
Siliciure de sodium
- NH4Br:
-
Ammonium bromide
- NP :
-
Nanoparticules
- OD :
-
Densité optique
- PBS :
-
Solution saline tamponnée au phosphate
- PDMPO:
-
2-(4-Pyridyl)-5-((4-(2-dimethylaminoethy- laminocarbamoyl) methoxy) phenyl) oxazole
- RT-qPCR :
-
Réaction en chaîne par polymérase quantitative de transcription inverse
- TEOS :
-
Orthosilicate de tétraéthyle
Nanomatériaux
- Surveillance des applications SaaS :voir la forêt et les arbres
- Le développement et l'application de tissu en fibre de verre
- Préparation de nanoparticules mPEG-ICA chargées en ICA et leur application dans le traitement des dommages cellulaires H9c2 induits par le LPS
- Absorbeur parfait à bande ultra-étroite et son application en tant que capteur plasmonique dans la région visible
- La toxicité potentielle pour le foie, le cerveau et l'embryon des nanoparticules de dioxyde de titane sur des souris
- Formation et propriétés luminescentes de nanocomposites Al2O3:SiOC à base de nanoparticules d'alumine modifiées par le phényltriméthoxysilane
- La préparation de la nanostructure jaune-enveloppe Au@TiO2 et ses applications pour la dégradation et la détection du bleu de méthylène
- Aptitude verte dans la synthèse et la stabilisation des nanoparticules de cuivre :activités catalytiques, antibactériennes, cytotoxiques et antioxydantes
- Film de condensateur :les propriétés, la construction et l'application



