le microARN-18a des macrophages M2 inhibe le TGFBR3 pour favoriser la progression du carcinome du nasopharynx et la croissance tumorale via la voie de signalisation TGF-β
Résumé
Objectifs
Le carcinome nasopharyngé (NPC) est un type de maladie nasopharyngée avec des propriétés métastatiques et d'invasion élevées. Il est prouvé que les macrophages activés alternatifs (M2) associés à la tumeur se connectent avec NPC. Sur cette base, cette étude vise à explorer le mécanisme et la participation du microARN-18a (miR-18a) des macrophages M2 dans NPC.
Méthodes
Les cellules mononucléées du sang périphérique ont été différenciées en macrophages et les macrophages ont été polarisés en type M2 par l'interleukine-4. Des cellules SUNE-1 et CNE2 ont été transfectées avec un miR-18a restauré ou appauvri ou un récepteur du facteur de croissance transformant bêta III (TGFBR3) pour explorer leurs rôles dans la progression des NPC avec l'implication de la voie de signalisation TGF-β. Ensuite, les cellules SUNE-1 et CNE2 ont été co-cultivées avec des macrophages M2 qui avaient été traités avec miR-18a ou TGFBR3 restaurés ou épuisés pour comprendre leurs rôles combinés dans NPC avec l'implication de la voie de signalisation TGF-β.
Résultats
MiR-18a était fortement exprimé et TGFBR3 était faiblement exprimé dans les cellules NPC. La restauration de MiR-18a, le knockdown de TGFBR3 ou la co-culture avec des mimes miR-18a, ou des macrophages M2 transfectés par si-TGFBR3 ont favorisé la progression des cellules SUNE-1, la croissance tumorale chez la souris, une diminution de p-Smad1/t-Smad1 et une augmentation de p- Smad3/t-Smad3. La régulation négative de miR-18a, la surexpression de TGFBR3 ou la co-culture avec des inhibiteurs de miR-18a ou des macrophages M2 transfectés par OE-TGFBR3 ont diminué la progression des cellules CNE2, la croissance tumorale chez la souris, une augmentation de p-Smad1/t-Smad1 et une diminution de p-Smad3/ t-Smad3.
Conclusion
Notre étude élucide que le miR-18a des macrophages M2 favorise la progression des cellules NPC et la croissance tumorale chez les souris nude via la répression de TGFBR3, ainsi que l'inactivation de Smad1 et l'activation de Smad3.
Introduction
Le carcinome du nasopharynx (CNP) est une tumeur épithéliale maligne qui est encline à l'infiltration locale et aux métastases précoces à distance [1]. Les patients NPC se plaignent souvent de paralysie du sixième nerf et du syndrome de Horner [2]. À l'heure actuelle, le traitement appliqué consiste principalement en une radiothérapie, et une radiothérapie et une chimiothérapie intégrées [3]. Malheureusement, la radiothérapie et la chimiothérapie s'accompagnent de complications inattendues, et la résistance acquise à la radiothérapie entrave les résultats de la NPC [3]. Étant donné que la tâche d'explorer une thérapie ciblée potentielle est une priorité.
Il est documenté que les microARN dérégulés (miARN) sont impliqués dans la tumorigenèse des NPC, les métastases, l'invasion et la résistance à la radiothérapie et à la chimiothérapie [1]. En tant que sous-famille de miARN, il s'avère que miR-18a facilite la progression des NPC par l'inhibition de la morphogenèse dans les organes génitaux 1 et l'activation de la voie mTOR [4]. En plus de cela, miR-18a est en outre vérifié pour activer la prolifération et la métastase des cellules NPC via la régulation DICER1 [5]. De plus, il est prouvé que la progression des cellules NPC est entraînée par miR-18a via une altération de la biogenèse des miARN [6]. De plus, il a été démontré que miR-18a fonctionnait de manière critique dans les métastases de NPC [7]. Les macrophages activés alternatifs (M2) sont des composants importants des malignités solides et hématologiques et sont liés à la progression, aux métastases et à la résistance au traitement [8]. Les macrophages associés aux tumeurs polarisées M2 sont associés à un mauvais pronostic de NPC [9]. Il est intéressant de noter les différences des macrophages M2 dans les NPC négatifs pour le virus d'Epstein-Barr et positifs pour le virus d'Epstein-Barr [10]. Il existe une étude décrivant que miR-18a inhibe les métastases hépatiques des cellules cancéreuses du côlon en induisant les macrophages M1 [11]. Le récepteur du facteur de croissance transformant bêta III (TGFBR3) est un co-récepteur du TGF-β qui fournit le ligand du récepteur TGF-β de type II pour stimuler la signalisation et l'équilibre de la surface cellulaire, et le TGFBR3 soluble est un régulateur au cours de la progression du cancer [12]. Il est rapporté que le TGFBR3 faiblement exprimé induit un microenvironnement tumoral immunotolérant [13]. A l'inverse, la surexpression transitoire de TGFBR3 induit l'apoptose dans les cellules NPC humaines [14]. À notre connaissance, la polarisation des macrophages M2 induite par miR-181a fait progresser la métastase des cellules tumorales médiées par les macrophages M2 via le facteur 6 de type Kruppel et l'axe α de la protéine de liaison CCAAT/enhancer [15].
Collectivement, bien que de nombreuses études aient découvert le rôle indépendant des macrophages miR-18a, TGFBR3 et M2 dans les NPC, les interactions combinées entre ces trois facteurs sont encore insaisissables. Considérant que cette étude est lancée pour décrypter les mécanismes et la participation de ces facteurs au NPC.
Matériaux et méthodes
Déclaration d'éthique
L'expérience a été approuvée par le comité d'éthique du Third Xiangya Hospital, Central South University et répondait aux normes d'éthique médicale. Cette étude a été menée avec le consentement écrit de tous les donneurs. Les expérimentations animales étaient conformes aux exigences des réglementations nationales sur la gestion et l'utilisation des animaux de laboratoire.
Collecte de cellules mononucléées du sang périphérique
Les monocytes ont été obtenus à partir de sang périphérique de donneurs sains par méthode adhérente. Des échantillons de sang périphérique ont été obtenus auprès de donneurs sains du département d'hématologie du troisième hôpital de Xiangya, Université Central South. Les macrophages dérivés des monocytes ont été obtenus par adhérence plastique de cellules mononucléées du sang périphérique (PBMC) préalablement isolées par centrifugation en gradient de densité (Ficoll-Paque, GE Healthcare) à partir de préparations de couche leucocytaire de sang de donneurs sains. Ensuite, 2,0 × 10 6 Les PBMC ont été cultivées dans du milieu Eagle modifié de Dulbecco (DMEM) additionné de 10 % de sérum humain (Millipore, Bedford, MA, USA) et de pénicilline/streptomycine sur des plaques 12 puits (NalgeNunc, NY, USA). Lorsque les PBMC ont été collées à la paroi pendant 2 à 3 h, le surnageant et les PBMC en suspension ont été retirés à plusieurs reprises pour obtenir des monocytes adhérents.
Polarisation des macrophages
Les monocytes ont été induits à se différencier en macrophages par le facteur de stimulation des colonies de macrophages humains (M-CSF) et polarisés en macrophages M2 par l'interleukine (IL)-4.
Induction de macrophages
Les monocytes ont été cultivés dans 20 % de sérum bovin foetal (FBS)-DMEM et ajoutés avec du M-CSF (100 ug, Peprotech, NJ, USA) à la concentration finale de 100 ng/mL. Le milieu a été renouvelé par moitié tous les 3 jours puis complété par 100 ng/mL de M-CSF. Cultivées jusqu'au 7-8ème jour, une partie des cellules a été récoltée et les marqueurs de surface des macrophages CD68 [16], CD163 [17] et CD206 [18] ont été testés par immunofluorescence [19] pour identifier les macrophages.
Polarisation des macrophages
Les macrophages ont été polarisés en macrophages M2 en ajoutant 20 ng/mL d'IL-4 (Peprotech) au milieu de différenciation pendant 24 heures supplémentaires. Une partie des échantillons de macrophages M2 a été utilisée pour la détection par cytométrie en flux. Les échantillons ont été divisés en 3 tubes :le tube 1 était le même lot de macrophages adhérents sans stimulation IL-4; les tubes 2 et 3 étaient les macrophages adhérents stimulés par l'IL-4. Les échantillons ont été dilués à environ 10 000 cellules lors du chargement et ajoutés avec de l'immunoglobuline G non spécifique pour bloquer le récepteur Fc. Ensuite, les échantillons ont été ajoutés avec un anticorps d'isotype non spécifique, un anticorps CD68 marqué au PE et un anticorps CD163 marqué au PE (tous deux de Biolegend, CA, USA). Incubés pendant 30 min et rincés avec 0,5% d'albumine de sérum bovin dans une solution saline tamponnée au phosphate (PBS), les échantillons ont été centrifugés et transformés en suspension cellulaire avec 500 L de PBS pour la détection.
Réaction en chaîne par polymérase quantitative par transcription inverse
Une amplification en chaîne par polymérase quantitative par transcription inverse (RT-qPCR) a été utilisée pour détecter miR-18a, CCL22, le récepteur activé par les proliférateurs de peroxysomes γ (PPAR-γ) et l'expression de l'ARNm du TGFBR3 dans les cellules collectées.
L'ARN total a été extrait des cellules par Trizol (Invitrogen, Carlsbad, CA, USA) et inversement transcrit en ARN complémentaire par Mir-X miRNA First Strand Synthesis Kit (Clontech, Mountain View, CA, USA) pour miR-18a et le PrimeScriptTM RT Kit Master Mix (Takara, Dalian, Chine) pour CCL22, PPAR-γ et TGFBR3. U6 et la glycéraldéhyde-3-phosphate déshydrogénase (GAPDH) étaient des témoins de charge pour miR-18a, CCL22, PPAR-γ et TGFBR3. SYBR @ Le prémélange Ex Taq™ II (Perfect Real Time) (Takara) dans un système LightCycler 480 II (Roche Diagnostics, Indiana, USA) a été utilisé en PCR. Le calcul des données a été évalué par 2 -△△CT méthode. Les amorces de PCR ont été présentées dans le tableau 1.
Dosage Western Blot
Le test Western blot a été appliqué à la détection de la protéine TGFBR3, totale (t)-Smad1, phosphorylée (p)-Smad1, t-Smad3 et p-Smad3 dans les cellules collectées.
La protéine totale des cellules a été extraite et la concentration en protéine a été déterminée sur la base du kit d'acide bicinchoninique. L'échantillon de protéine a été chargé dans les puits dans une électrophorèse sur gel de polyacrylamide dodécyl sulfate de sodium et transféré sur une membrane de fluorure de polyvinylidène (PVDF). La membrane PVDF a été bloquée avec du lait écrémé et incubée avec des anticorps primaires TGFBR3 (1:2000, R&D Systems, Minneapolis, MN, USA), t-Smad1 (1:1000), p-Smad1 (1:1000, Santa Cruz Biotechnology) , t-Smad3 (1:1000), p-Smad3 (1:1000) et GAPDH (1:1000, tous de Abcam, Cambridge, MA, UK) qui a été suivie d'une incubation avec l'anticorps secondaire marqué à la peroxydase de raifort ( 1:500, Jackson ImmunoResearch Laboratories, PA, États-Unis). Lavée 3 fois par une solution saline tris-tamponnée avec du Tween 20, la membrane a été développée par chimiluminescence renforcée. La quantification des signaux a été réalisée par les National Institutes of Health ImageJ Imaging. Logiciel d'analyse de traitement avec intensité de signalisation normalisée à GAPDH.
Culture cellulaire et criblage
Les lignées cellulaires humaines NPC CNE2, TW03, C666-1 et SUNE-1 et la lignée cellulaire nasopharyngée humaine normale NP96 (Shanghai Institutes for Biological Sciences, Chinese Academy of Sciences, Shanghai, Chine) ont été cultivées au Roswell Park Memorial Institute (RPMI) - Milieu 1640 (Gibco, CA, USA) contenant du FBS (Gibco), 100 g/mL de pénicilline et 100 g/mL de streptomycine et repiqué à 80% de confluence. La RT-qPCR a été utilisée pour détecter l'expression de miR-18a. Parmi ces lignées cellulaires NPC, les cellules CNE2 et SUNE-1 ont montré la différence la plus grande et la plus petite dans l'expression de miR-18a par rapport aux cellules NP96, elles ont donc été sélectionnées pour les tests de régulation à la baisse ou à la hausse de miR-18a.
Regroupement et traitement des cellules
Parmi toutes les lignées cellulaires NPC, les cellules SUNE-1 présentant la plus petite différence par rapport aux cellules NP96 dans l'expression de miR-18a ont été sélectionnées. Guidées par les spécifications de la Lipofectamine 2000 (Invitrogen), les cellules SUNE-1 ont été transfectées avec miR-18a mimics, miR-18a mimics negative control (NC), si-TGFBR3 ou si-TGFBR3 NC.
Parmi toutes les lignées cellulaires NPC, les cellules CNE2 présentant la plus grande différence par rapport aux cellules NP96 dans l'expression de miR-18a ont été sélectionnées et transfectées avec des inhibiteurs de miR-18a, des inhibiteurs de miR-18a NC, une surexpression (OE)-TGFBR3 ou OE-TGFBR3 NC par Lipofectamine 2000 (Invitrogen).
Guidés par les spécifications de la Lipofectamine 2000 (Invitrogen), les macrophages M2 ont été transfectés avec des miR-18a mimics, miR-18a mimics NC, si-TGFBR3, si-TGFBR3 NC, des inhibiteurs miR-18a, des inhibiteurs miR-18a NC, OE-TGFBR3, ou OE-TGFBR3 NC.
Co-culture de macrophages M2 et de cellules NPC
La co-culture cellulaire dans la chambre Transwell a été adoptée pour explorer les effets du miARN du macrophage M2 sur les cellules NPC. La chambre supérieure était remplie de macrophage M2 dont la taille des pores était de 0,4 um, ce qui n'empêchait que les cellules de la chambre supérieure de traverser, mais pas les petites molécules sécrétées par les cellules telles que les vésicules, les facteurs de croissance, les nutriments, etc. La chambre inférieure s'est propagé avec des cellules de PNJ.
SUNE-1 et CNE2 ont été incubés dans du FBS normal (Gibco). Les cellules en phase de croissance logarithmique ont été adoptées pour les expériences.
Les cellules SUNE-1 et CNE2 ont été co-cultivées avec des macrophages M2 dans du milieu 10 % FBS-RPMI-1640 (tous deux de Gibco) dans une boîte de culture cellulaire à insert Transwell (Coring, Corning, NY, USA) avec une taille de pores de 0,4 m .
Les cellules SUNE-1 n'ont pas été co-cultivées avec des macrophages M2, ni co-cultivées avec des macrophages M2, miR-18a imite les macrophages M2 transfectés, miR-18a imite les macrophages M2 transfectés par NC, les macrophages M2 transfectés si-TGFBR3 ou si -TGFBR3 macrophages M2 transfectés par NC.
Les cellules CNE2 n'ont pas été co-cultivées avec des macrophages M2, ni co-cultivées avec des macrophages M2, des macrophages M2 transfectés par des inhibiteurs de miR-18a, des macrophages M2 transfectés par des inhibiteurs de miR-18a, des macrophages M2 transfectés par OE-TGFBR3 ou OE-TGFBR3 Macrophages M2 transfectés par NC.
Dosage du bromure de 3-(4,5-diméthylthiazol-2-yl)-2, 5-diphényltétrazolium
La viabilité cellulaire a été testée par dosage du bromure de 3-(4,5-diméthylthiazol-2-yl)-2, 5-diphényltétrazolium (MTT), qui était un dosage colorimétrique appliqué pour déterminer l'activité de la déshydrogénase mitochondriale, qui a réduit le MTT en formazan.
Trypsinisé et ensemencé dans des plaques 96 puits à 4 × 10 4 cellules/puits, les cellules ont été privées de milieu de culture respectivement aux 0, 12, 24, 36 et 48 h et supplémentées en solution de MTT (500 μL, 0,5 g/L). Incubé pendant 4 h, le surnageant a été jeté et les cellules ont été incubées avec 200 µL de solution de diméthylsulfoxyde. Les valeurs de densité optique (DO, 490 nm) ont été mesurées sur un lecteur de microplaque (ELX808IU, BioTek, VT, USA). Chaque groupe a été mis en place avec 6 puits parallèles.
Test de formation de colonie
La capacité de formation de colonies des cellules NPC a été testée par un test de formation de colonies, qui reflétait la dépendance de la population cellulaire et la prolifération clonale cellulaire.
Cultivées pendant 24 h et détachées avec 0,25% de trypsine, 300 cellules ont été ensemencées dans une boîte de 35 mm avec 3 puits parallèles dans chaque groupe. Avec du milieu de culture renouvelé tous les 3 jours, les cellules ont été cultivées pendant une semaine et fixées avec 5 mL de paraformaldéhyde 4%. Après cela, les cellules ont été colorées par une solution de coloration cristal violet et séchées à l'air. La boite a été inversée sur laquelle un film transparent de grilles a été superposé, et le nombre de colonies (plus de 50 cellules) a été compté au microscope (Olympus, Tokyo, Japon).
Test de grattage
La migration cellulaire a été testée par scratch test. Les cellules ont été trypsinisées, ensemencées dans des plaques à 6 puits avec 3 puits parallèles pour chaque groupe et cultivées à 90 % de confluence. Ensuite, les cellules ont été incubées dans le milieu avec 2% de FBS et des rayures verticales ont été tracées par une pointe de 100 µL. Les cellules ont été photographiées les 0 et 24 h au microscope inversé pour mesurer la distance de migration cellulaire.
Test de Transwell
L'invasion et la migration cellulaires ont été testées par test Transwell. La chambre supérieure de la chambre Transwell a été pré-immergée et additionnée de 100 L de Matrigel (Coring) qui avait été dilué par du milieu RPMI 1640 sans sérum à 1:100. Les chambres supérieure et inférieure ont été additionnées de 200 L et 600 L de milieu RPMI 1640 sans sérum séparément. Par la suite, la chambre inférieure a été ajoutée avec 600 L de milieu RPMI 1640 contenant 10 % de FBS tandis que la chambre supérieure avec 200 L de suspension cellulaire (12,5 × 10 4 cellules/mL). Incubées pendant 40 h, les cellules ont été colorées par une solution de coloration cristal violet et essuyées avec un coton-tige pour compter les cellules passant à travers le Matrigel sous un microscope.
Cytométrie en flux
L'apoptose cellulaire et la distribution du cycle cellulaire ont été déterminées par cytométrie en flux.
La distribution du cycle cellulaire a été évaluée par coloration à l'iodure de propidium (PI). Les cellules ont été ensemencées dans des plaques à 6 puits à 4 × 10 5 cellules/puits, et cultivées à 70-80% de confluence. Fixées dans de l'éthanol à 70 % pré-refroidi pendant la nuit, les cellules ont été centrifugées (le surnageant a été jeté), ajoutées avec de l'ARNase (1 g/L, 200 L) et du Triton X-100 (2 L), et colorées par une solution de coloration PI pour 30 minutes. Après cela, la distribution du cycle cellulaire a été détectée par un cytomètre en flux (BD Bioscience, NJ, États-Unis) à 488 nm selon différentes intensités de fluorescence cellulaire à chaque phrase (phrase G0/G1, phrase S et phrase G2/M).
L'apoptose cellulaire a été mesurée par l'isothiocyanate d'annexine V-fluorescéine (FITC) et une double coloration PI. Les cellules ont été remises en suspension dans 500 L de tampon de liaison et colorées par 5 L de solution de coloration Annexine V-FITC et 10 L de solution PI. L'apoptose cellulaire a également été testée par un cytomètre en flux (BD Bioscience) dans les 30 minutes sans exposition à la lumière. Sur le nuage de points, les cellules vivantes dans le quadrant inférieur gauche (Q4) étaient FITC − /PI − , les cellules apoptotiques au stade précoce dans le quadrant inférieur droit (Q3) étaient FITC + /PI − , et les cellules nécrotiques et apoptotiques au stade tardif dans le quadrant supérieur droit (Q2) étaient FITC + /PI + . Taux d'apoptose =pourcentage d'apoptose précoce (Q3) + pourcentage d'apoptose tardive (Q2).
Xénogreffes tumorales chez des souris nues
La croissance tumorale a été observée en établissant un modèle de NPC chez la souris nude. Détachées par 0,25% de trypsine, les cellules SUNE-1 et CNE2 en phase logarithmique ont été configurées en suspension unicellulaire à 5 × 10 7 cellules/mL. La suspension cellulaire (0,2 ml) a été injectée dans l'aisselle droite de souris par un micro-injecteur pour établir des modèles de souris. Les souris modélisées ont été élevées dans un environnement spécifique exempt d'agents pathogènes. A partir du 4ème j, une croissance tumorale a été observée et les souris ont été pesées tous les 4 jours. Des souris nude ont été euthanasiées le 20ème jour après l'injection et les tumeurs ont été réséquées, pesées par une balance électronique et photographiées.
Dosage du gène rapporteur double luciférase
Un système de gène rapporteur double luciférase a été adopté pour confirmer les sites de liaison de miR-18a et de la région non traduite en 3' (UTR) de l'ARNm de TGFBR3. Un site de prédiction biologique (http://www.microrna.org/microrna/home.do) a été utilisé pour analyser le gène cible de miR-18a, et découvrir l'existence du site de liaison complémentaire de miR-18a sur le 3′ UTR de TGFBR3. Le test du gène rapporteur double luciférase a été utilisé pour vérifier davantage si TGFBR3 était directement ciblé par miR-18a. Un type pmirGLO-TGFBR3-sauvage (WT) et un type pmirG-LO-TGFBR3-mutant (MUT) du site de liaison 3'UTR de TGFBR3 ont été construits. Les imitateurs TGFBR3-WT ou TGFBR3-MUT et miR-18a ou imitateurs NC ont été co-transfectés dans des cellules SUNE-1 et CNE2 par Lipofectamine 2000 (Invitrogen) et incubés pendant 48 h. Un kit de dosage de la luciférase (Promega, Madison, WI, USA) a été utilisé pour analyser les cellules.
Analyse statistique
Le logiciel statistique SPSS21.0 (IBM Corp. Armonk, NY, USA) a été utilisé pour l'analyse. Les données ont été exprimées en moyenne ± écart type. Les différences entre deux groupes ont été analysées par t tester les différences entre plusieurs groupes par une analyse de variance à un facteur (ANOVA), suivie du test post hoc de Tukey. Une différence significative a été considérée à P <0,05.
Résultats
Identification des macrophages M2
Les monocytes enrichis par la méthode adhérente et les monocytes induits par le M-CSF ont été collectés à partir de sang périphérique de donneurs sains. La détection par immunofluorescence de CD68, CD206 et CD163 a confirmé que les PBMC induites par le M-CSF in vitro se sont transformées en macrophages avec des caractéristiques moléculaires typiques, qui répondent à nos exigences (Fig. 1a).
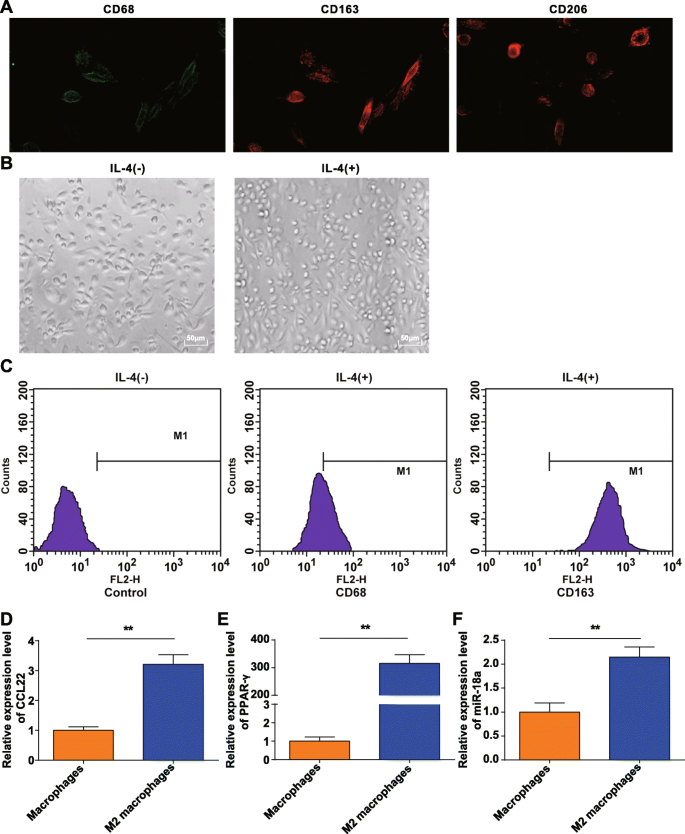
Identification des macrophages M2. un . CD68, CD206 et CD163 ont été exprimés à la surface de macrophages obtenus après induction de monocytes in vitro. b Les macrophages polarisés par l'IL-4 étaient des macrophages M2. c CD163 était fortement exprimé et CD68 était faiblement exprimé dans les macrophages M2 polarisés par l'IL-4. d CCL22 était fortement exprimé dans les macrophages M2. e PPAR-γ était fortement exprimé dans les macrophages M2. f miR-18a était fortement exprimé dans les macrophages M2 ; *P <0,05 ; **P <0,01. Les données de mesure ont été exprimées en moyenne ± écart type, N =3. Les comparaisons entre deux groupes ont été analysées par t tester
Les macrophages obtenus ont été polarisés par IL-4 et observés au microscope pour la morphologie. Il s'est avéré que les macrophages (M0) sans stimulation par l'IL-4 étaient divers et irréguliers, montrant une forme ronde, ovale ou fusiforme. Stimulés par l'IL-4, les macrophages M2 sont devenus plus gros et se sont principalement tournés vers une forme ronde, ce qui était conforme aux caractéristiques morphologiques des macrophages M2 décrites précédemment [20] (Fig. 1b).
La cytométrie en flux a testé les antigènes de surface des cellules adhérentes stimulées par 20 ng/mL d'IL-4 pendant 24 h et a constaté que CD68 était exprimé à 21,16% tandis que CD163 était à 98,69 % du nombre total de cellules (Fig. 1c), confirmant les cellules adhérentes étant des macrophages M2. La RT-qPCR a révélé qu'avec les cellules M0 en revanche, CCL22 et PPAR-γ (molécules polarisantes typiques) augmentaient dans les macrophages M2 (Fig. 1d, e), indiquant l'induction réussie des macrophages M2.
La RT-qPCR a également manifesté que l'expression de miR-18a augmentait dans les macrophages M2 contrairement aux macrophages M0 (P ˂ 0,05) (Fig. 1f).
MiR-18a est fortement exprimé et TGFBR3 est mal exprimé dans les cellules NPC
Le site Web de bioinformatique (miRanda) a prédit que les cibles potentielles de miR-18a et TGFBR3 étaient considérées comme une cible de miR-18a (Fig. 2a). Le test du gène rapporteur double luciférase a été mis en œuvre pour vérifier que miR-18a ciblait 3'UTR de TGFBR3. TGFBR3-WT ou TGFBR3-MUT ont été clonés dans le vecteur pmirGLO et co-transfectés avec des mimes miR-18a ou NC dans des cellules SUNE-1 et CNE2. Les imitateurs de miR-18a n'avaient aucune influence sur l'activité luciférase de TGFBR3 3′UTR-MUT mais ont altéré celle de TGFBR3 3′UTR-WT dans les cellules SUNE-1 et CNE2, suggérant que TGFBR3 était un gène cible régulé par miR-18a (Fig. . 2b).
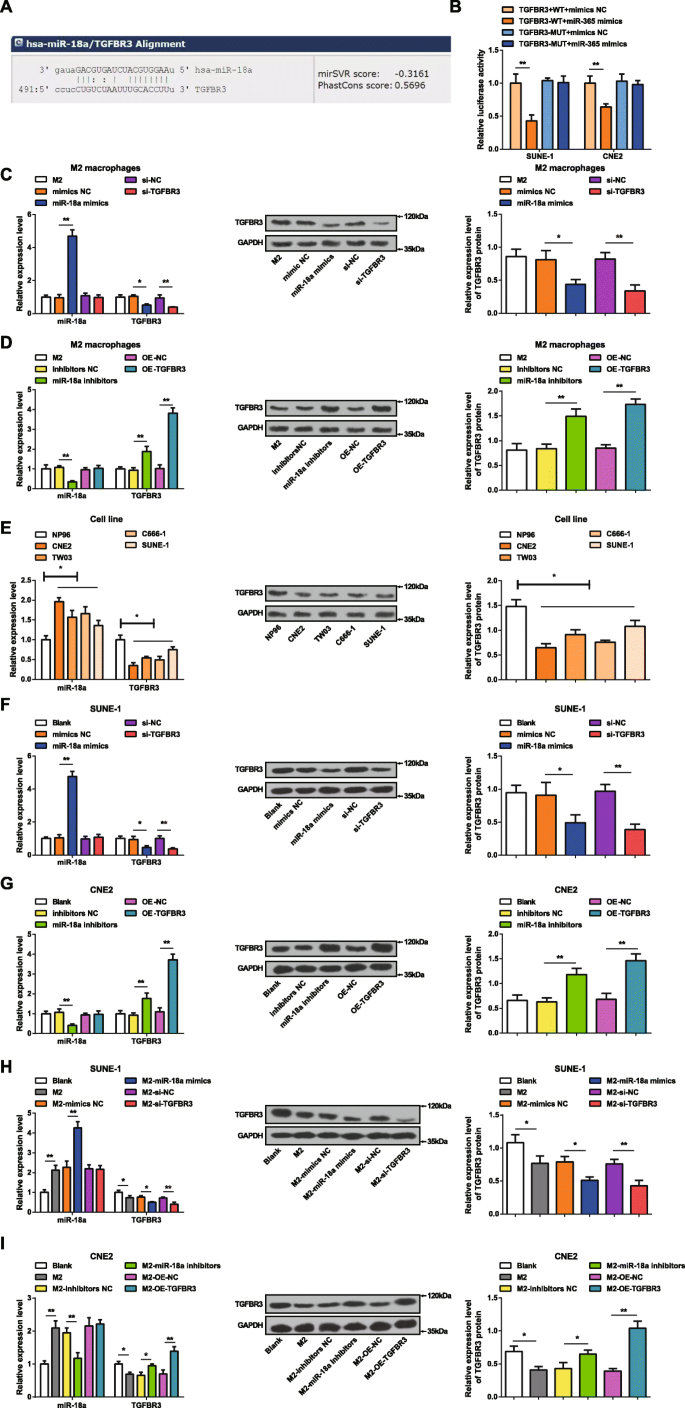
MiR-18a est fortement exprimé et TGFBR3 est faiblement exprimé dans les cellules NPC. un miRanda a prédit miR-18a ciblant TGFBR3. b Le test du gène rapporteur double luciférase a vérifié le miR-18a ciblant TGFBR3. c L'expression de miR-18a était élevée et l'expression de TGFBR3 était diminuée dans les macrophages M2 transfectés miR-18a. d L'expression de miR-18a était diminuée et l'expression de TGFBR3 était élevée dans les macrophages M2 transfectés par des inhibiteurs de miR-18a. e L'expression de miR-18a était élevée et l'expression de TGFBR3 était diminuée dans les lignées cellulaires NPC par rapport aux cellules NP96. f L'expression de miR-18a était élevée et l'expression de TGFBR3 était diminuée dans les cellules SUNE-1 transfectées miR-18a. g L'expression de miR-18a était diminuée et l'expression de TGFBR3 était élevée dans les cellules CNE2 transfectées par des inhibiteurs de miR-18a. h L'expression de miR-18a était élevée et l'expression de TGFBR3 était diminuée dans les cellules SUNE-1 co-cultivées avec des macrophages M2 transfectés miR-18a. je L'expression de miR-18a était diminuée et l'expression de TGFBR3 était élevée dans les cellules CNE2 co-cultivées avec des macrophages M2 transfectés par des inhibiteurs de miR-18a; *P <0,05 ; **P <0,01. Les données de mesure ont été exprimées en moyenne ± écart type, N =3. Les comparaisons entre deux groupes ont été analysées par t test. Les comparaisons entre plusieurs groupes ont été analysées par ANOVA à un facteur, suivie du test post hoc de Tukey
L'efficacité de la transfection des imitateurs miR-18a ou des inhibiteurs de miR-18a dans les macrophages M2 s'est manifestée par la détermination de l'expression de miR-18a et de TGFBR3 dans les macrophages M2 par RT-qPCR et test Western blot. Il était évident que (Fig. 2c, d) la surexpression de miR-18a augmentait l'expression de miR-18a et réduisait l'expression de TGFBR3 dans les macrophages M2. À l'opposé, l'inhibition de miR-18a a réduit l'expression de miR-18a et augmenté l'expression de TGFBR dans les macrophages M2. Aucune différence n'a été observée dans l'expression de miR-18a tandis que l'expression de TGFBR3 a diminué lorsque les macrophages M2 ont été transfectés avec si-TGFBR3. Aucune différence n'a été observée dans l'expression de miR-18a tandis que l'expression de TGFBR3 s'est développée lors de la transfection d'OE-TGFBR3 aux macrophages M2.
L'expression de miR-18a et TGFBR3 dans les lignées cellulaires CNE2, TW03, C666-1, SUNE-1 et NP96 a été testée par RT-qPCR et Western blot. Dans les cellules NPC, par comparaison avec les cellules NP96, l'expression de miR-18a a augmenté et l'expression de TGFBR3 a diminué (Fig. 2e). En raison des cellules CNE2 et des cellules SUNE-1 se manifestant avec les différences les plus importantes et les plus petites dans l'expression de miR-18a par rapport aux cellules NP96, elles ont été sélectionnées pour les tests de régulation à la baisse et à la hausse des miARN, respectivement.
Pour identifier les effets de miR-18a et TGFBR3 sur les cellules NPC, des cellules SUNE-1 ont été transfectées avec des mimes miR-18a ou si-TGFBR3 tandis que des cellules CNE2 avec des inhibiteurs miR-18a ou OE-TGFBR3. La RT-qPCR et le test Western blot ont démontré que miR-18a imite l'expression élevée de miR-18a et réduit l'expression de TGFBR3 dans les cellules SUNE-1. La transfection de si-TGFBR3 n'a eu aucun effet sur l'expression de miR-18a tout en réduisant l'expression de TGFBR3 dans les cellules SUNE-1. Les inhibiteurs de miR-18a ont diminué l'expression de miR-18a et ont augmenté l'expression de TGFBR3 dans les cellules CNE2. La transfection d'OE-TGFBR3 dans les cellules CNE2 n'a pas affecté l'expression de miR-18a mais a augmenté l'expression de TGFBR3 (Fig. 2f, g).
Pour explorer les effets de miR-18a à partir de macrophages M2 sur des cellules NPC, des macrophages M2 transfectés miR-18a ou si-TGFBR3 ou des inhibiteurs de miR-18a ou des macrophages M2 transfectés OE-TGFBR3 ont été co-cultivés avec SUNE-1 ou des cellules CNE2 dans la chambre Transwell, respectivement. La RT-qPCR et le test Western blot ont testé l'expression de miR-18a et TGFBR3 dans les cellules SUNE-1 ou CNE2. Des cellules SUNE-1 co-cultivées avec des macrophages M2 non transfectés ou transfectés miR-18a ont démontré une expression de miR-18a élevée et une expression de TGFBR3 dégradée. Aucune différence n'a été remarquée dans l'expression de miR-18a et l'expression de TGFBR3 réduite dans les cellules SUNE-1 qui avaient co-cultivé avec des macrophages M2 transfectés par si-TGFBR3 (Fig. 2h). Après co-culture avec des macrophages M2 non transfectés, les cellules CNE2 présentaient une expression accrue de miR-18a et une expression réduite de TGFBR3. Cependant, une expression réduite de miR-18a et une expression élevée de TGFBR3 se sont présentées dans des cellules CNE2 qui ont été préalablement co-cultivées avec des macrophages M2 transfectés par des inhibiteurs de miR-18a. Aucune différence n'a été reconnue dans l'expression de miR-18a et l'expression de TGFBR3 a augmenté dans les cellules CNE2 co-cultivées avec des macrophages M2 transfectés par OE-TGFBR3 (Fig. 2i).
miR-18a de M2 Macrophages favorise la viabilité des cellules NPC et la capacité de formation de colonies
Le test MTT et le test de formation de colonies ont été appliqués pour identifier les impacts de miR-18a et TGFBR3 sur la viabilité et la capacité de formation de colonies des cellules SUNE-1 et des cellules CNE2. Les mimes miR-18a, les inhibiteurs miR-18a, si-TGFBR3 ou OE-TGFBR3 ont été transfectés dans des cellules SUNE-1 ou des cellules CNE2. Il a été indiqué que dans les cellules SUNE-1, la régulation à la hausse de miR-18a ou la régulation à la baisse de TGFBR3 améliorait la viabilité cellulaire et le nombre de colonies élevé (Fig. 3a, e). Dans les cellules CNE2, la viabilité cellulaire était altérée et le nombre de colonies était réduit par l'inhibition de miR-18a ou la surexpression de TGFBR3 (Fig. 3b, f).
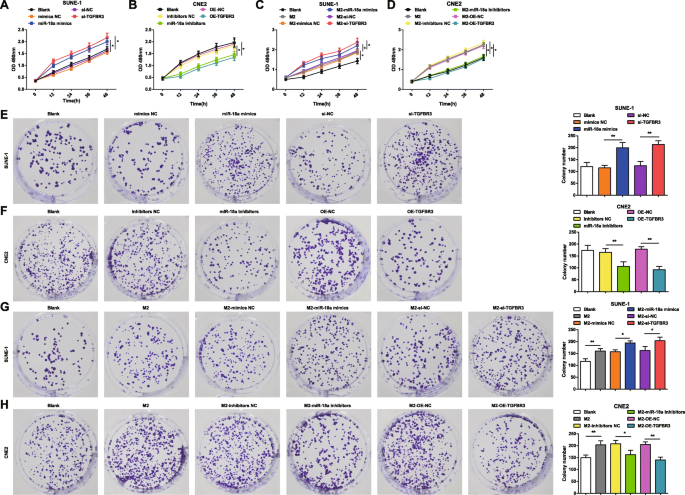
miR-18a des macrophages M2 induit la viabilité des cellules NPC et la capacité de formation de colonies. un miR-18a imite ou si-TGFBR3 a augmenté la viabilité des cellules SUNE-1. b Les inhibiteurs de miR-18a ou OE-TGFBR3 ont diminué la viabilité des cellules CNE2. c La co-culture avec miR-18a imite les macrophages M2 transfectés ou si-TGFBR3 a augmenté la viabilité des cellules SUNE-1. d La co-culture avec des inhibiteurs de miR-18a ou des macrophages M2 transfectés par OE-TGFBR3 a diminué la viabilité des cellules CNE2. e miR-18a imite ou si-TGFBR3 a augmenté le nombre de colonies de cellules SUNE-1. f Les inhibiteurs de miR-18a ou OE-TGFBR3 ont diminué le nombre de colonies de cellules CNE2. g La co-culture avec des macrophages M2 miR-18a ou transfectés par si-TGFBR3 a augmenté le nombre de colonies de cellules SUNE-1. h La co-culture avec des inhibiteurs de miR-18a ou des macrophages M2 transfectés par OE-TGFBR3 a diminué le nombre de colonies de cellules CNE2 ; *P <0,05 ; **P <0,01. Les données de mesure ont été exprimées en moyenne ± écart type, N =3. Les comparaisons entre plusieurs groupes ont été analysées par ANOVA à un facteur, suivie du test post hoc de Tukey
Pour examiner les effets de miR-18a du macrophage M2 sur la viabilité et la capacité de formation de colonies de cellules NPC, des macrophages M2 transfectés miR-18a, inhibiteurs miR-18a, si-TGFBR3- ou OE-TGFBR3 ont été co-cultivés avec des cellules SUNE-1 ou des cellules CNE2 dans la chambre Transwell. Le MTT et le test de formation de colonies ont montré que les cellules SUNE-1 co-cultivées avec des macrophages M2 non transfectés, ou transfectés par miR-18a ou transfectés par si-TGFBR3 présentaient une viabilité cellulaire renforcée et un nombre accru de colonies (Fig. 3c, g) .
Les cellules CNE2 co-cultivées avec des macrophages M2 non transfectés ont été mises en évidence avec une viabilité cellulaire renforcée et des colonies accrues. Cependant, les cellules CNE2 co-cultivées avec des macrophages M2 transfectés par des inhibiteurs de miR-18a ou transfectés par OE-TGFBR présentaient une viabilité cellulaire et des colonies réduites (Fig. 3d, h).
miR-18a de M2 Macrophages favorise l'invasion des cellules NPC et les capacités de migration
Pour une meilleure compréhension de la façon dont miR-18a et TGFBR3 ont affecté la migration et l'invasion des cellules NPC, un test de rayure et un test Transwell ont été mis en œuvre. Les résultats ont révélé que les cellules SUNE-1 transfectées avec des mimes miR-18a ou si-TGFBR3 étaient caractérisées par une distance de migration cellulaire et des cellules d'invasion accrues (Figs. 4a et 5a).
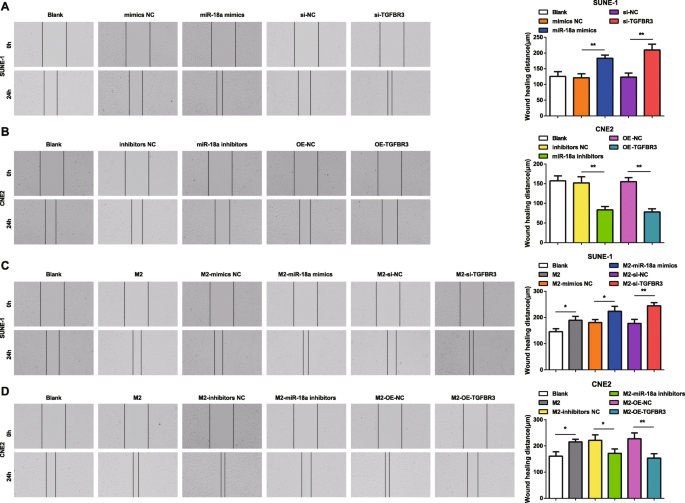
miR-18a des macrophages M2 favorise la capacité de migration des cellules NPC. un miR-18a imite ou si-TGFBR3 a augmenté la migration des cellules SUNE-1. b Les inhibiteurs de miR-18a ou OE-TGFBR3 ont diminué la migration des cellules CNE2. c Co-culture with miR-18a mimics- or si-TGFBR3-transfected M2 macrophages increased migration of SUNE-1 cells. d Co-culture with miR-18a inhibitors or OE-TGFBR3-transfected M2 macrophages decreased migration of CNE2 cells; *P <0.05; **P <0.01. Measurement data were expressed as mean ± standard deviation, N =3. Comparisons among multiple groups were analyzed by one-way ANOVA, followed by Tukey’s post hoc test
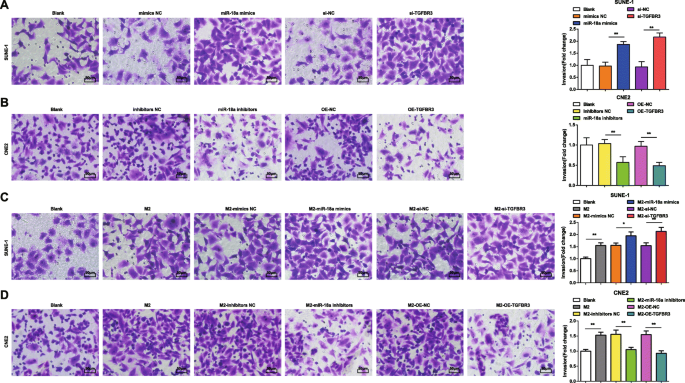
miR-18a from M2 macrophages promotes NPC cell invasion ability. un miR-18a mimics or si-TGFBR3 increased invasion of SUNE-1 cells. b miR-18a inhibitors or OE-TGFBR3 decreased invasion of CNE2 cells. c Co-culture with miR-18a mimics- or si-TGFBR3-transfected M2 macrophages increased invasion of SUNE-1 cells. b Co-culture with miR-18a inhibitors or OE-TGFBR3-transfected M2 macrophages decreased invasion of CNE2 cells; *P <0.05; **P <0.01. Measurement data were expressed as mean ± standard deviation, N =3. Comparisons among multiple groups were analyzed by one-way ANOVA, followed by Tukey’s post hoc test
In CNE2 cells transfected with miR-18a inhibitors or OE-TGFBR3, the reductions appeared in cell migration distance and invasion cells (Figs. 4b and 5b).
How miR-18a from M2 macrophages influenced invasion and migration abilities of NPC cells were deciphered by miR-18a mimics-, miR-18a inhibitors-, si-TGFBR3-, or OE-TGFBR3-transfected M2 macrophages co-culturing with SUNE-1 or CNE2 cells. The results highlighted that SUNE-1 cells co-cultured with untransfected, or miR-18a mimics-transfected or si-TGFBR3-transfected M2 macrophages were manifested with increased cell migration distance and invasion cells (Figs. 4c and 5c).
Both cell migration distance and invasion cells increased in CNE2 cells co-cultured with untransfected M2 macrophages. Upon co-culture with miR-18a inhibitors-transfected or OE-TGFBR3-transfected M2 macrophages, CNE2 cells were showed with reduced migration distance and invasion cells (Figs. 4d and 5d).
miR-18a from M2 Macrophages Arrests Fewer NPC Cell at G0/G1 Phase and Suppresses Apoptosis
Cell cycle distribution and apoptosis were tested by flow cytometry to stratify the effects of miR-18a and TGFBR3 on NPC cells. It was indicated that transfection of miR-18a mimics or si-TGFBR3 reduced SUNE-1 cells arrested in the G0/G1 phase, increased cells in the S and G2/M phases, and reduced cell apoptosis rate (Figs. 6a and 7a).
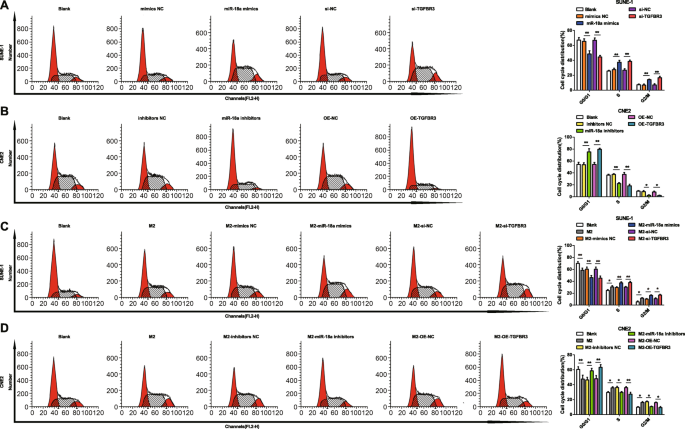
miR-18a from M2 macrophages arrests fewer NPC cell at G0/G1 phase. un miR-18a mimics or si-TGFBR3 decreased SUNE-1 cells in the G0/G1 phase, and increased SUNE-1 cells in the S and G2/M phases. b miR-18a inhibitors or OE-TGFBR3 increased CNE2 cells in the G0/G1 phase, and decreased CNE2 cells in the S and G2/M phases. c Co-culture with miR-18a mimics- or si-TGFBR3-transfected M2 macrophages decreased SUNE-1 cells in the G0/G1 phase, and increased SUNE-1 cells in the S and G2/M phases. d Co-culture with miR-18a inhibitors or OE-TGFBR3-transfected M2 macrophages increased CNE2 cells in the G0/G1 phase, and decreased CNE2 cells in the S and G2/M phases; *P <0.05; **P <0.01. Measurement data were expressed as mean ± standard deviation, N =3. Comparisons among multiple groups were analyzed by one-way ANOVA, followed by Tukey’s post hoc test
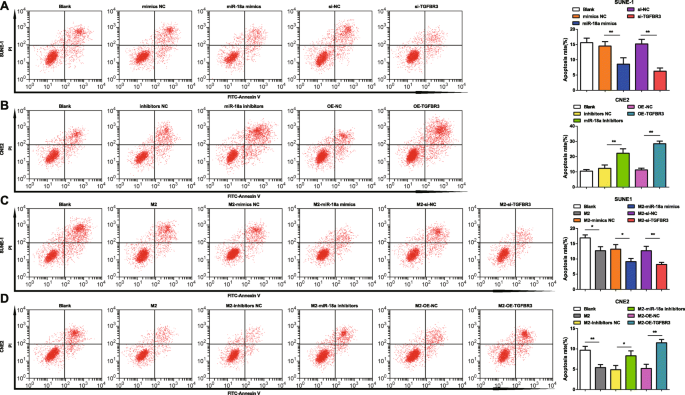
miR-18a from M2 macrophages inhibits NPC cell apoptosis. un miR-18a mimics or si-TGFBR3 decreased apoptosis of SUNE-1 cells. b miR-18a inhibitors or OE-TGFBR3 increased apoptosis of CNE2 cells. c Co-culture with miR-18a mimics- or si-TGFBR3-transfected M2 macrophages decreased apoptosis of SUNE-1 cells. d Co-culture with miR-18a inhibitors or OE-TGFBR3-transfected M2 macrophages increased apoptosis of CNE2 cells; *P <0.05; **P <0.01. Measurement data were expressed as mean ± standard deviation, N =3. Comparisons among multiple groups were analyzed by one-way ANOVA, followed by Tukey’s post hoc test
Upon transfection with miR-18a inhibitors or OE-TGFBR3, CNE2 cells in the G0/G1 phase trended toward an elevation while those in the S and G2/M phases toward a reduction, and cell apoptosis rate raised (Figs. 6b and 7b).
With the purpose to decode the mechanism of miR-18a from M2 macrophages in NPC cell cycle distribution and apoptosis, M2 macrophages transfected with miR-18a mimics, miR-18a inhibitors, si-TGFBR3, or OE-TGFBR3 were co-cultured with SUNE-1 cells or CNE2 cells in the Transwell chamber. Co-cultured with untransfected, or miR-18a mimics-transfected or si-TGFBR3-transfected M2 macrophages, reduced SUNE-1 cells were displayed in the G0/G1 phase and increased cells in the S and G2/M phases, and SUNE-1 cell apoptosis rate decreased (Figs. 6c and 7c).
Co-cultured with untransfected M2 macrophages, CNE2 cells in the G0/G1 phase reduced, cells in the S and G2/M phases increased, and apoptosis rate declined. On the contrary, co-cultured with miR-18a inhibitors-transfected or OE-TGFBR3-transfected M2 macrophages, CNE2 cells in the G0/G1 phase elevated while those in the S and G2/M phases decreased, and apoptosis rate elevated (Figs. 6d and 7d).
miR-18a from M2 Macrophages Reduces p-Smad1/t-Smad1 and Elevates p-Smad3/t-Smad3 in NPC Cells
Western blot assay detected TGF signaling pathway-related proteins in NPC cells to further explain the effects of miR-18a and TGFBR3 on TGF signaling pathway.
It was explained that transfection of miR-18a mimics or si-TGFBR3 reduced p-Smad1/t-Smad1 and elevated p-Smad3/t-Smad3 in SUNE-1 cells (Fig. 8a).
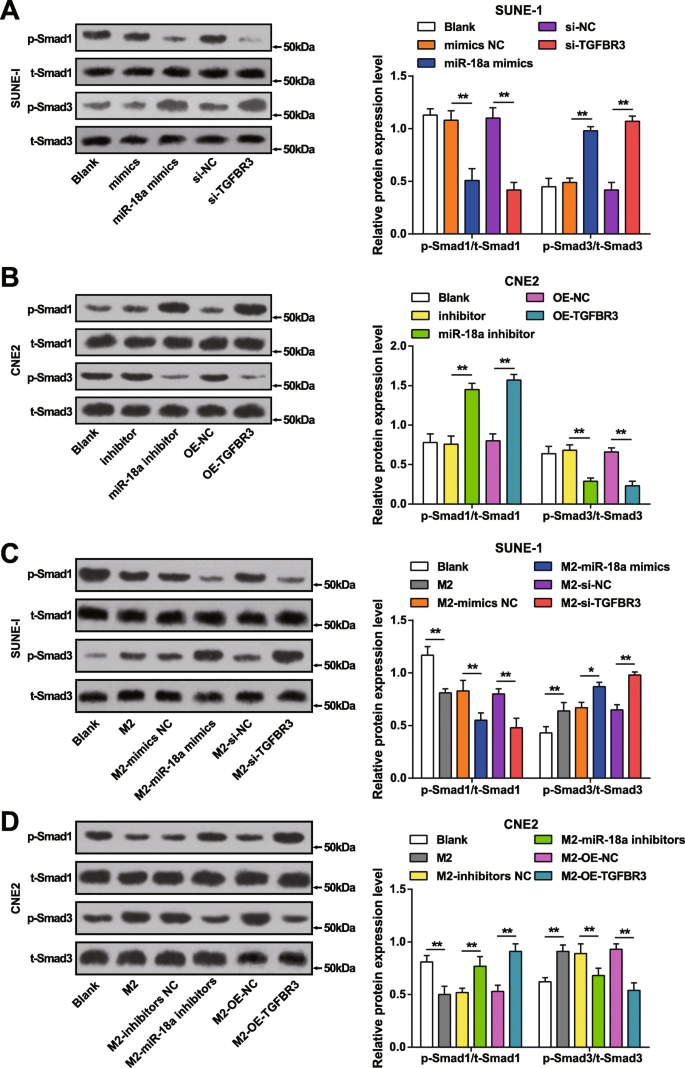
miR-18a from M2 macrophages decreases p-Smad1/t-Smad1 and increases p-Smad3/t-Smad3 in NPC cells. un miR-18a mimics or si-TGFBR3 in SUNE-1 cells decreased p-Smad1/t-Smad1 and elevated p-Smad3/t-Smad3. b miR-18a inhibitors or OE-TGFBR3 in CNE2 cells increased p-Smad1/t-Smad1 and decreased p-Smad3/t-Smad3. c Co-culture with miR-18a mimics- or si-TGFBR3-transfected M2 macrophages decreased p-Smad1/t-Smad1 and elevated p-Smad3/t-Smad3. d Co-culture with miR-18a inhibitors or OE-TGFBR3-transfected M2 macrophages increased p-Smad1/t-Smad1 and decreased p-Smad3/t-Smad3; *P <0.05; **P <0.01. Measurement data were expressed as mean ± standard deviation, N =3. Comparisons among multiple groups were analyzed by one-way ANOVA, followed by Tukey’s post hoc test
Transfection with miR-18a inhibitors or OE-TGFBR3 led to declined p-Smad3/t-Smad3 and increased p-Smad1/t-Smad1 in CNE2 cells (Fig. 8b).
miR-18a from M2 macrophages influencing TGF signaling pathway in NPC cells was determined by Western blot assay through testing TGF signaling pathway-related proteins in SUNE-1 cells and CNE2 cells which had co-cultured with miR-18a mimics-, miR-18a inhibitors-, si-TGFBR3-, or OE-TGFBR3-transfected M2 macrophages in the Transwell chamber.
SUNE-1 cells co-cultured with untransfected, miR-18a mimics-transfected, or si-TGFBR3-transfected M2 macrophages were manifested with reduced p-Smad1/t-Smad1 and incremental p-Smad3/t-Smad3 (Fig. 8c).
Co-cultured with untransfected M2 macrophages, CNE2 cells trended toward declined p-Smad1/t-Smad1 and elevated p-Smad3/t-Smad3. In an opposite way, CNE2 cells were demonstrated with increased p-Smad1/t-Smad1 and decreased p-Smad3/t-Smad3 when co-cultured with miR-18a inhibitors-transfected or OE-TGFBR3-transfected M2 macrophages (Fig. 8d).
miR-18a from M2 Macrophages Induces Tumor Growth in Nude Mice with NPC
Tumor xenografts were conducted on nude mice to further elucidate the impacts of miR-18a and TGFBR3 on tumor growth of NPC.
It was indicated that injected with miR-18a mimics-transfected or si-TGFBR3-transfected SUNE-1 cells, mice were manifested with enlarged tumor volume and heavier tumor weight (Fig. 9a).
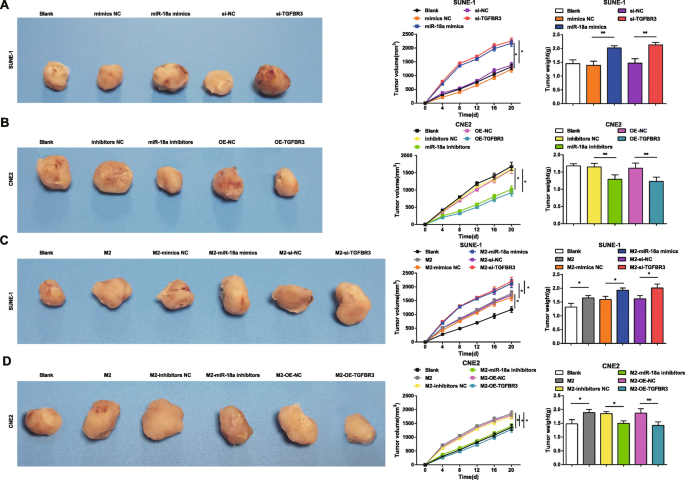
miR-18a from M2 macrophages promotes tumor growth in nude mice with NPC. un miR-18a mimics or si-TGFBR3 in SUNE-1 cells increased tumor volume and weight. b miR-18a inhibitors or OE-TGFBR3 in CNE2 cells decreased tumor volume and weight. c Co-culture with miR-18a mimics- or si-TGFBR3-transfected M2 macrophages increased tumor volume and weight. d Co-culture with miR-18a inhibitors or OE-TGFBR3-transfected M2 macrophages decreased tumor volume and weight; *P <0.05; **P <0.01. Measurement data were expressed as mean ± standard deviation, three nude mice in each group. Comparisons among multiple groups were analyzed by one-way ANOVA, followed by Tukey’s post hoc test
Reduced tumor volume and weight were presented in mice with injection of miR-18a inhibitors-transfected or OE-TGFBR3-transfected CNE2 cells (Fig. 9b).
Tumor growth was observed in mice which had injected with miR-18a mimics-, miR-18a inhibitors-, si-TGFBR3-, or OE-TGFBR3-transfected M2 macrophages to illustrate the mechanism of miR-18a from M2 macrophages in NPC.
After co-culture with untransfected, miR-18a mimics-transfected or si-TGFBR3-transfected M2 macrophages, SUNE-1 cells were injected into mice and mice were observed with larger tumor volume and heavier tumor weight (Fig. 9c).
CNE2 cells were co-cultured with M2 macrophages and injected into mice with the results suggesting growing tumor volume and weight. Both tumor volume and weight were inclined to reduce when mice were injected with CNE2 cells which had co-cultured with miR-18a inhibitors-transfected or OE-TGFBR3-transfected M2 macrophages (Fig. 9d).
Discussion
NPC refers to a polygenic disease threatened by a wide range of factors [21]. MiRNAs are previously implied to participate in the pathogenesis of NPC via regulation of their target genes which are indicators of cellular processes and pathways [22]. Concretely, miR-18a advances NPC progression by miRNA biogenesis impairing [6]. Given that this study goes forward to decipher the combined interactions of miR-18a from M2 macrophages and TGFBR3 in NPC with the conclusion elucidating that miR-18a from M2 macrophages stimulates NPC progression via TGFBR3 inhibition (Fig. 10).
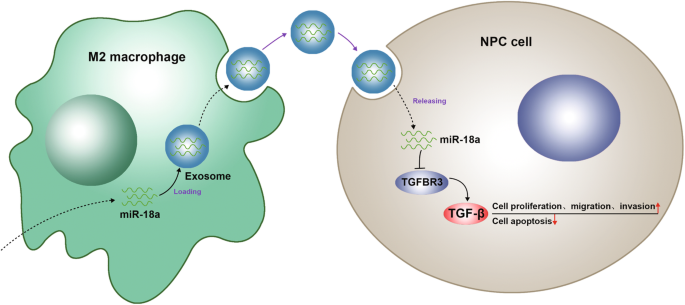
Schematic representation of macrophage-derived exosomal miR-18a in NPC and the involvement of TGFBR3-mediated TGF-β signaling pathway
At the start of this study, macrophages are stimulated by IL-4 to differentiate to M2 macrophages which are found to enrich the expression of miR-18a. As we know, during macrophage polarization, miRNA’s expression was altered [23]. In addition, M2 polarization enriches genes which are involved in the cell cycle and metabolic processes, and the M2 phenotype is conducive to tumor growth and angiogenesis in neoplastic tissues [24]. Based on the M2 macrophages-enriched miR-18a, a series of experiments were successfully conducted.
Initially, our study has uncovered that miR-18a is highly expressed while TGFBR3 is poorly expressed in NPC cells. Drawn from a previous study, it is concluded that miR-18a is overexpressed in NPC tissues with its association with lymph node metastasis and clinical stage [5]. Besides that, miR-17-92 cluster members including miR-18a are documented to be overexpressed in NPC tissues [25]. Furthermore, upregulated miR-18a is reported to demonstrate in NPC tissues which is connected with tumor node metastasis stage and tumor size [4]. Experimentally, except for the downregulated TGFBR3 in tongue squamous cell carcinoma [26], there has been another study depicting reduced TGFBR3 in clear-cell renal cell carcinomas accompanied by unwanted prognosis [27]. Anyhow, the results in this study are consistent with these study findings to some extent.
In order to explore the roles of miR-18a and TGFBR3 in NPC cell progression, we have conducted a series of assays with the results indicating that upregulated miR-18a or downregulated TGFBR3 triggers NPC cell progression while miR-18a repression or TGFBR3 elevation has the opposite effects on NPC cells. Widely, suppression of miR-18a is evidenced to hamper cell progression in malignancies including ovarian cancer, colitis-associated colorectal cancer, and hepatocellular carcinoma [28,29,30]. Narrowly, an existed study has pronounced that upregulated miR-18a promotes NPC cell progression via mediation of DICER1 [6]. In addition, it is noticed that overexpressed miR-18a in NPC is believed to connect with NPC metastasis and repressed miR-18a partially contributes to better prognosis of NPC patients [31]. Lately, it is surveyed that downregulation of miR-18a is capable of discouraging NPC proliferation, invasion, and migration [4]. Additionally, a decrease in TGFBR3 expression is regarded to link with laryngeal squamous cell carcinoma (LSCC) invasion and miR-223/TGFBR3 axis regulation takes part in LSCC progression inhibition [32]. TGFBR3 elevation is documented to restrict NPC cell viability, induce apoptosis, and activate pro-apoptosis signaling pathways [14]. Previously, a study has indicated that upregulation of TGFBR3 promotes apoptosis and cells arrested in the G2/M phase, resulting in impaired cell viability and migration in salivary gland adenoid cystic carcinoma [33]. Intriguingly, it is formerly described that induction of TGFBR3 contributes to disrupt intrahepatic cholangiocarcinoma progression [34].
Despite the protective role of lowly expressed miR-18a and overexpressed TGFBR3 in NPC cell in vitro, we have performed tumor xenografts in nude mince in vivo for further verification with the results explaining that miR-18a knockdown or TGFBR3 elevation restrains tumor growth in nude mice. As demonstrated in a prior study, miR-18a-injected nude mice show with enhanced tumor growth [5] and conversely, the miR-18a antagomir-injected nude mice are displayed with suppressed tumor growth in NPC [4]. In the light of the TGFBR3 reduction in tumor growth, it is suggested that poorly expressed TGFBR3 provokes tumor formation in clear-cell renal cell carcinoma [27]. In the opposite way, an increase in TGFBR3 is recognized to hinder tumor growth in lung cancer with the presence of long non-coding RNA ADAM metallopeptidase with thrombospondin type 1 motif, 9 antisense RNA 2 elevation, and miR-223-3p suppression [35]. This study has also predicted and verified that TGFBR3 is a target gene of miR-18a. But, more studies still need to be conducted for further verification.
Conclusion
Generally speaking, this study elaborates the concrete mechanisms that miR-18a from M2 macrophages inhibits TGFBR3 expression to exacerbate the progression of NPC via TGF-β signaling pathway, the results of which is abrogated by miR-18a knockdown or TGFBR3 elevation. This study updates the therapeutic target for NPC. However, a large cohort researches are still in need for in-depth explorations.
Disponibilité des données et des matériaux
Non applicable.
Nanomatériaux
- Composite hybride nanostructuré silice/or-cellulose Amino-POSS par procédé Sol-Gel et ses propriétés
- Amélioration de l'efficacité antitumorale et de la pharmacocinétique de la bufaline via les liposomes pégylés
- Préparation de nanosphères polymères à empreinte ionique de palladium(II) et son élimination du palladium(II) d'une solution aqueuse
- Nanoassemblages 5-aminolévulinique-squalène pour la photodétection et le traitement des tumeurs :études in vitro
- Promotion de la croissance cellulaire SH-SY5Y par des nanoparticules d'or modifiées avec de la 6-mercaptopurine et un peptide pénétrant dans les neurones
- Les nanoparticules de maghémite agissent comme des nanozymes, améliorant la croissance et la tolérance au stress abiotique chez Brassica napus
- Synthèse et caractérisation de BiOCl modifiés et leur application à l'adsorption de colorants à faible concentration à partir de solutions aqueuses
- Propriétés matérielles et optiques des points quantiques de carbone fluorescent fabriqués à partir de jus de citron par réaction hydrothermale
- Croissance MOCVD auto-amorcée et photoluminescence considérablement améliorée des nanofils InGaAs/InP Core–Shell



