Biocapteur ultrasensible pour la détection de l'ADN de Vibrio cholerae avec des nanosphères composites polystyrène-acide acrylique
Résumé
Un biocapteur électrochimique ultrasensible pour la détermination des Vibrio cholerae pathogènes (V . cholérae ) L'ADN a été développé sur la base d'une matrice de support d'ADN composite de nanosphères de latex et de nanoparticules d'or (PSA-AuNPs) de polystyrène-co-acide acrylique (PSA). Une voltampérométrie différentielle (DPV) utilisant un marqueur oligonucléotidique anthraquninone électroactif a été utilisée pour mesurer la réponse du biocapteur. Le chargement de nanoparticules d'or (AuNPs) sur l'électrode de particules d'ADN-latex a considérablement amplifié le courant faradique d'hybridation d'ADN. Avec l'utilisation d'une sonde rapportée, le biocapteur a démontré une sensibilité élevée. Le biocapteur d'ADN a donné une plage de réponse linéaire reproductible et large pour cibler l'ADN de 1,0 × 10 −21 à 1,0 × 10 −8 M (écart type relatif, RSD = 4,5 %, n = 5) avec une limite de détection (LOD) de 1,0 × 10 −21 M (R 2 = 0,99). Le biocapteur a obtenu des valeurs de récupération satisfaisantes entre 91 et 109% (n = 3) pour la détection de V . cholérae L'ADN dans les échantillons enrichis et pourrait être réutilisé pour six tests d'ADN consécutifs avec une valeur de répétabilité RSD de 5 % (n = 5). La réponse du biocapteur électrochimique était stable et maintenable à 95 % de sa réponse d'origine jusqu'à 58 jours de période de stockage.
Contexte
Vibrio cholerae , un agent pathogène d'origine alimentaire, peut provoquer des épidémies de choléra chez l'homme par le biais d'une diarrhée aqueuse aiguë. L'épidémie de choléra reste un problème grave dans certaines parties du monde, par ex. l'Asie et l'Afrique, et conduit à un faible statut socio-économique [1,2,3,4]. Cet agent pathogène entérique est une cause majeure de morbidité et de mortalité, en particulier dans les pays en développement [5]. L'épidémie et la pandémie de choléra dans diverses régions sont principalement causées par V. cholerae sérogroupes O1 et O139 [1, 2, 6]. V. cholerae Le sérogroupe O1 a deux sérotypes principaux, à savoir Inaba et Ogawa, qui peuvent alterner entre les épidémies de choléra. Le troisième sérotype, Hikojima, existe également mais est rare et instable. Les gènes responsables de la biosynthèse de l'antigène O1 ont été désignés comme rfb. La mutation qui définit les sérotypes Inaba et Ogawa est une mutation par délétion unique dans le gène rfbT [7]. Cependant, des épidémies occasionnelles d'origine alimentaire chez l'homme souffrant de diarrhée sévère ont également été signalées comme étant causées par le non-O1/non-O139 V. cholerae par l'ingestion de fruits de mer insuffisamment cuits [8] ou l'exposition à un environnement aquatique contaminé [9]. La première épidémie de V. cholerae O139 s'est produit en 1992 au Bangladesh et en Inde, puis s'est rapidement propagé à d'autres pays d'Asie du Sud-Est [10]. Dans le monde, en 2005, un total de 131 943 cas de choléra et 2 272 décès ont été signalés à l'Organisation mondiale de la santé (OMS) [11].
La recherche d'une méthode efficace de surveillance ou de diagnostic de V. cholerae bactérie est impératif pour contrôler l'épidémie de choléra. Identification traditionnelle de V. cholerae est souvent obtenu par l'isolement et le criblage des bactéries, où il implique un pré-enrichissement en eau peptonée alcaline (APW) suivi de l'isolement de V. cholerae sur milieu de culture thiosulfate citrate bile salt sucrose agar (TCBS) et identification par test d'agglutination sur lame avec des antisérums spécifiques [12]. Cependant, cette technique est très chronophage et laborieuse, et le résultat obtenu plusieurs jours plus tard aurait entraîné un retard dans le diagnostic clinique et le traitement du patient. Méthode moléculaire impliquant l'amplification PCR pour la détection de V. cholerae [13] a réduit le temps de diagnostic [14], mais la méthode PCR nécessite une infrastructure professionnelle qualifiée et coûteuse qui est difficile à mettre en œuvre dans les pays à faibles ressources. Des tests de diagnostic rapide basés sur le principe de l'immunochromatographie ont été rapportés pour la détection discrète ou simultanée de V. cholerae sérogroupes O1 et O139. Certaines autres techniques basées sur le dosage immunologique utilisées pour la détection de V. cholerae sont tels que le dosage immuno-enzymatique (ELISA), la coagglutination, l'immunofluorescence et la microbalance à cristaux de quartz (QCM). Cependant, la plupart de ces techniques nécessitent une instrumentation sophistiquée, une longue durée de dosage et un personnel hautement qualifié possédant des connaissances techniques détaillées [15,16,17,18,19,20].
Les méthodes électrochimiques ont attiré une attention considérable dans la détection des acides nucléiques en raison de leur haute sensibilité, spécificité, simplicité et protocole économique, ainsi que de leur détection rapide et compatible avec la technologie de microfabrication [21, 22]. De plus, une méthode électrochimique couplée à des technologies de miniaturisation peut être utilisée pour une analyse décentralisée in situ, par exemple, le dispositif de biocapteur d'ADN à puce microfluidique, qui est très utile pour un cadre pratique [23]. Il existe une large gamme d'électrodes utilisées dans les mesures électrochimiques telles que l'électrode en carbone vitreux (GCE), l'électrode en pâte de carbone (CPE), l'électrode en or et l'électrode en platine. Récemment, des études se sont concentrées sur l'utilisation d'électrodes sérigraphiées (SPE) en raison de certaines de leurs propriétés uniques, telles que la fourniture d'un faible courant de fond et d'une large fenêtre de potentiel, rentables car l'encre au carbone est peu coûteuse et peut être produite en série. .
Il existe quelques méthodes électrochimiques rapportées pour la détection de V. cholerae consistait en une série d'étapes complexes. Un V. cholerae génosensor rapporté par Liew et al. [24] ont utilisé la méthode d'adsorption électrochimique pour immobiliser la sonde d'ADN sur le carbone SPE. Les particules de PSA multicouches modifiées par AuNPs lyophilisées avec des polyélectrolytes ont formé un bioconjugué avec de l'avidine pour fonctionner comme marqueur rapporteur dans le test d'hybridation d'ADN sandwich. Cependant, l'ajout de stabilisateur de sorbitol était nécessaire pour préserver les bioconjugués PSA-AuNPs-avidine afin d'allonger la période opérationnelle du biocapteur d'ADN jusqu'à 30 jours. Enzymatique électrochimique V. cholerae Le biocapteur d'ADN a été récemment conçu par Yu et al. [25], où la sonde d'ADN marquée à la phosphatase alcaline conjuguée à l'anti-fluorescéine thiolée (anti-FCAP) était liée à l'or SPE par la chimie or-thiol. L'ADN cible a été marqué avec une fluorescéine universelle pour permettre la reconnaissance d'hybridation d'ADN obtenue via la conversion enzymatique du phosphate de -naphtyle en α-naphtol électroactif. Néanmoins, ce schéma de détection a nécessité un long temps de dosage d'environ 95 min pour l'hybridation de l'ADN, le marquage des hybrides d'ADN avec une enzyme fonctionnelle suivi d'une incubation de l'électrode dans un substrat de phosphate de -naphtyle électroinactif avant qu'une mesure ampérométrique puisse être effectuée. Un autre biocapteur d'ADN électrochimique enzymatique basé sur du carbone revêtu d'avidine conjugué à une sonde d'ADN biotinylée a été développé par Low et les membres de l'équipe [26]. Une sonde rapporteur marquée à la digoxigénine (DIG) a également été utilisée dans cette stratégie d'hybridation double qui flanquait la séquence d'ADNc. Un anti-DIG lié à la peroxydase de raifort (anti-DIG-HRP) a été utilisé comme marqueur électrochimique, qui pourrait simultanément catalyser l'oxydation de la 3,3′,5,5′-tétraméthylbenzidine (TMB) avec la réduction de H 2 O2 pour produire un transfert d'électrons à la surface de l'électrode pour la transduction électrochimique de l'événement d'hybridation d'ADN. Patel et al. [22] pour la détection rapide de V. cholerae , et le bleu de méthylène a été utilisé comme indicateur d'hybridation d'ADN. Cependant, la plage de détection linéaire du système est limitée aux niveaux μM, ce qui a limité son application dans les échantillons cliniques.
La nanoparticule de latex-or a été précédemment utilisée comme marqueur d'hybridation d'ADN via la liaison avidine/biotine à la sonde d'ADN dans la détection de V. cholerae [24], agent pathogène des poissons Aphanomyces invadans [27], E. coli [28] et l'hybridation d'ADN non spécifique [29], dans laquelle les sphères de latex ont été recouvertes d'une multicouche de polyélectrolyte avant que les colloïdes chargés négativement de nanoparticules d'or ne soient attachés électrostatiquement à celles-ci. Kawde et Wang [29] ont attaché les particules de latex de PSA à une sonde rapporteur d'ADN à utiliser comme marqueur d'hybridation d'ADN via le chargement de particules de latex recouvertes de streptavidine avec des AuNP recouvertes de biotine. Kuan et al. [24, 27] et Liew et al. [24, 27] ont également rapporté la même méthode de liaison avidine-biotine en utilisant des conjugués de sonde or-PSA-ADN. Pinijsuwan et al. [28] ont rapporté l'utilisation d'une méthode électrostatique pour le chargement des particules de PSA attachées à la sonde rapporteur d'ADN et les particules de PSA recouvertes d'or de polyélectrolyte ont été utilisées comme marqueur pour l'hybridation qui a amplifié la réponse de courant DPV.
Dans cette étude, nous rapportons une approche différente d'immobilisation d'ADN utilisant les nanoparticules de latex-or comme substrat d'immobilisation de sonde d'ADN pour développer un système de détection hautement sensible pour V. cholerae ADN. L'immobilisation de l'ADN a été réalisée avec une procédure très simple et rapide en utilisant la chimie du chlorhydrate de 1-éthyl-3-(3-diméthylaminopropyl) carbodiimide/N-hydroxysuccinimide (EDC/NHS) comme réactif de couplage pour améliorer l'efficacité d'immobilisation [30] sur le latex carboxylé. surface des particules. La détection d'hybridation d'ADN était basée sur le test de type sandwich, qui impliquait une réaction d'hybridation entre la sonde d'ADN immobilisée et la séquence cible suivie d'une sonde signal/rapporteur. Le sel de sodium monohydraté de l'acide anthraquinone-2-sulfonique (AQMS) a été utilisé comme marqueur électrochimique pour surveiller l'événement d'hybridation. Les particules de latex de taille submicronique proposées ont amélioré la capacité de liaison de la sonde d'ADN et la sensibilité du biocapteur d'ADN a été améliorée grâce à l'incorporation de nanoparticules d'or hautement conductrices (AuNP). Le biocapteur d'ADN a démontré une sensibilité exceptionnelle à la détection de V. cholerae ADNc et limite de détection extrêmement faible aux niveaux zeptomolaires par rapport à la technologie avidine-biotine signalés jusqu'à présent [24, 26].
Méthodes
Produits chimiques et réactifs
Le styrène (St) et l'acide acrylique (AA) ont été achetés auprès de Fluka. HAuCl4 ·3H2 L'O, le citrate trisodique déshydraté, le dodécyl sulfate de sodium (SDS), le chlorhydrate de 1-éthyl-3-(3-diméthylaminopropyl) carbodiimide (EDC) et le N-hydroxysuccinimide (NHS) ont été obtenus auprès de Sigma-Aldrich. Le persulfate d'ammonium (APS), l'acide bromhydrique et le brome ont été fournis par Riedel-De Haën, Ajax Finechem et Panreac, respectivement. Toutes les solutions chimiques ont été préparées avec de l'eau déminéralisée. Les oligonucléotides synthétiques cibles et mésappariés de 30 bases ont été obtenus auprès de Bio Service Unit (NSTDA). L'ADN non complémentaire (ADNc) et la sonde signal provenaient de Sigma et la sonde de capture modifiée en 5'-amino provenait de Bioneer. La sonde de capture a été préparée dans 0,05 M de tampon phosphate de potassium (pH 7), tandis que les solutions d'ADN cible, de cible mésappariée, de sonde rapporteur et d'ADN non complémentaire ont été préparées dans du tampon phosphate de sodium (0,05 M, pH 7). Les séquences oligonucléotidiques utilisées dans la présente étude sont présentées dans le tableau 1.
Appareil
La mesure électroanalytique a été réalisée avec un potentiostat/galvanostat (Autolab PGSTAT12, Metrohm) équipé du logiciel GPES (4.0.007). Les expériences voltamétriques ont été réalisées avec un système conventionnel à trois électrodes composé d'une électrode de travail sérigraphiée en pâte de carbone (SPE) (Quasense, Bangkok, Thaïlande), d'une électrode de référence Ag/AgCl (3 M de KCl) et d'une tige de platine ( contre-électrode de 2 mm de diamètre). La technique de voltampérométrie différentielle (DPV) a été utilisée pour toutes les investigations électrochimiques à un potentiel de pas de 0,02 V et une vitesse de balayage de 0,5 V/s de 0,0 à +1,0 V dans un tampon de mesure de 4,5 ml (0,05 M de tampon phosphate de potassium) à pH 7 et température ambiante. Tous les potentiels mesurés dans cette étude ont été rapportés à l'électrode Ag/AgCl, et des solutions homogènes ont été préparées à l'aide d'un bain sonicateur (Elma S30H). Un microscope électronique à balayage (MEB, LEO 1450VP) a été utilisé pour déterminer la taille et la distribution des sphères de latex.
Méthodes
Synthèse de nanoparticules d'or colloïdal
Les AuNP colloïdales ont été préparées par réduction au citrate de sodium selon la méthode de Turkevich [31]. Brièvement, environ 10 mL de 5 mM de HAuCl4 ·3H2 O a été dissous dans 180 ml d'eau désionisée et bouilli dans des conditions d'agitation continue sur un dispositif combiné à plaque chauffante et agitateur magnétique. Dix millilitres de 0,5 % (w /v ) de citrate trisodique a ensuite été ajouté à la solution bouillante, et on a observé que la couleur de la solution passait progressivement du rouge pâle au rouge rubis.
Préparation des particules de latex
Les particules de latex ont été préparées par réaction de copolymérisation en émulsion sans savon comme décrit par Polpanich et al. [23] avec quelques modifications. En bref, environ 190 g d'eau désionisée ont été purgés avec de l'azote gazeux dans un ballon à trois cols immergé dans un bain d'eau pendant environ 1 h sous agitation à 350 tr/min. Vingt grammes de St et 0,5 g d'AA ont ensuite été ajoutés, et la température a été maintenue à 70°C. Après cela, environ 0,2 g d'APS a été ajouté dans 10 ml d'eau déminéralisée, puis versé dans la formulation dans le ballon à trois cols pour initier la réaction de polymérisation radicalaire, et le processus de polymérisation a été poursuivi pendant 8 h. Les sphères de latex carboxyle telles que synthétisées ont été récoltées par centrifugation avec de l'eau désionisée deux fois à 13 000 tr/min pendant 20 min [23, 27, 28] et remises en suspension dans de l'eau désionisée à température ambiante (25 °C) jusqu'à utilisation. La morphologie et la taille moyenne des particules de latex PSA ont été déterminées par microscopie électronique à balayage (MEB).
Modification de la surface SPE et de l'immobilisation de la sonde ADN
Avant la modification de la surface, le carbone SPE a été rincé abondamment avec de l'eau déminéralisée, puis enduit de gouttes avec la suspension de PSA à 3 mg/mL et laissé sécher à l'air dans des conditions ambiantes, suivi d'une coulée en gouttes avec 5 mg/mL d'AuNPs colloïdaux. . Les caractéristiques électrochimiques de la SPE de carbone avant et après modification avec des particules de latex et des AuNPs ont été examinées par la méthode CV. La SPE de carbone modifié au latex-AuNPs (PSA-AuNPs-SPE) a ensuite été rincée à l'eau déminéralisée et immergée dans 0,1 M de tampon phosphate de potassium (pH 5) contenant des réactifs de réticulation carbodiimide, soit 0,002 M d'EDC et 0,005 M de NHS pendant 2 h [32] avant de tremper une nuit dans 0,05 M de tampon phosphate de potassium (pH 7) contenant 5 M de sonde de capture. Après cela, l'ADN modifié PSA-AuNPs-SPE (DNA-PSA-AuNPs-SPE) a été soigneusement lavé avec du tampon phosphate de potassium (0,05 M, pH 7) pour éliminer la sonde physiquement adsorbée. L'électrode d'ADN a été immergée dans 0,05 M de tampon phosphate de sodium à pH 7 contenant de l'ADN cible linéaire (1 M) et un marqueur d'hybridation d'ADN AQMS (5 mM) pour une hybridation partielle pendant 1 h, puis plongée dans 0,05 M de tampon phosphate de sodium (pH 7) conditionné avec 1 uM de sonde rapporteur et 5 mM d'AQMS pendant encore 1 h pour le processus d'hybridation d'ADN complet. Enfin, l'électrode d'ADN a été rincée avec du tampon phosphate de potassium (0,05 M, pH 7) pour l'élimination des séquences oligonucléotidiques non hybridées. La réponse électrochimique de chaque substance étendue fixée sur la SPE a été étudiée avec la méthode DPV. La figure 1 représente la procédure par étapes pour le développement de V. cholerae Biocapteur d'ADN basé sur un support solide colloïdal PSA-AuNPs.
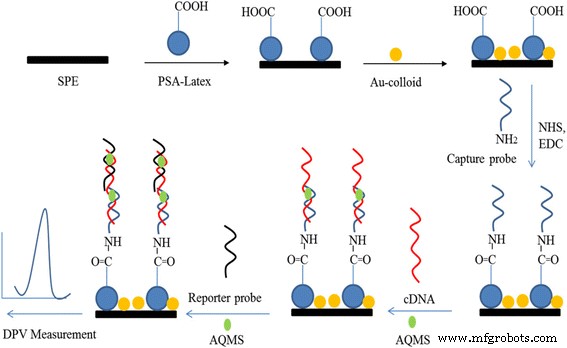
Représentation schématique de la procédure de fabrication par étapes du biocapteur d'ADN
Optimisation de l'hybridation d'oligonucléotides synthétiques
L'effet de divers paramètres tels que la sonde ADN et les concentrations AQMS, le pH, la force ionique et la capacité tampon sur la réponse d'hybridation de la sonde ADN immobilisée avec la sonde signal et l'ADN cible a été examiné avant la détermination de la plage linéaire dynamique de l'ADN. biocapteur. De plus, les durées d'immobilisation de la sonde de capture et d'hybridation de l'ADN ainsi que la durée de vie et la régénération du biocapteur ont également été évaluées avant le développement du V. cholerae Le biocapteur d'ADN était prêt pour l'application dans l'expérience de pointe et de récupération. Les charges de la sonde de capture et de l'AQMS ont été optimisées en faisant varier leurs concentrations de 1 à 6 M et 0,1 à 5 mM, respectivement, tandis que la concentration de l'ADN cible et de la sonde rapporteur a été maintenue à 5 M dans 0,05 M de tampon phosphate de sodium (pH 7,0). Des études sur l'effet du pH et la concentration du tampon ont été menées en changeant le pH et la concentration du tampon phosphate de sodium de pH 5,5 à 8,0 et 0,001 à 1 000 M, respectivement. La présence de différents cations dans la réponse d'hybridation d'ADN du biocapteur électrochimique d'ADN a été réalisée en ajoutant Na + , K + , Ca 2+ , et Fe 3+ ions à 1,0 M dans le tampon d'hybridation d'ADN contenant 1 mM d'AQMS et 5 µM d'ADNc et sonde de détection de pH 7,0. L'effet de la force ionique a été examiné en faisant varier la concentration de NaCl sur la plage de 0,1 à 3,0 M à pH 7,0. La plage dynamique du biocapteur d'ADN a ensuite été déterminée en 1,0 × 10 −21 à 1,0 × 10 −8 M V. cholerae ADNc en utilisant une concentration de sonde de signal constante à 5 M et pH 7,0. La durée d'immobilisation de la sonde ADN a été déterminée en trempant l'électrode ADN dans 5 M de solution de sonde de capture (pH 7,0) entre 1 et 13 h, et la réponse DPV a été prise toutes les 1 à 2 h. Pendant ce temps, le temps d'hybridation de l'ADN a été déterminé en permettant à la réaction d'hybridation de l'ADN de se produire entre 15 et 180 min, et la réponse du biocapteur d'ADN a été enregistrée toutes les 15 à 30 min. La durée de conservation du biocapteur d'ADN a été déterminée en mesurant périodiquement la réponse du biocapteur d'ADN vers la détection de 5 M V. cholerae ADNc pendant 120 jours. L'analyse a été réalisée en cinq répétitions en utilisant une nouvelle électrode d'ADN pour chaque essai d'hybridation en sandwich. La régénération de l'électrode d'ADN a été effectuée en utilisant 0,1 M de solution de NaOH pendant 4 min, et la réhybridation du biocapteur d'ADN (60 min) a été réalisée en utilisant une solution de réhybridation contenant 5 M d'ADNc et une sonde de détection et 1 mM d'AQMS à une force ionique de 2,0 M dans 0,05 M de tampon phosphate de potassium (pH 7,0). L'expérience de régénération a été menée en six répétitions.
V. cholerae Quantification à l'aide d'un biocapteur d'ADN électrochimique basé sur PSA-AuNPs
Divers V. cholerae souches bactériennes à savoir J2126-I, J2126-II, J3324-I, J3324-II, J3330-I, J3330-II, CDHI 5294-II et UVC1324, y compris Citrobacter freundii (CF-I) et Citrobacter freundii (CF-II) ont été obtenus auprès du Laboratoire de microbiologie, Faculté des sciences appliquées, Université AIMST, Kedah. L'extraction d'ADN génomique a ensuite été réalisée sur ces bactéries à l'aide de QIAGEN Genomic-tip 500/G selon le protocole du fabricant. L'ADN extrait a ensuite été dilué 100 fois en utilisant du tampon phosphate de sodium (0,05 M, pH 7,0). Environ 300 ml de l'ADN extrait contenant 2,0 M de NaCl et 1 mM d'AQMS ont été soniqués pendant 15 min pour libérer les cassures d'ADN. Ensuite, la sonde d'ADN immobilisée a été trempée pendant 1 h pour permettre au processus d'hybridation d'ADN de se dérouler et lavée soigneusement avec du tampon phosphate de potassium 0,05 M (pH 7,0) pour éliminer l'ADN non lié. L'évaluation de la réponse du biocapteur d'ADN basée sur le courant de crête DPV a été mesurée et comparée à la réponse de courant générée par l'électrode d'ADN sans réaction avec l'ADNc comme signal de contrôle. Chaque expérience a été réalisée en triple dans les mêmes conditions expérimentales. t commun test a été utilisé pour déterminer la différence significative entre la réponse du biocapteur d'ADN et la réponse du contrôle. La récupération de V. cholerae J3324 et V. cholerae ADN UVC1324 à 1,0 × 10 −4 g μL −1 , 1,0 × 10 −5 g μL −1 , et 1,0 × 10 −6 g μL −1 dopés dans le tampon d'hybridation ont ensuite été effectués à l'aide du biocapteur d'ADN électrochimique à base de PSA-AuNPs proposé.
Résultats et discussion
Morphologie des particules de latex et comportement électrochimique de la SPE modifiée par nanoparticules de latex et d'or
La figure 2 montre la micrographie SEM des sphères de latex carboxylé avec une taille de particule moyenne de 186,1 Â ± 4,6 nm. La distribution de taille uniforme des sphères de PSA a permis une immobilisation homogène des molécules d'ADN sur la surface du latex pour améliorer la réponse de reproductibilité du biocapteur d'ADN. Un microscope électronique à balayage (MEB, LEO 1450VP) a été utilisé pour déterminer la taille et la distribution des sphères de latex.

Micrographie SEM des sphères de latex PSA telles que synthétisées à 10 000 grossissements
Les résultats électrodynamiques de la SPE modifiée sont présentés dans le tableau 2. Le pic de séparation de potentiel (ΔEp) indique la cinétique de transfert d'électrons du système. La SPE modifiée par PSA (PSA-SPE) a montré la valeur ΔEp la plus élevée en raison du processus de transfert de charge lent dans la couche de particules de copolymère colloïdal, ce qui a fait passer le système à un état quasi-réversible. Cependant, lorsque les AuNPs ont été chargés sur le PSA-SPE, la diminution du ΔEp implique une amélioration du taux de transfert d'électrons à la surface de l'électrode.
La figure 3 montre les voltamogrammes d'impulsion différentiels de la réponse d'oxydation AQMS sur le SPE modifié au latex et la réponse d'hybridation séquentielle du V. cholerae Biocapteur d'ADN. Une différence significative de courant de crête DPV a été observée entre les électrodes contenant uniquement des microsphères modifiées au latex et sans ADN de capture immobilisé (expériences (a) et (b)) et modifiées avec des sondes d'ADN de capture immobilisées en présence d'ADNc et une sonde rapportée (expérience (g )). Cela indique que les sondes de capture d'ADN aminé ont été immobilisées avec succès sur les sphères de latex PSA carboxylées enduites via le protocole de couplage EDC/NHS [33]. L'expérience (g) montre également une réponse DPV beaucoup plus élevée par rapport aux électrodes d'ADN en présence d'ADNc et de la sonde rapporteur rapportée (expérience (d)), dans l'ADN non apparié et la sonde rapporteur rapportée (expérience (e)), et l'ADNc cible avec aucune sonde rapportée (expérience (f)). Cela est dû à l'hybridation complète de l'ADN cible avec des sondes de capture et de reportage par réaction d'hybridation en sandwich sur la surface du biocapteur d'ADN comme démontré dans l'expérience (g). Cela montre également que l'utilisation de la sonde rapportée pourrait améliorer le signal de l'hybridation d'ADN. Néanmoins, le courant DPV résultant de l'hybridation observée en présence d'ADN cible sans incorporation d'une sonde rapportée (expérience (f)) est toujours plus élevé que les signaux de courant DPV observés pour l'ADN non hybridé (expériences (c), (d) et (e)).
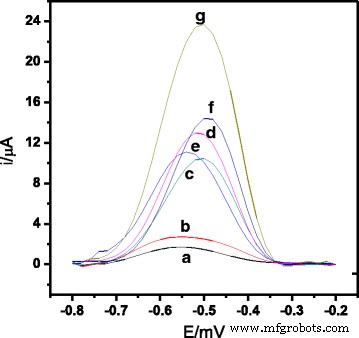
Signal de voltamogrammes à impulsions différentielles provenant de l'AQMS des électrodes (a ) PSA-SPE, (b ) PSA-AuNPs-SPE, (c ) capture sonde-PSA-AuNPs-SPE et en présence de (d ) ADNc et sonde rapporteur, (e ) ADN non concordant et sonde rapporteur, (f ) l'ADNc seul, et (g ) ADNc et sonde rapporteur
Effet du chargement de la sonde ADN et de la concentration AQMS
L'effet de la concentration de sonde d'ADN sur la réponse d'hybridation d'ADN a été observé à travers la réponse d'oxydation électrochimique AQMS. La figure 4a montre que la réponse du biocapteur d'ADN a augmenté progressivement avec l'augmentation de la quantité de sonde d'ADN immobilisée sur le PSA-AuNPs-SPE de 1 à 4 uM. Cela a été attribué à la quantité croissante d'AQMS électroactifs intercalés dans l'ADN double brin (dsDNA) pour effectuer un transfert d'électrons à travers l'hélice d'ADN immobilisée. La réponse DPV du biocapteur d'ADN a été observée pour devenir presque plateau entre 4 et 6 M de sonde d'ADN, ce qui indique qu'un chargement optimal de la sonde d'ADN sur la surface de l'électrode a été atteint [34]. Par conséquent, une sonde de capture de 4 uM a été sélectionnée comme charge optimale de sonde d'ADN dans les expériences suivantes. La concentration du marqueur AQMS a également été optimisée dans l'électrolyte de mesure entre 0,1 et 5,0 mM, et la concentration d'AQMS à 1 mM s'est avérée suffisante pour une réaction optimale d'intercalation de l'ADN (Fig. 4b).
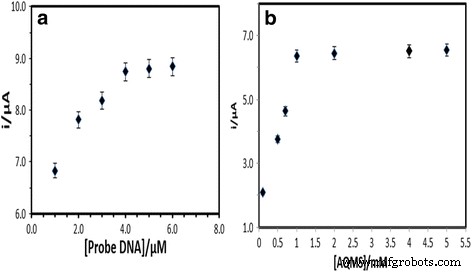
L'effet de la sonde de capture (a ) et les concentrations AQMS (b ) sur la réponse du biocapteur d'ADN réalisée avec 5 M d'ADNc et une sonde signal dans du tampon phosphate de sodium 0,05 M (pH 7,0)
Effet du pH, de la force ionique et de la capacité du tampon
La vitesse de réaction d'hybridation de l'ADN dépend beaucoup du pH de la solution. Comme on peut le voir sur la figure 5a, dans un environnement plus acide, la protonation du squelette phosphodieter de l'ADN a réduit la solubilité de la molécule d'ADN, ce qui a finalement diminué la vitesse de réaction d'hybridation de l'ADN. Alors qu'en milieu basique, il brisait les faibles liaisons hydrogène maintenant les paires de bases d'ADN ensemble. La réaction d'hybridation d'ADN optimale était plus favorable dans des conditions neutres, ce qui a permis à davantage de sondes de capture de s'hybrider avec l'ADN cible et a ensuite permis l'intercalation de sondes redox AQMS pour faire de la reconnaissance de l'hybridation d'ADN une activité commerciale. Ainsi, 0,05 M de tampon phosphate de sodium à pH 7,0 a été utilisé comme milieu d'hybridation d'ADN pour les études de biocapteur d'ADN ultérieures. Ions chargés positivement tels que Ca 2+ , Na + , K + , et Fe 3+ Les ions peuvent interagir avec la chaîne phosphodiester chargée négativement de l'ADN. Cette réaction ionique neutralisera la charge de la molécule d'ADN, diminuant ainsi les répulsions stériques entre les molécules d'ADN pour faciliter la réaction d'hybridation de l'ADN. La figure 5b illustre l'effet de certains cations sur la réponse d'hybridation d'ADN. On a remarqué que la réponse d'hybridation d'ADN augmentait en présence d'ions chargés positivement de l'ordre de Na + > K + > Fe 3+ > Ca 2+ . Les deux Ca 2+ et Fe 3+ ions ont été trouvés pour réduire considérablement la réponse d'hybridation de l'ADN en raison des interactions ioniques de Ca 2+ et Fe 3+ avec les ions phosphate de la solution tampon, ce qui a conduit à la formation de composés de phosphate insolubles. Cela a réduit la teneur ionique du milieu, augmentant ainsi la répulsion électrostatique entre les molécules d'ADN. Le courant d'hybridation d'ADN le plus élevé a été obtenu en présence de Na + ion en raison de sa plus petite taille et de sa plus grande affinité envers le squelette ADN sucre-phosphate par rapport à K + ion pour surmonter l'encombrement stérique et la répulsion électrostatique entre les groupes phosphate chargés négativement des ADN.
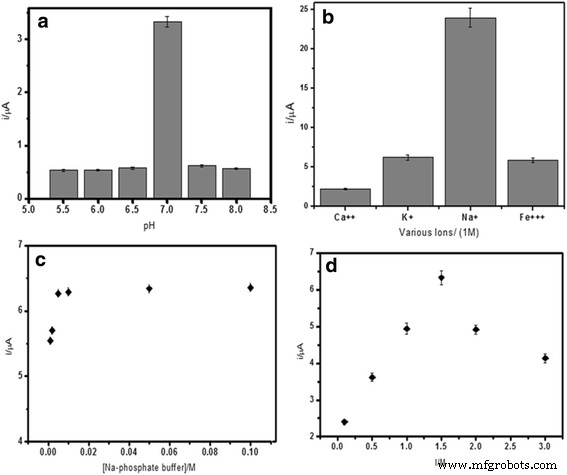
L'effet du pH (a ) divers cations (b ), concentration du tampon (c ) et la force ionique (d ) sur la réponse d'hybridation d'ADN du V. cholerae Biocapteur d'ADN. L'hybridation a été réalisée avec 5 M d'ADNc et une sonde rapporteur suivie d'une intercalation avec 1 mM d'AQMS
De plus, la force ionique de la solution affecterait également la réponse du biocapteur d'ADN. La figure 5c, d montre la capacité tampon optimale et la force ionique a été obtenue en utilisant 0,05 M de tampon phosphate de sodium avec le pH fixé à pH 7,0 et 2,0 M de NaCl, respectivement. Dans cette condition, il a favorisé au maximum la réaction d'hybridation de l'ADN; par conséquent, une réponse DPV élevée a été obtenue. À une capacité tampon et à une force ionique optimales de la solution, la répulsion électrostatique entre les molécules d'ADN a diminué, améliorant ainsi la réaction d'hybridation de l'ADN. En revanche, lorsqu'une teneur ionique trop faible ou trop élevée était utilisée, l'encombrement stérique et la répulsion électrostatique devenaient dominants et limitaient l'hybridation des molécules d'ADN.
Mise en place de V. cholerae Courbe d'étalonnage du biocapteur d'ADN
D'après le résultat illustré à la figure 6a, la réponse du biocapteur d'ADN a augmenté proportionnellement à l'augmentation de la concentration d'ADNc de 1,0 × 10 −21 à 1,0 × 10 −8 M (R 2 =0,99) avec une limite de détection de 1,0 × 10 −21 M. La limite de détection a été calculée sur la base de trois fois l'écart type de la réponse du biocapteur à la courbe de réponse approchant la limite de détection divisée par la pente d'étalonnage linéaire. La large plage de détection linéaire du biocapteur d'ADN était due aux particules de latex de PSA hautement monodispersées et sphériques de taille submicronique utilisées comme matrice de support pour l'immobilisation de l'ADN. La couche riche en acide acrylique sur la surface des particules de latex offrait un grand site de liaison pour la fixation des sondes de capture d'ADN afin de créer une surface maximale couverte par la couche réceptrice d'ADN. De plus, l'incorporation d'AuNPs sur la SPE modifiée par PSA a encore amplifié le signal analytique de la réponse d'hybridation d'ADN, ce qui a rendu la sensibilité élevée du biocapteur d'ADN (Fig. 6b).
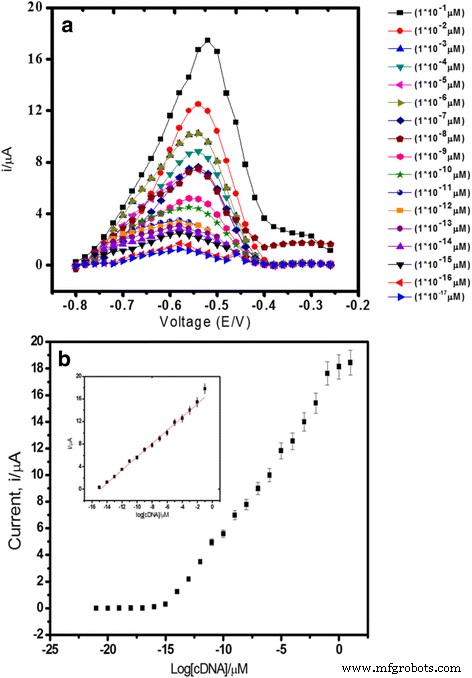
Differential pulse voltammograms (a ) and DNA biosensor linear range (b ) obtained using various cDNA concentrations from 1.0 × 10 −15 to 1.0 × 10 −1 μM V. cholerae target DNA and 5 μM signal probe
DNA Probe Immobilisation and Hybridization Times
It took about 8 h for the capture probe to be immobilised on the PSA copolymer particles surface, as illustrated by the DNA biosensor response in Fig. 7a, which showed a current increment from 1.0 to 8.0 h of capture probe immobilisation time, after which no obvious change in the DPV current was observed. Longer immobilisation time resulted in a higher amount of DNA probes immobilised onto the latex. After 8.0 h of exposure to the DNA probes, the hydrophilic functional latex with reactive carboxyl groups at the surface was presumably fully attached with the DNA probes. DNA hybridisation time, on the other hand, is the rate limiting step, which determines the response time of the DNA biosensor. Based on the DNA biosensor response trend in Fig. 7b, the response time of the V. cholerae DNA biosensor developed in this study was estimated to be about 60 min for the dual hybridisation processes to complete.
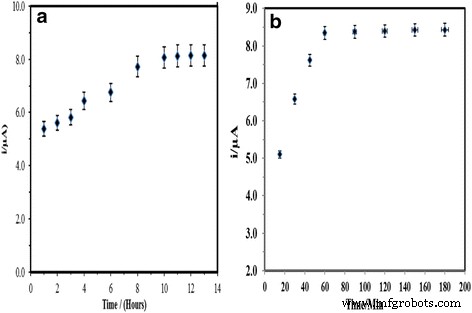
DNA probe immobilisation duration on the immobilised PSA latex colloidal particles (a ) and DNA hybridization duration of the DNA biosensor (b ) in 0.05 M potassium phosphate buffer at pH 7.0 containing 5 μM target DNA and reporter probe and 1 mM AQMS at 2.0 M ionic strength
Long-Term Stability and Regeneration of V.cholerae DNA Biosensor
Figure 8 shows the shelf life of the V. cholerae DNA biosensor. The DNA biosensor showed the highest response to the detection of 5 μM of V. cholerae cDNA for the first month of the experimental period. The electrochemical DNA biosensor was able to retain 95% of its initial DPV current after 58 days of storage period. The DNA hybridisation response was then gradually decreased to about 75% of its original response on the 75th day and exhibited 40% of its initial performance on the 100th operational day. The bioactivity of the immobilised capture probe was finally declined to 30% after 3 months of storage period. The reproducibility of each calibration point, which was repeated on five replicate DNA electrodes, gave satisfactory relative standard deviation (RSD) between 2.4 and 4.5% (n =5).
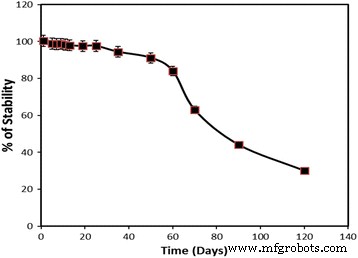
The life span profile of the fabricated V. cholerae DNA sensing electrode. The electrode was stored in 0.05 M potassium phosphate buffer (pH 7.0) at 4 °C after every DPV measurement was taken
Regeneration of biosensor indicates whether the biosensor is reusable for a series of consecutive analyses. The regeneration method used in this study was conducted based on previously reported protocol in other studies [11, 21] with slight modifications. In this study, 0.1 M of NaOH solution was used as the regeneration solution to break the hydrogen bonds between base pairs of hybridised dsDNA. With the result from Fig. 9, it is notable that the DNA biosensor response declined significantly after incubation in 0.1 M of NaOH and the percentage of the DNA biosensor response reduced from 35.1 to 5.2% relative to the DNA biosensor initial response after incubation in 0.1 M of NaOH solution from 30 to 240 s. The DNA biosensor response decreased with the increasing incubation time signifies the hydrogen bonds between hybridised dsDNA were broken up by the alkaline regeneration solution. However, rehybridisation of the DNA biosensor was able to attain almost 100% of its initial response for a consecutive six DNA analyses with a reversibility RSD of 5%.
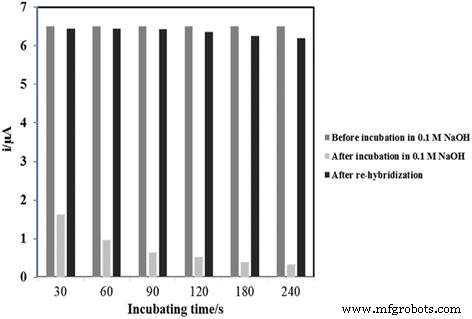
Repeatability of V. cholerae DNA biosensor using 0.1 M NaOH regeneration solution and rehybridization solution containing 5 μM cDNA and detection probe and 1 mM AQMS at 2.0 M ionic strength in 0.05 M potassium phosphate buffer (pH 7.0)
Determination of V. cholerae Bacteria with the Developed DNA Biosensor
The optimised DNA biosensor has been applied to quantify the V. cholerae DNA extracted from various V. cholerae bacterial strains. Table 3 presents the results acquired from DNA tests carried out with the hybridisation medium spiked with different strains of V. cholerae DNAs and other bacterial species at a concentration within the calibration range of the DNA biosensor. The DNA biosensor showed superior selectivity towards V. cholerae J3324–I, V. cholerae J3324–II, and V. cholerae UVC1324 with high DPV current response and low current signals were obtained for the evaluation of both Citrobacter freundii (CF-I) and Citrobacter freundii (CF-II).
Recovery of V. cholerae J3324 and V. cholerae UVC1324 DNAs at three different concentrations spiked into the hybridisation buffer demonstrated 91.4 ± 2.2% to 108.9 ± 4.8% (n =3) of recoveries percentage (Table 4). This result suggests that the proposed PSA-AuNPs-based electrochemical DNA biosensor could be adopted for highly reliable and accurate detection of V. cholerae DNA in environmental and clinical samples.
Performance Comparison with Other Reported V. cholerae DNA Biosensors
Based on the data summarised in Table 5, the proposed electrochemical DNA biosensor based on PSA-AuNPs immobilisation material shows an exceptional broad linear quantification range compared to other planar two-dimensional electrodes as the DNA supporters. This clearly demonstrates the advantage of the micro-sized latex particles where the polymeric PSA is capable to intensify the probe binding capacity with a simple loading method via the classical EDC/NHS coupling compared to avidin-biotin technology [24, 26] and ultra-low detection limit in zeptomolar range with reasonable assay time.
Conclusions
This study reports the development of an electrochemical DNA biosensor for the detection of one of the most devastating high-risk V. cholerae pathogens. The PSA-AuNPs-modified DNA biosensor can be used for direct detection of DNA of interest from the extracted DNA without the need of amplification reaction via conventional PCR method, which is commonly used in those previously reported V. cholerae DNA biosensors. In addition, no further dilution of the extracted DNA is needed as the high-capacity AuNPs-doped latex microspheres-based DNA biosensor is highly sensitive for the quantitation of DNA at extremely low level in sub zeptomolar range. Therefore, the electrochemical DNA biosensor is greatly suitable as a surveillance and diagnostic tool to control the epidemic of the fatal intestinal infection.
Nanomatériaux
- Le potentiel d'intégration de données visuelles avec l'IoT
- Démonstration d'un biocapteur flexible à base de graphène pour la détection sensible et rapide des cellules cancéreuses de l'ovaire
- Détection photo-électrochimique améliorée de l'acide urique sur une électrode de carbone vitreuse modifiée par nanoparticules d'Au
- Un biocapteur d'ADN électrochimique hautement sensible à partir d'un nanocomposite acrylique-or pour la détermination du sexe du poisson Arowana
- Nanosphères de carbone monodispersées à structure poreuse hiérarchique comme matériau d'électrode pour supercondensateur
- Stabilité améliorée des nanoparticules magnétiques d'or avec du poly(acide 4-styrènesulfonique-co-acide maléique) :propriétés optiques adaptées pour la détection des protéines
- Synthèse contrôlée de BaYF5:Er3+, Yb3+ avec une morphologie différente pour l'amélioration de la luminescence de conversion ascendante
- Un aptacapteur fluorescent à base d'oxyde de graphène pour la détection d'activation de CCRF-CEM
- Impossible Objects s'associe à BASF pour l'impression 3D composite



