Identification des macromolécules caractéristiques des génotypes d'Escherichia coli par cartographie mécanique à l'échelle nanoscopique au microscope à force atomique
Résumé
La catégorisation des souches microbiennes est classiquement basée sur la méthode moléculaire, et rarement les caractéristiques morphologiques des souches bactériennes étudiées. Dans cette recherche, nous avons révélé les structures macromoléculaires de la surface bactérienne via une cartographie mécanique AFM, dont la résolution n'était pas seulement déterminée par la taille de la pointe nanométrique, mais également par les propriétés mécaniques de l'échantillon. Cette technique a permis l'étude à l'échelle nanométrique des structures membranaires des souches microbiennes avec une préparation simple des échantillons et des environnements de travail flexibles, qui ont surmonté les multiples restrictions de la microscopie électronique et des méthodes d'analyse biochimiques. Les macromolécules caractéristiques situées à la surface des cellules ont été considérées comme des protéines de la couche de surface et se sont avérées spécifiques à Escherichia coli génotypes, à partir desquels les tailles moléculaires moyennes ont été caractérisées avec des diamètres allant de 38 à 66 nm, et les formes moléculaires étaient en forme de rein ou rondes. En conclusion, les structures macromoléculaires de surface ont des caractéristiques uniques qui se lient à E. coli génotype, ce qui suggère que les effets génomiques sur les morphologies cellulaires peuvent être rapidement identifiés en utilisant la cartographie mécanique AFM.
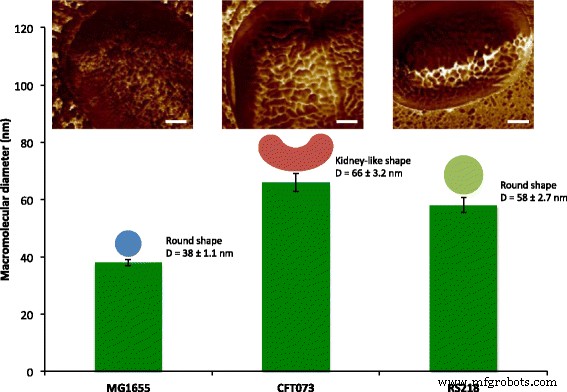
Quantification des macromolécules de surface de E. coli cellules en utilisant la cartographie mécanique AFM. Macromolécules de surface de la surface cellulaire de trois E. coli les génotypes, MG1655, CFT073 et RS218, ont été caractérisés avec des tailles allant de 38 à 66 nm et avec des formes rondes ou en forme de rein. Les images topographiques ont été colorées avec une cartographie d'adhérence avec les barres d'échelle = 200 nm.
Contexte
En raison de la résolution inférieure de la microscopie optique et de l'environnement de travail restreint de la microscopie électronique, les chercheurs dans le domaine microbien envisagent rarement d'utiliser l'apparence de cellules bactériennes mais adoptent plutôt des méthodes d'analyse moléculaire ou chimique pour l'identification de fragments génomiques, l'expression de protéines, etc. Il n'est pas surprenant que ces méthodes présentent un certain nombre d'inconvénients, notamment le fait qu'elles demandent beaucoup de travail et de temps, et par conséquent, des approches plus simples, efficaces et flexibles sont nécessaires. Inventé en 1982 par Binnig et al., le microscope à force atomique (AFM) est conçu pour utiliser une sonde nanométrique contrôlée par un faisceau laser pour l'observation de la surface d'un échantillon avec une résolution nanoscopique voire atomique [1]. Imagerie de la morphologie via une pointe de sonde physique, cette technique surmonte la limite de résolution et les restrictions environnementales de la microscopie optique et électronique et présente un certain nombre d'avantages, tels qu'une préparation simple des échantillons et des environnements de travail flexibles dans l'air ambiant ou dans des conditions de fluides [2, 3]. Néanmoins, en ce qui concerne les études microbiennes, les applications actuelles de l'AFM concernent principalement l'imagerie qualitative des morphologies statiques ou dynamiques des cellules bactériennes ou l'expression des flagelles et des pili [4,5,6], alors que peu d'études se sont focalisées sur l'ultrastructure de surface des les cellules microbiennes et l'analyse quantitative des propriétés cellulaires.
Dans cette recherche, l'AFM a été sélectionné pour l'étude de surface de Escherichia coli (E. coli ) et les formes et dimensions de la bactérie individuelle ont été observées par la topographie AFM et les images de phase. De plus, une cartographie mécanique simultanée s'est avérée révéler des informations biomécaniques supplémentaires sur les composants de surface, où de petites différences dans les caractéristiques adhésives entre les macromolécules et la matrice environnante pouvaient être détectées lors de chaque contact physique entre la pointe et l'échantillon. En appliquant ces techniques avancées aux champs microbiens, nous avons examiné trois E. coli génotypes contenant une souche de laboratoire et deux souches pathogènes humaines pour l'identification des macromolécules de surface. Les résultats ont montré que la technique pouvait offrir une résolution d'image dépassant l'échelle de la pointe de l'AFM en détectant la distribution mécanique de l'échantillon. En conclusion, nous suggérons qu'un tel développement de la science des surfaces fournirait non seulement aux chercheurs travaillant dans les domaines microbiens des détails sur l'apparence cellulaire, mais contribuerait également à notre connaissance des caractéristiques de surface d'un autre système de bio- ou nanomatériaux.
Méthodes
Échantillons microbiens
Les trois E. coli les souches testées dans cette recherche ont été isolées cliniquement et fournies par le laboratoire du professeur Ching-Hao Teng à l'Institut de médecine moléculaire de l'Université nationale Cheng Kung. MG1655 est la souche intestinale et la souche de laboratoire de type sauvage de E. coli K-12, et les deux autres souches sont des agents pathogènes humains - CFT073, la principale cause d'infections des voies urinaires, et RS218, associée à la méningite néonatale chez les nourrissons [7].
Substrat fonctionnel
Deux étapes de modifications de surface ont été appliquées pour la liaison covalente entre la surface solide et les cellules microbiennes. Tout d'abord, une solution de 3-aminopropyltriéthoxysilane (APTES, Sigma-Aldrich Co. LLC, USA) a été utilisée pour former un NH2 initial -couche fonctionnalisée sur la surface, où le revêtement APTES stable a contribué aux liaisons Si-O stables - Si fourni par APTES et O de la surface oxydée. Possédant deux fonctions COOH, le glutaraldéhyde se combine au NH2 fonction d'APTES avec un COOH et fonctionne avec le NH2 sur la surface bactérienne avec l'autre COOH.
Les substrats propres ont été immergés dans une solution APTES, avec un mélange de 5% APTES dans de l'éthanol, pendant une heure et rincés par de l'éthanol et du ddH2 O. Les lames ont été séchées à l'aide d'un courant d'azote, puis placées dans une solution de glutaraldéhyde à 2 % dans du PBS pendant une nuit et lavées par du PBS.
Préparation de l'échantillon
Des colonies isolées de E. coli les souches ont été sélectionnées à partir des plaques de gélose au bouillon de lysogénie (LB) et incubées dans du bouillon LB. Après les temps de culture de 12 h, la solution bactérienne a ensuite été diluée au 1:100 dans du bouillon LB frais pré-équilibré. Après encore 12 h de culture microbienne, la solution bactérienne a été soumise à une centrifugation à 1500×g (4000 tr/min) pendant 3 min et remis en suspension dans du bouillon LB, ce processus étant répété deux fois. Deux cents microlitres de la solution bactérienne ont été déposés sur le substrat fonctionnalisé et laissés au repos pendant 30 min. L'échantillon a ensuite été immergé dans de l'eau distillée deux fois pour éliminer les cellules non attachées et immédiatement imagé sous AFM dans l'air ambiant.
Caractérisation AFM
Un instrument AFM (Bruker Nano, Santa Barbara, Californie, États-Unis) et une sonde en nitrure de silicium avec une constante de ressort calibrée de 0,7 N/m et un rayon de pointe de 10 nm ont été sélectionnés pour l'examen de surface des échantillons microbiens. Le taux de balayage AFM et les pixels de ligne étaient respectivement de 0,5 Hz et 256 lignes pour la taille de balayage de 10 μm pour la première détection de la topographie, et les paramètres ont ensuite été définis sur 0,3 Hz et 512 lignes pour la taille de balayage de 2 μm pour l'observation détaillée. Le mode nanomécanique quantitatif PeakForce (QNM) a été utilisé pour la cartographie nanomécanique, où les propriétés adhésives de la surface ont été calculées à partir de la force d'attraction maximale parmi les courbes force de retrait-distance.
Notre précédente étude sur Streptococcus mutans ont montré l'évolution mécanique sur la surface bactérienne pendant 2 h, surveillée par cartographie mécanique AFM continue, et il a été vérifié que les échantillons microbiens restaient en vie pendant cette durée [8,9,10]. Pour assurer la viabilité du E. coli échantillons utilisés dans ce travail, les pré-études sur les échantillons bactériens ont été menées, et le changement continu de l'adhérence de surface pendant 4 h impliquait que les cellules restent en vie pendant au moins 4 h après la préparation de l'échantillon (indiqué dans le fichier supplémentaire 1). Par conséquent, les échantillons microbiens testés dans ce travail ont été mesurés par AFM dans les 2 h suivant la préparation des échantillons. Tous les E. coli les génotypes ont été cultivés individuellement et à des moments différents, et les mesures de l'AFM ont été effectuées immédiatement après la préparation des échantillons. En d'autres termes, les échantillons bactériens n'étaient pas alignés en attendant l'examen, de sorte que les effets du temps de rétention sur les différences entre E. coli les contraintes ont été minimisées. Les données quantitatives pour chaque E. coli souches ont été recueillies à partir des mesures prises dans les 2 h de cette étude.
Analyse statistique
Prism (GraphPad Software, USA) a été utilisé pour l'analyse statistique dans ce travail. Les longueurs cellulaires et les tailles macromoléculaires ont été présentées comme des valeurs moyennes avec l'erreur standard de la moyenne (SEM). Les comparaisons multiples entre E. coli les génotypes ont été traités en utilisant une analyse de variance unidirectionnelle ordinaire (ANOVA). Le niveau de confiance de 95 % (p < 0,05) a été sélectionné et les astérisques indiquent le degré de différence significative trouvé. Le numéro d'échantillon n de chaque souche était> 40.
Résultats et discussion
Ultrastructure de surface par cartographie multiple
Lors de la numérisation avec AFM à une échelle d'observation de 10 μm sur un échantillon bactérien, plusieurs E. coli Des cellules MG1655 pouvaient être vues, et la forme cellulaire en trois dimensions pouvait être observée à partir de l'image topographique (Fig. 1a) ; les contours clairs des cellules montrés en deux dimensions ont été obtenus par l'image d'erreur de déviation (Fig. 1b), et plusieurs tubules en plus des cellules bactériennes ont pu être trouvés. Les filaments en forme de vague (Fig. 1c) étaient cohérents avec l'apparition de flagelles microbiens rapportés dans un autre travail, qui a confirmé une telle découverte comme flagelles, et les pili plus courts et ressemblant à des cheveux pouvaient également être vus [11]. Lors de la diminution de la zone d'observation de la sonde pour l'étude détaillée de cellules microbiennes individuelles, la topographie a montré une légère différence dans la direction verticale entre les surfaces cellulaires, comme le montre la figure 1d, et l'image d'erreur de déviation semblait fournir plus d'informations morphologiques tout en le bruit environnemental collecté était trop important pour étudier l'ultrastructure de la surface cellulaire (Fig. 1e). Lorsque les propriétés biomécaniques simultanées de l'échantillon ont été mesurées pendant le contact entre la pointe et l'objet testé, il a été constaté que la surface bactérienne était en fait composée d'une énorme quantité de macromolécules avec une forme et une taille spécifiques, comme l'a révélé la cartographie de la force d'adhérence ( Fig. 1f) ; ainsi, la résolution morphologique des images topographiques et d'erreur de déviation a été encore améliorée grâce aux informations biomécaniques.
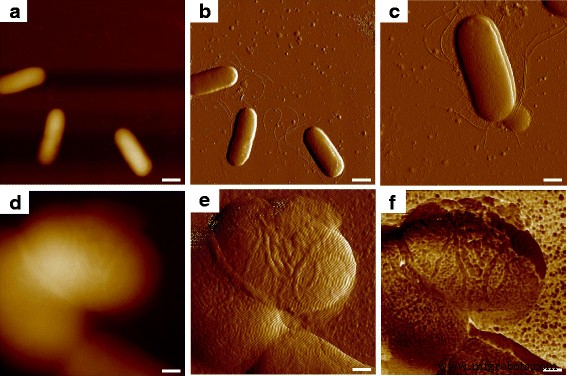
Ultrastructure de surface de E. coli MG1655 utilisant le mappage multiple AFM. un et b étaient les images d'erreur topographique et de déviation, et c était l'image détaillée de l'erreur de déviation de la cellule bactérienne avec l'expression des flagelles et des pili. Une seule cellule a ensuite été focalisée, où d et e étaient les images d'erreur topographique et de déviation, et f était la cartographie d'adhérence correspondante. Les barres d'échelle = 1 μm dans a et b , 500 nm en c , et 200 nm en d –f
Sur la figure 2a, l'expression des flagelles par E. coli MG1655 pouvait évidemment être vu, où la taille des filaments était similaire à celle de la figure 1b, avec des propriétés d'adhérence relativement inférieures à celles du substrat. De plus, la surface bactérienne s'est avérée être composée de composants circulaires caractérisés comme étant moins adhésifs que la matrice environnante. Cette observation est similaire à nos découvertes précédentes sur les couches tissulaires de la peau de souris, et les grains récurrents et comparables ont été considérés comme des macromolécules, dont la structure est plus dense et cohérente que celles observées dans la région intermoléculaire, de sorte que les différences de performances d'adhésion pourraient être facilement détecté [12]. La couche la plus externe de l'enveloppe cellulaire chez les bactéries Gram-négatives est une couche de protéines d'auto-assemblage, comme illustré sur la figure 2b, qui est connue sous le nom de protéine de couche de surface (couche S) [13]. Les structures de la couche S étaient traditionnellement mesurées par microscopie électronique, tandis que les exigences des environnements sous vide et du revêtement conducteur perdaient les informations natives et en temps réel sur les protéines. Bien que certaines recherches aient extrait les protéines de la couche S et les aient réassemblées sur un substrat de mica pour le balayage AFM, les résultats manquaient pour les performances in situ et en temps réel de la structure de la couche S [14, 15]. Sur la base de l'architecture cellulaire et de certaines images précédentes de la surface microbienne par microscopie électronique, nous avons considéré les macromolécules observées comme des protéines de la couche S [16].
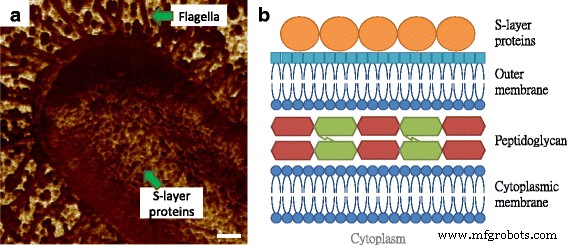
Illustration des structures verticales et de surface de E. coli cellules. un Les macromolécules de surface sur E. coli Cellule MG1655 imagée par cartographie d'adhérence AFM. b L'architecture moléculaire de l'enveloppe cellulaire chez les microbes à Gram négatif, qui se compose d'une membrane cytoplasmique, d'un peptidoglycane, d'une membrane externe et d'une couche S. La barre d'échelle = 200 nm
En comparant les mesures de l'AFM et de la microscopie électronique à transmission (MET), la première présente plusieurs avantages par rapport à la seconde, tels qu'une préparation plus simple des échantillons, des exigences expérimentales moins restreintes et des applications d'imagerie plus respectueuses de la biologie. La sélection de la MET dans le domaine biologique est généralement choisie en raison de sa capacité à voir les organites intercellulaires et de l'imagerie ultra-résolution (généralement nanométrique ou sub-nanométrique) qui peut être obtenue. La cartographie de l'adhésion AFM dans le travail actuel a amélioré la résolution des caractéristiques et a présenté les arrangements des macromolécules de surface d'une manière qui ne dépendait pas de la taille de la pointe, mais plutôt de la structure intrinsèque de l'échantillon lui-même. De plus, cette approche permet une résolution à l'échelle nanométrique de la surface microbienne sur une plage de dizaines de micromètres carrés.
Différences de manipulation génomique dans les caractéristiques morphologiques
Après avoir observé l'ultrastructure de surface de E. coli MG1655, une question d'intérêt est de savoir comment les macromolécules sont arrangées dans d'autres souches. Les agents pathogènes humains E. coli CFT073 et RS218 ont donc été examinés par AFM avec les mêmes paramètres expérimentaux, et il n'y avait pas de différences significatives dans les formes et les dimensions cellulaires entre ces trois génotypes à la taille caractéristique de 10 μm, comme illustré sur la figure 3a–c. La cartographie de la force d'adhérence a été utilisée pour identifier les protéines de la couche S, et des structures dissemblables avec diverses formes et tailles ont été détectées parmi les différents E. coli souches, comme le montre la Fig. 3d–f. Pour faciliter la comparaison, les images détaillées de cartographie d'adhérence du E. coli les souches ont été affichées sur la figure 3g–i. Les macromolécules de surface ont été caractérisées comme ayant une forme ronde dans les cellules MG1655 et RS218, bien que des diamètres moléculaires différents, qui étaient 38 ± 1,1 nm (n = 80) pour MG1655 et 58 ± 2,7 nm (n = 46) pour RS218. D'autre part, les cellules CFT073 possédaient une forme unique de protéines de la couche S, qui ressemblaient à des reins avec une différence de longueur entre deux extrémités de 66 ± 3.2 nm (n = 44). Après avoir analysé les tailles des protéines de la couche S de ces trois génotypes pour de multiples comparaisons, les résultats ont démontré les différences significatives entre ces souches (Fig. 4).
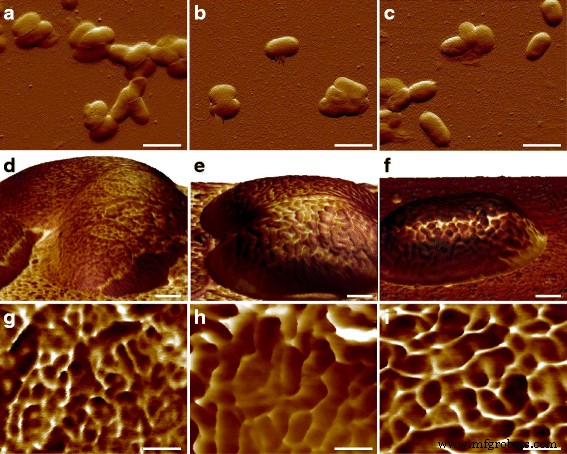
Les caractéristiques morphologiques de l'E. coli génotypes. La rangée supérieure affichait les images d'erreur de déviation de a MG1655, b CFT073 et c RS218. La rangée du milieu montrait les topographies 3D colorées avec la cartographie d'adhérence sur d MG1655, e CFT073 et f Cellules RS218. La rangée inférieure était la cartographie détaillée de l'adhérence sur g MG1655, h CFT073 et i RS218. Dans la cartographie d'adhérence, la couleur la plus foncée faisait référence à des performances d'adhérence moindres et vice versa. Les barres d'échelle étaient de 2 μm pour a –c , 200 nm pour d –f , et 100 nm pour g –je
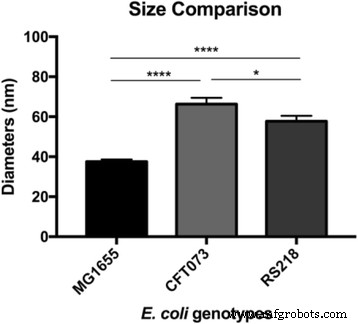
Tailles moléculaires de E. coli génotypes. Les diamètres des protéines de surface ont été détectés par AFM et traités par ANOVA à sens unique pour les comparaisons multiples. ****p < 0.001 et *p < 0,05
On rapporte que les couches S microbiennes jouent un rôle important dans de nombreuses fonctions, qui contiennent des cellules protectrices contre les environnements sévères, les attaques de phagocytose et les bactéries prédatrices. De plus, les couches S servent également d'adhésine qui permet une colonisation efficace [17]. Les structures de la couche S ont été bien étudiées par MET et ont été classées en divers types de réseau avec un espace entre centre à centre compris entre 4 et 35 nm [16]. La variation entre nos résultats AFM et les rapports TEM de la littérature a été considérée comme les différentes méthodologies d'imagerie, où TEM donne la morphologie 2D de la structure de la couche S et l'AFM capture la topographie 3D qui inclut les multiples influences contribuées par le radian cellulaire et la rugosité et le géométrie de la sonde AFM.
On pensait à l'origine que différents types de structures de la couche S pouvaient utiliser leurs diverses caractéristiques taxonomiques pour distinguer les espèces bactériennes, bien qu'il ait ensuite été découvert que même pour une seule espèce, les taches microbiennes pouvaient posséder différents réseaux de protéines [13, 16, 18, 19]. Alors que certaines études ont étudié le rôle de la couche S dans la formation des filaments, les types de protéines sur les membranes cellulaires et les diversités génomiques de taille parmi E. coli génotypes, les différences dans les protéines de la couche S ont rarement été notées [20,21,22]. Les résultats de la présente étude ont révélé les différences de caractéristiques morphologiques entre E. coli MG1655, CFT073 et RS218 et suggèrent que les apparitions des macromolécules de surface étaient probablement spécifiques à l'individu E. coli génotype.
Conclusions
Dans ce travail, des informations nanostructurales spécifiques à la génomique sur la surface bactérienne ont été détectées par cartographie mécanique AFM, qui distinguait les différences d'adhérence entre les macromolécules et la matrice environnante. Les macromolécules de surface des cellules microbiennes ont été considérées comme les protéines de la couche de surface, selon l'architecture moléculaire des microbes à Gram négatif. Les dispositions et les tailles de ces macromolécules se sont avérées spécifiques au E. coli génotypes avec des formes et des tailles distinctes, ces différences étant significatives par analyse statistique. En conclusion, nous considérons que la structure de la couche S bactérienne est dépendante du génome et peut être la méthode potentielle pour le diagnostic rapide des maladies associées aux microbes ou des souches microbiennes. Pour mettre en œuvre l'application pratique de la caractéristique de la couche S, l'examen sur plus de génotypes bactériens est requis pour le catalogue du complément. Nous établissons actuellement la base de données reliant les caractéristiques morphologiques bactériennes et les performances physiologiques/pathologiques et pensons que ce sera un progrès prometteur pour l'application pratique de l'examen AFM.
Abréviations
- AFM :
-
Microscope à force atomique
- ANOVA :
-
Analyse unidirectionnelle de la variance
- APTES :
-
3-Aminopropyltriéthoxysilane
- LB :
-
Bouillon de lysogénie
- QNM :
-
Nanomécanique quantitative
- Couche S :
-
Couche de surface
- TEM :
-
Microscopie électronique à transmission
Nanomatériaux
- Les scientifiques d'IBM inventent un thermomètre pour l'échelle nanométrique
- Pionniers de l'AFM récompensés par le prix Kavli
- Le prochain jaillissement de Big Oil est à l'échelle nanométrique
- Les non-conducteurs conduisent un courant à l'échelle nanométrique
- Synthèse biogénique, caractérisation et évaluation du potentiel antibactérien de nanoparticules d'oxyde de cuivre contre Escherichia coli
- Effet de surface sur le transport du pétrole dans les nanocanaux :une étude de dynamique moléculaire
- Diffusion aux petits angles à partir de fractales de graisse à l'échelle nanométrique
- Dépendance de résonance de plasmon de surface localisée sur un dimère de nanoprisme Ag tronqué mal aligné
- Fabrication à haut débit de nanofibres de qualité à l'aide d'un électrofilage à surface libre modifiée



