Analyse de l'actine et de l'organisation de l'adhésion focale dans des cellules U2OS sur des nanostructures polymères
Résumé
Contexte
Dans ce travail, nous explorons comment les cellules U2OS sont affectées par des réseaux de nanopiliers polymères fabriqués sur des surfaces de verre plates. Nous nous concentrons sur la description des changements dans l'organisation du cytosquelette d'actine et dans l'emplacement, le nombre et la forme des adhérences focales. À partir de nos résultats, nous identifions que les cellules peuvent être classées en différents régimes en fonction de leur comportement de propagation et d'adhésion sur les nanopiliers. Une analyse quantitative suggère que les cellules ensemencées sur des réseaux de nanopiliers denses sont suspendues au-dessus des piliers avec des adhérences focales se formant plus près de la périphérie des cellules par rapport aux surfaces planes ou aux réseaux de piliers clairsemés. Ce changement est analogue à des réponses similaires pour les cellules ensemencées sur des substrats mous.
Résultats
Dans ce travail, nous explorons comment les cellules U2OS sont affectées par des réseaux de nanopiliers polymères fabriqués sur des surfaces de verre plates. Nous nous concentrons sur la description des changements dans l'organisation du cytosquelette d'actine et dans l'emplacement, le nombre et la forme des adhérences focales. À partir de nos résultats, nous identifions que les cellules peuvent être classées en différents régimes en fonction de leur comportement de propagation et d'adhésion sur les nanopiliers. Une analyse quantitative suggère que les cellules ensemencées sur des réseaux de nanopiliers denses sont suspendues au-dessus des piliers avec des adhérences focales se formant plus près de la périphérie des cellules par rapport aux surfaces planes ou aux réseaux de piliers clairsemés. Ce changement est analogue à des réponses similaires pour les cellules ensemencées sur des substrats mous.
Conclusion
Dans l'ensemble, nous montrons que la combinaison de la nanofabrication à haut débit, de la microscopie optique avancée, des outils de biologie moléculaire pour visualiser les processus cellulaires et l'analyse des données peut être utilisée pour étudier comment les cellules interagissent avec les surfaces nanostructurées et contribuera à l'avenir à créer des substrats de culture qui induisent des fonction cellulaire.
Résumé graphique
Contexte
In vivo, les cellules résident généralement dans un environnement 3D complexe appelé matrice extracellulaire (ECM). L'ECM ne sert pas seulement d'échafaudage structurel pour les cellules, il est également un transporteur de signaux biomécaniques et biochimiques et régule ainsi une gamme de processus tels que la morphogenèse des tissus, l'homéostasie et la différenciation. Il est composé d'eau, de polysaccharides et de protéines [1,2,3,4], et la composition varie selon les types de tissus.
Motivés par le besoin de créer des modèles de culture cellulaire qui représentent mieux les conditions in vivo, les chercheurs ont de plus en plus commencé à étudier le comportement cellulaire également dans des matrices 3D et dans des systèmes « semi-3D ». Un certain nombre de différences dans les phénotypes cellulaires entre les substrats plats et les systèmes avec une dimensionnalité plus élevée ont été identifiés [5, 6]. Par exemple, des caractéristiques telles que la viabilité, la prolifération, la différenciation et la morphologie sont connues pour différer entre les cellules sur des surfaces planes et les cellules intégrées dans des matrices 3D [3, 7].
Les substrats de type in vivo vont des substrats « semi-3D »/2,5D, tels que les surfaces planes décorées avec diverses nanostructures, aux systèmes « vrai-3D » tels que les gels de collagène ou les matrices de matrigel [8,9,10,11]. De plus, le positionnement contrôlé des ligands sur les surfaces peut donner de nouvelles informations sur la façon dont les cellules interagissent avec divers modèles chimiques [12,13,14]. Il a également été démontré que des facteurs mécaniques tels que la rigidité de la structure ou même la chimie de surface influencent la fonction cellulaire [15,16,17]. À cette fin, un grand nombre de substrats différents pour les études cellulaires ont été développés [3, 18,19,20,21,22].
Il a également été suggéré que les systèmes de culture 3D pourraient prédire plus précisément l'effet in vivo d'un médicament et ainsi ces systèmes pourraient trouver des applications dans la découverte de médicaments [16, 23, 24]. Le contrôle précis des motifs topographiques à l'échelle nanométrique peut également être utilisé pour réguler la morphologie cellulaire. Par exemple, les rides et les rainures peuvent être utilisées pour recréer l'alignement strié des cardiomyocytes et ainsi mieux représenter les conditions physiologiquement pertinentes pour modéliser diverses maladies [25, 26].
Le cytosquelette de la cellule est connecté à l'ECM est facilité par les adhérences focales (FA), un complexe multiprotéique comprenant des intégrines de surface cellulaire et des protéines d'échafaudage. En fonction d'un ensemble complexe de mécanismes de régulation, les AG se forment et se désassemblent à un taux de renouvellement nécessaire au mouvement vers l'avant, par exemple lors de la migration cellulaire. Les AG sont connus pour exercer une force mécanique sur l'ECM, et inversement, l'ECM exerçant une force sur les cellules est connue pour influencer l'affinité et l'avidité des intégrines dans la membrane [27].
L'une des protéines connues pour faire partie intégrante des AG est la vinculine. C'est l'une des protéines de liaison impliquées dans l'ancrage de la F-actine au complexe d'intégrine. Le manque de vinculine altère la morphologie, l'adhésion et la motilité des cellules [28], et altère la capacité des cellules à transmettre la force au substrat [29,30,31]. La vinculine n'est pas seulement impliquée dans la connexion mécanique du cytosquelette d'actine aux complexes d'intégrines, elle a également la capacité de réticuler et de regrouper les filaments d'actine [32,33,34], de modifier les faisceaux d'actine existants [35], de coiffer les filaments d'actine, nucléer de nouveaux sites de polymérisation d'actine [36] et recruter des modificateurs d'actine [37].
Les cellules répondent aux matrices 3D en modifiant le nombre et le type d'adhésion cellule-substrat et induisent des changements dans l'organisation spatiale du cytosquelette. Ces changements influencent à leur tour la distribution, la taille et la dynamique des adhérences formées [4, 38, 39, 40, 41]. Ce réarrangement peut entraîner des changements dans la prolifération, la morphologie et la motilité des cellules [42].
Afin de comprendre l'influence des environnements 3D complexes sur les cellules, il est nécessaire de développer de nouveaux systèmes modèles où les processus cellulaires peuvent être étudiés et comparés à des contrôles plats. Comme la réponse cellulaire est connue pour dépendre des caractéristiques physiques, mécaniques et chimiques du substrat de culture, il est souhaitable de fabriquer des substrats cellulaires avec des propriétés contrôlées avec précision [43,44,45]. De plus, il est très avantageux que les cellules et le substrat puissent être facilement étudiés en utilisant des techniques d'analyse déjà établies telles que la microscopie optique.
Un type de substrat qui a récemment attiré l'attention sont les surfaces planes décorées de nano-piliers ou de nano-fils [18, 21, 46,47,48,49,50,51,52,53]. Par rapport aux hydrogels par exemple, ces surfaces structurées n'imitent pas le véritable environnement 3D, mais ont une topographie de surface bien définie. Ces substrats sont généralement appelés 2.5D. De tels systèmes ont déjà été appliqués pour faciliter l'administration de molécules biologiquement pertinentes dans les cellules [54, 55], pour surveiller l'activité enzymatique [56], pour tester la mécanique nucléaire [57] et pour étudier comment le réglage de la courbure de la membrane influence divers éléments liés à la membrane cellulaire. processus [58,59,60]. En fabriquant des nanostructures sur des substrats transparents, il est possible d'intégrer cette approche à la microscopie optique.
Le nombre de combinaisons possibles de différentes lignées cellulaires, le type de nanostructure et la géométrie est élevé, et les exemples de la littérature sont abondants. Li et al. ont décrit le comportement des cellules sur des surfaces décorées de nanostructures de phosphure de gallium positionnées de manière aléatoire et ont quantifié la fraction de cellules avec de grands AG [61]. La morphologie de la cellule et de l'AF a été étudiée sur la surface avec diverses densités de surface de nanofils. Les résultats ont indiqué que les cellules ensemencées sur des surfaces à faible densité étaient en contact avec le substrat et formaient de grandes AF autour des bords des cellules. Des AG de grande taille ont été détectés dans une fraction élevée de cellules sur ces puces. Pour les densités surfaciques élevées de nanofils, les cellules ont été suspendues au sommet des réseaux de nanofils et des FA ponctuels sous les cellules ont été observées. Une fraction inférieure de cellules sur ces réseaux a montré de grandes AF par rapport aux cellules sur la surface avec une faible densité de surface de nanofils.
Buch-Månson et al. ont étudié les interactions cellule-surface nanostructurées pour des matrices de nanocolonnes de silicium positionnées de manière aléatoire sur un substrat de Si [62]. Dans le processus de fabrication utilisé, la densité surfacique mais pas la distance pilier-pilier a été contrôlée. L'enquête sur les AF a montré que les cellules sur les réseaux avec la densité surfacique intermédiaire avaient le plus grand nombre d'AF qui avaient également la forme la plus asymétrique. Il a été suggéré que certains de ces AG se sont formés sur les parois latérales des nanocolonnes. Cela n'a pas été observé pour les surfaces avec une densité surfacique de nanocolonnes faible et élevée.
Dans des travaux antérieurs, nous avons décrit des protocoles détaillés pour la fabrication de nanostructures de polymère SU-8 sur des surfaces en verre plat [63], et exploré le comportement cellulaire pour deux lignées cellulaires différentes sur ces surfaces [45, 48]. Dans ce travail, nous utilisons la lithographie par faisceau d'électrons (EBL) pour fabriquer des surfaces décorées de structures polymères SU-8 alignées verticalement afin d'étudier les changements dans l'organisation du cytosquelette d'actine et de l'AF dans la lignée cellulaire épithéliale d'ostéosarcome U2OS. Nous effectuons une analyse à la fois qualitative et quantitative des changements induits par la surface avec différents indices topologiques.
Résultats
En utilisant des protocoles précédemment établis, nous avons fabriqué des lamelles de verre décorées avec des réseaux définis avec précision de nanopiliers SU-8 (NP) orientés verticalement avec une séparation variable et une géométrie définie [63]. Surfaces avec des densités surfaciques NP de 456, 205, 115 et 29 NPs/100 μm 2 (correspondant à des pas de 500 nm, 750 nm, 1000 nm et 2000 nm) ont été utilisés. Tout d'abord, nous examinons les tendances générales de la morphologie cellulaire, la structure du cytosquelette d'actine et les interactions cellule-substrat. Nous suivons ceci par une comparaison quantitative de la morphologie des cellules et des AG sur divers substrats nanostructurés et témoins de verre plat. Nous combinons la microscopie haute résolution avec la fabrication à haut débit pour effectuer une analyse qualitative d'au moins \({\environ 100}\) cellules pour chaque type de surface, avec imagerie après 24 h et 48 h. Au total, nous analysons> 400 images haute résolution et 20 ensembles de données 3D.
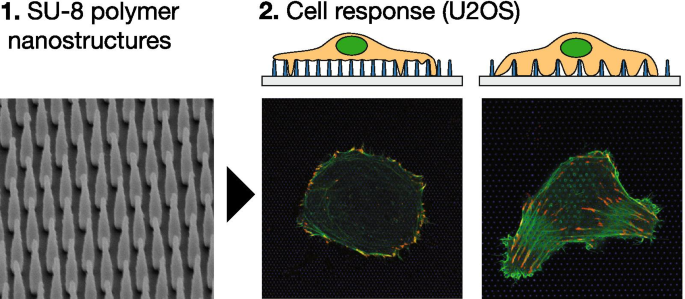
La figure 7 montre une représentation schématique des réseaux NP (A, B) et des images de microscopie électronique de substrats fabriqués (C, D). Des lames de verre contenant des structures nanofabriquées ont été montées à l'aide de paraffine sous fond creux, culture de 35 mm avec les structures dirigées vers le haut. Les images de microscopie électronique à vue latérale de haut en bas et titrées illustrées, sur la figure 1C, D, montrent des réseaux de nano-piliers avec un pas et une hauteur de 1 000 nm. Le tableau 1 montre les paramètres géométriques des tableaux utilisés dans ce travail, leur classification, ainsi que la densité numérique de zone NP correspondante. Nous classons les réseaux NP en denses et clairsemés en fonction du comportement d'adhésion cellulaire observé (voir ci-dessous).
La figure 1 montre des cellules U2OS représentatives cultivées sur du verre (A) et une surface nanostructurée (B–F) pendant 24 h. Les cellules ont été co-transfectées avec pCMV-LifeAct-GFP et pTAG-RFP-Vinculin, qui permettent la visualisation de la F-actine et de la vinculine via la production de protéines de fusion fluorescentes LifeAct-TagGFP2 (ci-après :LifeActGFP) et TagRFP-vinculine respectivement. Le réseau F-actine et les zones riches en vinculine dans les AG sont clairement détectés. À la périphérie de la cellule, le signal LifeActGFP est à proximité immédiate de la membrane et nous utilisons donc ce signal pour visualiser la morphologie cellulaire. Le signal des NP SU-8 est affiché en bleu (voir la section « Expérimental »).
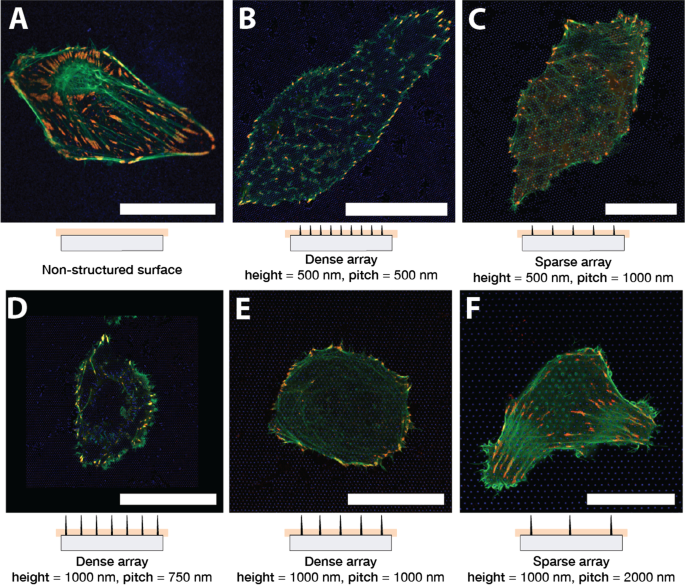
U2OS exprimant LifeActGFP fluorescent (vert) et TagRFP-vinculin (rouge) sur différents types de surfaces. La coloration jaune indique des signaux qui se chevauchent des canaux LifeActGFP et TagRFP-vinculine. Sous chaque micrographie, une vue latérale schématique de la matrice NP correspondante est affichée, ainsi que la position approximative du plan d'acquisition. Cellules imagées sur A surface de verre plat non structuré et sur des réseaux de piliers avec différents pas de réseau et hauteurs de structure :B Pas de 500 nm et hauteur de 500 nm, C Pas de 1 000 nm et hauteur de 500 nm, D Pas de 750 nm et hauteur de 1 000 nm, E Pas de 1 000 nm et hauteur de 1 000 nm, et F Pas de 2000 nm et hauteur de 1000 nm. Toutes les images présentées sont des cellules représentatives. Barre d'échelle 25 μm. Notez que C a une échelle différente des autres images
Des tests préliminaires utilisant des cellules transfectées ont montré que les cellules ensemencées à la fois sur le verre et les surfaces structurées semblaient être complètement étalées après environ 6 h. Aucune différence nette dans l'étalement des cellules entre les différentes surfaces n'a été observée, et l'inspection visuelle n'a révélé aucun signe de diminution de la viabilité des cellules en raison des réseaux de piliers à ce stade ou lors d'expériences ultérieures. Dans les expériences suivantes, les cellules ont été transfectées 6 h après l'ensemencement, puis imagées 24 h, 48 h après la transfection, correspondant à la transfection 30 h, 54 h après l'ensemencement. Dans la suite, ces deux points temporels seront référencés par le temps d'observation après semis, soit 24 h, 48 h.
Les cellules ont été ensemencées sur des matrices NP avec une hauteur de 500 nm, 1000 nm et un pas de 750 nm, 1000 nm, 2000 nm. Après l'étalement initial, on a observé que les cellules étaient soit rondes, soit allongées, comme c'est le cas sur des surfaces planes. Cette morphologie générale s'est avérée cohérente au cours de plusieurs expériences. Les cellules ensemencées sur des réseaux NP clairsemés avaient généralement une forme similaire aux cellules sur une surface de verre, voir la figure 1F représentant une cellule représentative sur un réseau à pas de 2000 nm. Des fibres d'actine F étaient également présentes à la base des NP et à proximité de la surface du verre, indiquant que les cellules étaient capables d'accéder à la zone proche du substrat. Comme observé précédemment [45, 62, 64], les cellules sur des réseaux denses semblaient généralement être suspendues au-dessus des NP (Fig. 1B, D, E). Les cellules sur des réseaux denses semblent avoir de l'actine F moins importante près de la surface du verre, ce qui indique que les fibres d'actine ne se sont pas formées entre les piliers à proximité du substrat. La relation entre la hauteur de NP et la séparation a déterminé si les cellules adhéraient au substrat ou étaient suspendues au sommet de la matrice NP. Ceci est par exemple illustré sur la figure 1C où les NP plus courtes conduisent la cellule à entrer en contact avec le substrat, tandis que les NP plus longues entravent le contact, figure 1E. Ces observations de fibres d'actine ont été corroborées par des z-stacks effectués pour certaines des surfaces, comme le montre la figure 2.
Pour obtenir une compréhension plus détaillée de la façon dont les cellules adhèrent à la surface structurée et non structurée, nous avons évalué la distribution des AG telle que visualisée par la présence de la protéine de fusion TagRFP-vinculine. Les cellules sur des surfaces planes formaient généralement des AF allongés répartis sous l'ensemble du corps cellulaire, comme le montre la figure 1A.
Sur les réseaux clairsemés, U2OS a pu entrer en contact avec la surface du verre entre les NP et a adhéré de la même manière aux cellules sur verre, illustrées à la Fig. 1C, F. Pour ces réseaux de NP, les AF se sont formés sur le verre entre les NP et le signal F-actine a également été détecté dans l'image acquise près de la base des NP. Cela indiquait que les cellules étaient capables de plier la membrane autour des nanostructures. Cependant, les cellules sur des matrices plus denses, telles que 750 nm, 1 000 nm nm de séparation et 1 000 nm de hauteur, étaient clairement empêchées d'adhérer au substrat entre les nanostructures, comme le montrent les images Fig. 1D, E qui ont été acquises près de la base du NPs. Cependant, à la périphérie, les cellules étaient généralement capables de se fixer au substrat entre les nanostructures formant des FA, souvent dirigées par la symétrie du réseau de piliers sous-jacent.
Les cellules s'étalant sur des réseaux NP de longueur plus courte et avec un espacement inter-piliers de 1 000 nm ont formé des adhérences à la fois vers la périphérie et sous le corps cellulaire. L'orientation des fibres d'actine F était dirigée par la symétrie du réseau sous-jacent, comme le montre la figure 1C. Cependant, l'emplacement et l'orientation des AG contenant de la vinculine n'ont montré aucun schéma clair, l'AF se formant entre les NP.
Les cellules U2OS sur des piliers de 500 nm avec une distance inter-piliers de 500 nm formaient généralement des adhérences moins nombreuses et plus petites par rapport à la surface plane, comme le montre la figure 1B. Pour les cellules ensemencées sur ce réseau, les fibres d'actine ont été principalement observées à proximité de la surface du verre à des positions où elles se terminaient par des AG. Encore une fois, c'est un signe que le réseau d'actine a été empêché d'entrer en contact avec la surface, et les cellules ont donc été supposées être suspendues au-dessus de la matrice. Cependant, les cellules semblaient avoir un réseau d'actine F plus intact se formant au-dessus des piliers. Des parties du réseau d'actine observées entre les piliers semblaient s'aligner sur les structures du réseau NP sous-jacent. Cela peut être vu sur la figure 1B, car les fibres d'actine F et les AG se forment principalement le long de l'une des directions du réseau, c'est-à-dire parallèlement aux « lignes » ouvertes du réseau de piliers.
Sur les réseaux denses et clairsemés, nous avons observé des structures d'actine F « en forme d'anneau » qui se sont formées autour de NP qui fait saillie vers le haut dans le corps cellulaire. La structure en anneau F-actine semble être plus importante sur les réseaux clairsemés, comme le montre la figure 1E, F.
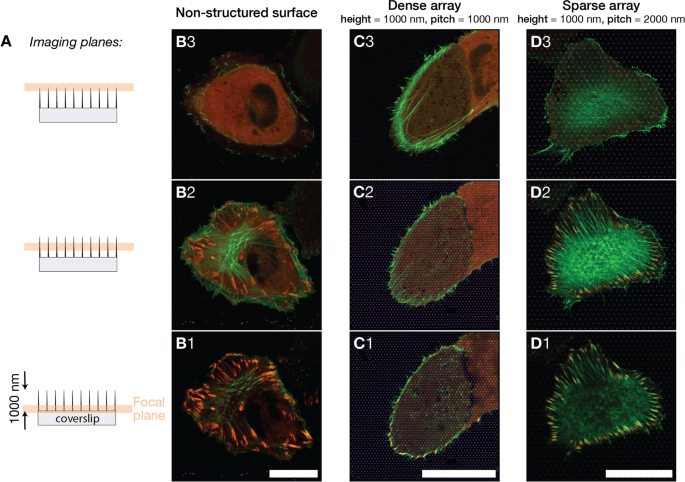
Un Des images de cellules U2OS sur différentes surfaces ont été acquises à trois plans focaux indiqués. Les images ont été obtenues près de la base du pilier, approximativement à mi-hauteur du pilier et près de l'apex du pilier. Les plans d'imagerie étaient situés à 0,0 μm, 0,4 μm, 0,8 μm de la surface du verre plat. B –D ) Images de fluorescence fusionnées montrant U2OS exprimant LifeActGFP fluorescent (vert) et TagRFP-vinculin (rouge) sur différents types de surface, la couleur jaune indique le chevauchement de LifeActGFP et TagRFP-vinculin. Les surfaces imagées étaient B surface vitrée non structurée, C Réseau de piliers inclinés à 1 000 nm, D Réseau de piliers inclinés à 2000 nm. Les images présentées sont représentatives des cellules de chaque type de surface. La distance verticale entre les plans focaux pour chaque type de surface est d'environ 400 nm. Barres d'échelle 25 μm
Sur la base des résultats présentés ci-dessus, nous avons sélectionné trois surfaces pour une description plus détaillée et quantitative de la morphologie cellulaire et des AG. Nous étudions des cellules sur des matrices denses (pas 1000 nm, longueur 1000 nm), des matrices clairsemées (pas 2000 nm, longueur 1000 nm) et comparons les résultats avec des cellules sur des surfaces de verre plates utilisées comme contrôle.
En utilisant le détecteur Airyscan avec le post-traitement d'image dédié, nous avons pu réaliser une imagerie avec une résolution xy d'environ 140 nm et une résolution z d'environ 400 nm [65]. La figure 2 montre des images de cellules sur les trois surfaces, avec des plans d'imagerie séparés d'environ 400 nm. L'étude des faisceaux d'actine F à différents z . Les cellules sur une surface plane avaient un réseau d'actine F clairement visible au même plan focal ou juste au-dessus des AF (voir Fig. 2-B2). Pour les cellules sur des réseaux denses, le réseau F-actine a été trouvé sur un plan focal plus élevé dans la cellule par rapport au plan FA, qui était en contact avec le support en verre (Fig. 2-C1 et C2/C3). Pour les cellules sur des matrices clairsemées, la situation était similaire à celle des cellules sur les contrôles en verre et le réseau d'actine et les AG ont été détectées à la même hauteur (Fig. 2-D2). Ces données corroborent l'observation initiale selon laquelle les cellules des réseaux clairsemés attachaient la surface entre les structures, tandis que les cellules des réseaux denses étaient principalement capables d'adhérer à la surface autour de la périphérie des cellules.
Pour analyser et quantifier les différences d'AF et de morphologie cellulaire pour les trois surfaces sélectionnées, un script d'analyse d'image basé sur Python a été utilisé (voir « Expérimental »). Pour l'analyse quantitative, plus de 300 images à haute résolution ont été analysées. Dans ces images,> 400 cellules et> 7700 AF ont été identifiés, le tableau 2 répertorie le nombre de cellules et d'AF détectés pour les trois types de surface inclus dans l'analyse. Pour toutes les surfaces, les cellules ont été imagées à la fois 24 h et 48 h après la transfection. Dans l'analyse suivante, les paramètres géométriques tels que la surface, la circularité et le rapport hauteur/largeur pour les cellules sur 3 types de surface et après 24 h, 48 h sont comparés. Une analyse supplémentaire peut être trouvée dans les informations supplémentaires. La surface, la circularité et le rapport hauteur/largeur des cellules sont indiqués sur la figure 3 et pour le nombre d'AF, la zone FA combinée par cellule et la fraction de la zone FA par cellule sur la figure 4. Les paramètres géométriques ont été définis comme décrit dans le « Experimental » section.
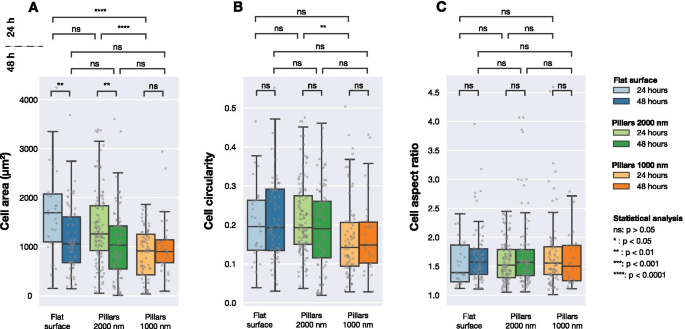
Surface cellulaire calculée, circularité cellulaire et rapport d'aspect cellulaire pour les cellules U2OS exprimant les protéines de fusion fluorescentes LifeActGFP et TagRFP-vinculine sur différentes surfaces imagées 24 h et 48 h post-transfection. La détection d'image a été effectuée sur la base de l'intensité du signal LifeActGFP. Chaque point gris correspond à une cellule et les box plots affichent les valeurs médianes (Q 2) ainsi que le premier (Q 1) et troisième quartile (Q 3). Les différences statistiques entre les distributions ont été évaluées à l'aide du test non paramétrique de Mann-Whitney
La figure 3 résume les données recueillies pour les surfaces planes et structurées. Comme le montre la figure 3A, des différences significatives dans la surface cellulaire ont été observées après 24 h de culture cellulaire. Cependant, après 48 h, il n'y avait pas de différence significative entre la surface cellulaire moyenne sur les surfaces étudiées. Lors de l'examen de la circularité cellulaire (Fig. 3B), aucune différence significative n'a été détectée entre les différentes surfaces, sauf entre les cellules ensemencées sur des piliers denses et clairsemés imagés après 24 h. Les cellules sur les trois surfaces avaient les mêmes rapports d'aspect moyens, comme le montre la figure 3C.
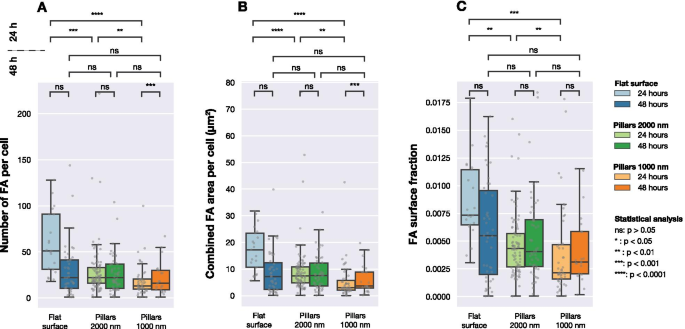
Nombre de FA, zone FA combinée par cellule et fraction de zone FA par zone cellulaire pour les cellules imagées sur trois types de surface différents (réseaux plats et à piliers avec des pas de 1 000 nm et 2 000 nm). Chaque point gris correspond à une observation d'une cellule. La signification statistique entre les distributions a été déterminée à l'aide du test non paramétrique de Mann-Whitneys
La figure 4 montre la distribution du nombre d'AF détectés par cellule, la surface totale des AG dans chaque cellule et le rapport entre l'aire d'AF et la surface de la cellule. Après 24 h, le nombre d'AF formés par les cellules sur les trois surfaces différentes était significativement différent. Comme le montre la figure 4B, la surface totale de FA par cellule était différente pour les cellules ensemencées sur des surfaces planes et structurées. La même chose peut être observée en comparant la quantité relative d'AF (la surface totale d'AF détectés divisée par la surface cellulaire totale) pour les différentes surfaces, comme le montre la figure 4C.
Cependant, après 48 h de culture, les différences significatives entre les populations cellulaires n'étaient plus observées. Lorsque l'on considère le nombre d'AF par cellule, la surface d'AF combinée par cellule ou la fraction de surface d'AF, aucune différence entre les trois surfaces n'a été trouvée.
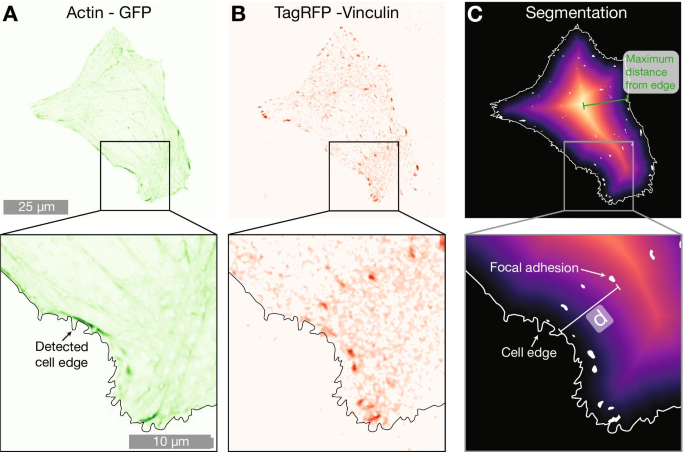
Exemple de cellule sur un tableau de piliers à 2 000 nm exprimant A LifeActGFP (vert) et B TagRFP-vinculine (rouge). Superposé sur les deux figures est le bord de cellule détecté déterminé en utilisant le signal de l'expression de LifeActGFP (comme indiqué dans A ). C Affiche une carte de distance depuis le bord de cellule détecté ainsi que les FA détectés. La distance la plus courte (notée d dans la figure) de chaque tache de vinculine segmentée (zones blanches) à la périphérie de la cellule (indiquée par une ligne blanche continue) a été calculée pour toutes les AF dans les images
Pour comprendre si la présence de NP influence la localisation des AF dans la cellule, nous avons effectué une analyse plus approfondie en utilisant l'emplacement des AF. Les données de microscopie ont indiqué que les AF dans les cellules sur des réseaux de NP denses étaient situés plus près de la périphérie des cellules, comme indiqué par les Fig. 1 et 2. Pour quantifier cette tendance, nous avons calculé la distance la plus courte entre chaque FA et le bord de la cellule. Cela a été réalisé comme illustré sur la figure 5. La F-actine a été utilisée pour déterminer l'emplacement de la périphérie et en construisant des cartes de distance, la distance entre chaque centre d'une FA détectée à la périphérie de la cellule a été calculée. Pour tenir compte des différences de tailles de cellules, nous avons normalisé les distances entre le bord de cellule détecté et FA par la distance maximale du bord au centre géométrique pour chaque cellule (une distance équivalente au rayon pour les cellules qui ont une forme circulaire). Les données normalisées par la distance maximale sont présentées dans la figure 6 et le tableau 3. Pour les cellules sur des surfaces planes, les FAs sont distribués plus vers le centre de la cellule, alors que sur les réseaux clairsemés de 2000 nm et les réseaux denses de 1000 nm, les FAs sont situés plus près de la périphérie de la cellule. Cet effet est particulièrement notable pour les réseaux denses avec un pas de 1 000 nm. Les résultats d'une approche de normalisation alternative, où les emplacements FA ont été normalisés par surface cellulaire, sont inclus dans les informations supplémentaires. Ces données montrent les mêmes tendances qualitatives que les données normalisées par la distance maximale au bord dans chaque cellule présentée sur la figure 6.
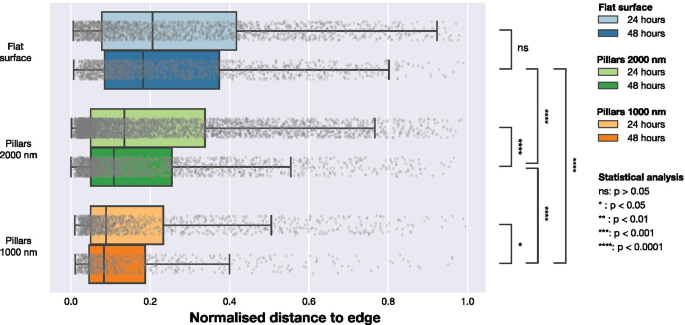
Distribution des positions des FAs par rapport au bord de cellule le plus proche normalisée par la distance maximale du centre géométrique de la cellule au bord. Les distances ont été obtenues en calculant la distance de chaque FA observée au bord de la cellule définie par le signal LifeActGFP et tracée pour les trois types de surface à 24 h et 48 h. Les points gris représentent les observations individuelles des AF et leurs distributions sont résumées dans les boîtes à moustaches. Pour tester la probabilité que les FA des différentes surfaces et points temporels appartenaient à la même distribution, des tests de Mann-Whitney ont été effectués sur toutes les distributions avec des niveaux de signification indiqués dans la figure
Discussion
L'organisation du cytosquelette d'actine et la formation d'adhérences sont des processus largement étudiés sur des surfaces planes. La présente étude a été conçue pour étudier et quantifier les changements dans l'organisation du cytosquelette d'actine et les adhérences focales sur les réseaux de nanopiliers. Dans cette étude, nous n'avons pas étudié comment les réseaux de nanopiliers affectent la migration cellulaire. Cependant, nous nous attendons à ce que les cellules des réseaux denses aient une motilité plus élevée, comme observé précédemment sur les mêmes réseaux à piliers pour les fibroblastes embryonnaires de souris [45].
Laisser les cellules se propager et adhérer à la surface pendant une période de temps prolongée permet d'observer l'organisation du cytosquelette d'actine et la présence d'AG complètement matures. 24 h après l'ensemencement, nous observons des différences significatives de surface cellulaire, de circularité et de rapport hauteur/largeur pour les cellules ensemencées sur les surfaces. Cependant, aucune différence significative n'a été détectée après 48 h, indiquant que les cellules après 24 h n'ont pas encore complètement adhéré aux surfaces, et que les nanostructures influencent principalement l'organisation de l'AF avant qu'elles ne soient complètement matures. Ni après 24 h ni après 48 h, aucune formation de FA n'est observée au-dessus ou sur les côtés des NP.
Les changements dans l'organisation du cytosquelette d'actine sont également liés à la façon dont une cellule interagit avec son environnement. Par exemple, les fibres de stress et les AG se développent lorsqu'elles sont soumises à un étirement et semblent être fonctionnellement interdépendantes [66]. D'autres ont rapporté que des cellules plus arrondies et une localisation AF autour des bords sont souvent observées pour les cellules ensemencées sur des surfaces molles ou souples [67]. Dans nos résultats, nous observons une tendance similaire. Les cellules sur des réseaux denses ont tendance à montrer des fibres autour du bord de la cellule, comme le montrent les Fig. 1E ou 2B. Les AF semblent se former près du bord de la cellule pour ces cellules. Nous supposons que lorsque les cellules n'ont pas de surface plane, comme lorsque les cellules sont suspendues au sommet des piliers, la distribution des AG ressemble aux AG sur des substrats mous, comme ceux utilisés par Prager-Khoutorsky et ses collègues [67].
D'après nos résultats, nous observons que les cellules suspendues au sommet des piliers semblent avoir un réseau d'actine développé au-dessus des piliers, mais sans AG se formant sur les piliers eux-mêmes. Cependant, pour les cellules sur des réseaux clairsemés, les cellules semblent être moins influencées par les NP et le réseau d'actine et les AF semblent plus « à surface plane ».
L'interaction entre les AG et le cytosquelette d'actine est complexe et encore mal caractérisée. Les AG reliant le cytosquelette d'actine à l'ECM sont connus pour agir comme des points de traction et pour favoriser la formation de fibres de stress dans les cellules. A l'inverse, les fibres d'actine influencent à nouveau l'organisation et la maturation des AG. De nombreuses études décrivent comment les cellules ont tendance à être suspendues au-dessus de réseaux de NP denses [61, 62, 68] et comment les membranes cellulaires interagissent avec des NP uniques [69, 70, 71]. Ces observations sont corroborées par des études théoriques [64] et le comportement cellulaire sur piliers est assez bien compris.
Le mécanisme derrière la formation et la fixation des FAs au substrat autour du bord de la cellule sur des réseaux denses reste incertain. A cet égard, la comparaison avec des cellules sur substrat mou est particulièrement intéressante. Pour les substrats mous, les fibres d'actine sont organisées en anneau près du bord de la cellule et les AG se forment autour de la périphérie de la cellule [67]. On the nanopillar arrays similar type of architecture is observed, but the actin fibres are typically shorter. Similar qualitative trends in terms of actin organisation and FA formation were observed by Li et al. for cells seeded on random nanowire arrays made from gallium phosphide [61].
In our studies we also observed formation of F-actin rings around NPs. The formation of F-actin rings around NP has previously been described for fibroblasts on similar surfaces [45] and for U2OS cells on nanostructures with a range of structure sizes [58].
Contrasting our results to other studies highlight an important aspect of studies on cellular response to NP arrays:cellular response may vary considerably depending on cell type, NP material, NP geometry and as well as other parameters. For example, Buch-Månson et al. studied fibroblasts and investigations of FAs showed that cells suspended on arrays with intermediate NP density had the highest number of FAs. In our results we do not see a similar trend. However, these studies cannot be directly compared as Buch-Månson et al. studied another cell line using a system with different array geometry, surface porosity and NPs length [62].
There are also studies describing the effect FAs placement has on cells [41]. By modelling cells on planar substrates Stolarska et al. suggest that the cells can control intra-cellular stresses by three mechanisms:FA position, FA size and attachment strength. FAs around the periphery allows the cells to be more sensitive to changes in the micro-environment. This could also be an underlying mechanisms for cells on NPs. Yet, it is not obvious that the results for the planar substrate are directly transferable to NP decorated surfaces.
Cell-interactions with the surrounding environment, for flat substrate, NPs arrays or in vivo ECM, are regulated by a complex set of relations between actin organisation, membrane mechanics, cell dynamics and contact with FAs. To further explore these relations, applying flat surfaces structured with NPs could be one promising approach. Such surfaces may also aid in exploring discrepancies in the cellular response to environmental cues between different cell lines.
Conclusions
In order to create more physiologically relevant systems for cellular studies, a plethora of 3D and 2.5D approaches have been proposed. One approach is to use flat-surfaces decorated with vertically aligned nanostructures as a simple model system. High resolution live cell imaging of co-transfected U2OS cells expressing pCMV-LifeAct-GFP and pTAGRFP-Vinculin have been used to study the influence of nanopillar arrays on actin cytoskeleton focal adhesion organisation. Our present results indicate that the U2OS cells spreading on surfaces decorated with nanopillars can be categorised into three different regimes by how they respond to the nano-structures. These observed changes are quantified by analysing more than 400 high-resolution images, and indicate that tuning geometrical properties of the nanostructured surface can be used to direct cell behaviour.
More specifically, the U2OS cells were found to either contact the substrate, attach preferably around the cell edge, or be fully suspended on top of the vertical NP arrays. In the latter case, we hypothesise that the resulting reorganisation of FA and cytoskeleton is an effect analogous to what is seen for softer substrates.
Increased understanding of how cells behave on nano-structured surfaces, such as pillar arrays, could help us discover more details about complex cellular processes. For example, it is still poorly understood how changes in the actin cytoskeleton and its architecture influence cell signalling. By studying the cell response on nanostructured surfaces in a systematic way, the potential connection between actin cytoskeleton, cell adhesions and a plethora of biochemical signalling pathways could be further explored. We therefore envision that further development of the presented platform and analysis could have implications for advanced in vitro applications or for development of smarter in vivo biointerfaces.
Methods
Fabrication of Nanostructures and Sample Mounting
SU-8 nanostructures were fabricated as previously explained [63]. Briefly, 24 mm by 24 mm glass cover slips (#1.5, Menzel-Gläser, thickness 170 μm) were cleaned by immersion in acetone, isopropyl alcohol, rinsed in de-ionised water and dried. The cover slips were then oxygen plasma treated for 2 min (Diener Femto plasma cleaner, power 100 W, base pressure 0.3 torr), followed by dehydration for 10 min on a 150 °C hot plate. Samples were then placed in a desiccator containing an open vial of Hexamethyldisilazane (HMDS, Sigma Aldrich product no:440191). HMDS was applied by vapour deposition, the desiccator was pumped to low vacuum using a diaphragm pump for 5 min and the samples were kept in HMDS atmosphere for 60 min.
Substrates for EBL were prepared directly after HMDS treatment by spin coating SU-8 2001 (Microchem Corp.) to a desired thickness of 500 nm and 1000 nm. SU-8 was made fluorescent by adding either Oxazine 170 perchlorate, Rhodamine 800 or Coumarin 102 (all Sigma Aldrich) to a final concentration of 100 μg mL −1 resist. After spin coating samples were dehydrated on a hot plate at 95 °C. To mitigate charging during EBL exposure samples were then covered by a layer of conductive polymer AR-PC 5091 Electra 92 (AllResist GmbH) by spin coating at 2000 rpm for 60 s to thickness of 50 nm.
An Elionix ELS-G100 100 kV EBL-system was used to fabricate SU-8 nanopillars (NPs) with processing parameters as described in our previous work [63]. Table 1 summarise the arrays fabricated for this work. Pillar arrays were exposed using the Elionix dot-pattern generator where each pillar is exposed in a single exposure. Arrays were exposed over an area of 2000 μm by 4000 μm, with a current of 500 pA in write fields of 500 μm by 500 μm. NPs had a tip diameter of about 100 nm as a base diameter of 150 nm and 200 nm for structures of length 500 nm and 1000 nm respectively.
After EBL exposure, the samples were rinsed in DI-water to remove the conductive polymer, then post exposure baked for 2.3 min at 95 °C and developed twice in mr-Dev 600 (Micro Resist Technology GmbH) developer for 20 s, rinsed in isopropyl alcohol and dried. Samples were then treated with oxygen plasma (Diener Femto plasma cleaner, power 50 W, base pressure 0.3 torr) for 30 s to render SU-8 hydrophilic and to give it similar surface chemistry as glass by oxidising surface epoxy-groups to hydroxyl.
Fabricated structures were imaged using Scanning electron microscopy (SEM) and samples sputter coated with 5 nm Platinum/Palladium alloy deposited with a 208 HR B sputter coater (Cressington Scientific Instruments UK). SEM was performed with a FEI Apreo SEM, at 5 kV and 0.2 nA with sample 45° pre-titled stage and with additional tilting of 30°.

Un Side view schematic representation of nano-structured surface mounted in petri dish. Glass slides are mounted using paraffin such that structures are pointing upwards. B Tilted schematic representation of nano-pillar array on flat surface, and two important parameters for the nano-pillar arrays (height and pitch). These figures are not drawn to scale. C , D Overview of the nanopillar arrays employed in this work. Top-down and tilted side-view scanning electron micrographs of fabricated nano-pillar array with pillars of height 1000 nm and pitch 1000 nm. Scalebars 2000 nm
When exposing the pillars, an indexing system was also exposed to make navigation during live-cell imaging more reliable. Arrays were optically inspected after fabrication to ensure free and standing pillars. The short Oxygen plasma treatment to render the SU-8 structures did not lead to any optically visible change to the structures. Lastly, the samples were mounted underneath 35 mm diameter dishes (Cellvis, Mountain View, CA, USA) with 14 mm holes and nano-structures pointing upwards, as indicated schematically in Fig. 7. As flat surfaces, areas outside the structured part of the same samples were used. Before usage, all dishes were disinfected with 70% ethanol twice and dried.
Cell Culture and Transfection
U2OS-cells (ATCC) were cultivated in Dulbecco’s modified Eagle’s Medium (DMEM Prod. 41965039, Fischer Scientific) with 10% fetal bovine serum (FBS) and kept at 5% CO2 and 37 °C. Before detachment, cells were washed with PBS and detached with Trypsin-ethylenediaminetetraacetic acid (trypsin-EDTA) and seeded on nanostrucutred or flat surfaces. For the diameter 14 mm glass wells 15,000 cells were seeded.
For the standard transfection experiments, cells were allowed 6 h for adhering to surfaces before transfection. U2OS cells were transiently transfected using Lipofectamine 2000 (Invitrogen, Fischer Scientific) by adapting the manufacturer protocol to our system. Briefly, 2 μL Lipofectamine 2000 was added to 50 μL Opti-MEM I Reduced Serum Media (Prod. 11058021, Gibco , Fischer Scientific) and incubated for 5 min at room temperature. Plasmid DNA coding for fluorescent LifeAct-TagGFP2 and TagRFP-vinculin fusion proteins were co-transfected by using 0.5 μg plasmid DNA (vinculin-pTagRFP and pCMVLifeAct plasmids) was diluted in 50 μL Opti-MEM I and incubated at room temperature for 5 min. For co-transfection of TagRFP-vinculin and pCMVLifeAct 0.5 μg of each plasmid was used.
The diluted DNA was added to the diluted Lipofectamine 2000 in a 1:1 ratio, and left to incubate for 20 min at room temperature. 40 μL of the combined transfection complex was then added to each well. After 18 h, 1.5 mL DMEM (Prod. 41965039) supplemented with 10% FBS and 1% 10000U/mL Penicillin-Streptomycin was added to each dish.
For reverse transfection experiments, the same amounts of reactants were used, but the transfection complex was added to a suspension of U2OS cells, and the suspension was then added to the wells.
Microscopy
Live cell imaging was performed usin g a Zeiss LSM 800 Airyscan with an inverted Axio Observer Z1 stand connect to a PeCon compact incubator. Imaging was performed in an humidified environment at 37 °C, with 5% CO2 flow. High resolution imaging was performed using a Zeiss Plan-Apochromat 63x/1.4NA DIC M27 oil objective with Cargille Immersion Oil Type 37 (n =1.51) suited for use at 37 °C. All images were taken using the system optimised pixel size both in-plane (typically 34 nm) and for stacks in the vertical axis (typically 180 nm).
To minimise imaging bias, imaging was performed in a standardised manner where each pillar array was raster scanned and cells expressing both LifeActGFP and Vinculin RFP were imaged. The high resolution images were then processed using a Zeiss algorithm for reconstruction of AiryScan images and exported as CZI-files for further manual and automatised image processing.
Image Analysis
For all cells, cell shape was based on the expression LifeActGFP fusion protein and expression of TagRFP-vinculin was used to identify FAs. Segmentation of images was performed using a script written in Python 3 [72] using CZIfile [73] (version 2017.09.12) for reading the microscopy images in Zeiss-format. The python packages Scipy [74] and Scikit-image [75] were used for multi-dimensional image processing and image segmentation respectively.
To reduce the influence from fluorescence cross-talk from pillars (due to Oxazine 170 perchlorate, Rhodamine 800 or Coumarin 102), the pillar/surface channel was used as a background and subtracted from the TagRFP-vinculin imaging channel. A median filter (size:10 pixels) was applied to remove noise from the TagRFP-vinculin channel, followed by classification of the image into regions based on their intensity value using a Multi-Otsu approach. Multi-Otsu thresholding with three classes was applied. The first class was typically the background, the second class constituted the cytosolic vinculin, whereas vinculin rich areas in FAs appeared brighter and could be classified into a third class. The quality of the image segmentation was briefly assessed by comparison to manual segmentation.
Area of cells and vinculin rich regions were described by counting pixel numbers and from this the actual area was found by correcting for the pixel size. Shape geometries were described by fitting each region with an ellipse with the same second-moment as the segmented region. In order to describe the cell area geometry, three measures were used:(1) Aspect ratio defined as the ratio of the ellipse major axis to the minor axis. (2) Circularity given as,
$$\begin{aligned} C =\frac{4\pi *\text{Area}}{\text{Perimeter}^2}, \end{aligned}$$ (1)and roundness given as,
$$\begin{aligned} R =\frac{4*\text{Area}}{\pi *\text{MajorAxis}^2}. \end{aligned}$$ (2)Segmented vinculin areas with a fitted ellipse that were too round (aspect ratio \(\le 1.5\)) or too elongated (aspect ratio \(\ge 8.5\)) were rejected. In addition, vinculin areas smaller than 0.05 μm 2 were filtered out. In order to find the distance between each vinculin area and the cell edge, the shortest euclidean distance between each centroid (the centre of the fitted ellipse for each vinculin area) and the cell edge was calculated.
Statistical Analysis
Statistical comparisons of distributions were performed by using the non-parametric two-tailed Mann-Whitney test neither assuming normal distribution nor equal standard deviation. P -values \(\ge {0.05}\) were considered to represent a non-significant (ns) difference between the two populations. Significant values were denoted with * for p in 0.01 to 0.05, ** for p in 0.001 to 0.01, *** for p in 0.0001 to 0.001 and lastly **** for p \(\le {0.0001}\).
Abréviations
- DMEM:
-
Dulbecco’s Modified Eagle’s Medium
- EBL:
-
Electron Beam Lithography
- ECM:
-
Extracellular Matrix
- EDTA:
-
Ethylenediaminetetraacetic Acid
- FA:
-
Focal Adhesion
- FBS:
-
Fetal Bovine Serum
- GFP:
-
Green Fluorescent Protein
- HMDS:
-
Hexamethyldisilazane
- NP:
-
Nanopillar
- RFP:
-
Red Fluorescent Protein
- SEM :
-
Scanning Electron Microscopy
Nanomatériaux
- Exemples de circuits et de listes d'interconnexions
- Piles spéciales
- Cellule solaire
- Composites de graphène et polymères pour applications de supercondensateurs :une revue
- Étude in vitro de l'influence des nanoparticules Au sur les lignées cellulaires HT29 et SPEV
- Synthèse de nanocristaux de ZnO et application dans des cellules solaires polymères inversées
- Étude de la force d'adhérence et de la transition vitreuse de films minces de polystyrène par microscopie à force atomique
- Promotion de la croissance cellulaire SH-SY5Y par des nanoparticules d'or modifiées avec de la 6-mercaptopurine et un peptide pénétrant dans les neurones
- Influence de la rigidité élastique et de l'adhérence de surface sur le rebond des nanoparticules



