Le microARN-326 exosomal dérivé de macrophages M1 supprime la progression des cellules du carcinome hépatocellulaire via la voie de signalisation NF-κB
Résumé
L'accumulation de preuves a montré que le microARN (miR) dérivé des exosomes dérivés des macrophages M1 peut réguler la progression du carcinome hépatocellulaire (CHC). Cependant, l'effet de miR-326 dérivé d'exosomes dérivés de macrophages M1 sur le CHC n'a pas été signalé. Par conséquent, l'objectif de la présente étude était d'explorer le mécanisme de miR-326 exosomal des macrophages M1 dans la régulation de la progression des cellules HCC. La RT-qPCR a détecté l'expression de miR-326 dans les lignées cellulaires HCC. L'expression de miR-326 dans le HCC a été altérée par la transfection, et l'effet de miR-326 sur l'expression de CD206 et de NF-κB, la prolifération cellulaire, la formation de colonies, la migration, l'apoptose et l'invasion a été détecté. Par la suite, des exosomes ont été isolés des macrophages M1. La RT-qPCR a identifié l'expression de miR-326 dans les exosomes dérivés des macrophages M1. L'expression de miR-326 dans les exosomes dérivés des macrophages M1 a été modifiée par transfection. Des exosomes dérivés des macrophages M1 ont été co-cultivés avec des cellules HCC pour déterminer leurs effets sur le progrès biologique des cellules HCC. Enfin, des expériences in vivo ont été réalisées pour vérifier les résultats in vitro. MiR-326 a été diminué dans les cellules HCC et enrichi en exosomes dérivés des macrophages M1. La régulation à la hausse de miR-326 inhiberait la prolifération des cellules HCC, la formation de colonies, la migration, l'invasion et l'expression de CD206 et NF-κB et favoriserait l'apoptose, et inhiberait la croissance des tumeurs HCC in vivo , tandis que la régulation à la baisse du miR-326 a montré des effets opposés. Les exosomes dérivés des macrophages M1 ont inhibé la prolifération des cellules HCC, la formation de colonies, la migration, l'invasion et l'expression de CD206 et NF-κB et ont amélioré l'apoptose, tandis que la surexpression de miR-326 a amélioré l'effet des exosomes dérivés des macrophages M1 sur les cellules HCC. Il est révélé que le miR-326 exosomal dérivé des macrophages M1 supprime la prolifération, la migration et l'invasion ainsi que l'apoptose du CHC grâce à la régulation négative de l'expression de NF-κB.
Introduction
Le carcinome hépatocellulaire (CHC) est le cinquième cancer le plus fréquent dans le monde et le cancer primitif du foie le plus fréquent [1]. Manifesté par les données du Registre national du cancer de Chine, le taux de mortalité par cancer primitif du foie est classé troisième, tandis que l'incidence est la quatrième parmi les tumeurs malignes courantes [2]. Les principaux facteurs de risque du CHC sont l'infection chronique par le virus de l'hépatite C et le virus de l'hépatite B, les aliments contaminés par les aflatoxines, l'obésité, le tabagisme, la consommation excessive d'alcool et le diabète de type 2 [3]. La chimioembolisation transartérielle est un traitement établi du CHC au stade intermédiaire, qui améliore la survie de la majorité des patients atteints de CHC à un stade intermédiaire ou avancé [4]. Actuellement, le diagnostic du CHC dépend majoritairement des biomarqueurs sériques et des techniques d'imagerie [5]. Le taux de survie à 5 ans lié au CHC n'est que d'environ 60 %, et l'incidence augmente d'année en année ces dernières années [6]. Compte tenu de cela, la recherche d'une cible thérapeutique précise est une priorité dans le traitement du CHC.
Les macrophages sont des cellules effectrices et des régulateurs majeurs du système immunitaire, exercent d'énormes fonctions sur le remodelage et la réparation des tissus et orchestrent des fonctions métaboliques dans presque tous les tissus in vivo [7]. Il est révélé que les macrophages M1 exercent des effets favorisant la tumeur et améliorent la motilité des cellules HCC [8]. L'exosome est une vésicule discoïde d'un diamètre de 40 à 150 nm [9]. Selon Xu et al., les microARN exosomal (miARN) ont des fonctions sur la prolifération, le caractère invasif, les métastases et la résistance aux médicaments du CHC en modulant l'expression des gènes dans les cellules cibles [10]. Une étude a démontré que les exosomes contenant miR-326 pourraient être une cible clinique potentielle dans la sclérose en plaques [11]. Les miARN pourraient agir comme des oncogènes ou des inhibiteurs de tumeurs en modulant l'expression d'un grand nombre de gènes codés par des protéines au moyen de la dégradation de l'ARNm et du blocage de la traduction dans une séquence spécifique [12]. Une étude a montré que miR-326 supprime la croissance des cellules HCC en perturbant la progression du cycle cellulaire ainsi qu'en améliorant l'apoptose, en plus de supprimer l'invasion cellulaire en diminuant le phénotype de transition épithéliale-mésenchymateuse [13]. Une autre étude a rapporté que miR-326 pourrait être une cible thérapeutique potentielle pour le traitement des patients atteints de CHC [14]. Par conséquent, cette présente étude a discuté du mécanisme du miR-326 exosomal régulant l'invasion et la migration des cellules HCC.
Matériaux et méthodes
Déclaration d'éthique
Toutes les expérimentations animales étaient conformes au Guide pour le soin et l'utilisation des animaux de laboratoire par les comités internationaux. Le protocole a été approuvé par le comité institutionnel d'utilisation des soins aux animaux du troisième hôpital de l'université de Jilin.
Induction et identification des macrophages
La lignée cellulaire monocytaire humaine THP-1 (Kunming Institute of Zoology, CAS, Kunming, China) a été cultivée dans du milieu RPMI 1640 (Gibco, CA, USA; Thermo Fisher Scientific, MA, USA) contenant 10 % de sérum bovin foetal inactivé par la chaleur ( FBS). Les cellules THP-1 ont été mises à réagir avec 150 ng/mL de phorbol 12-myristate 13-acétate (PMA; P8139, Sigma-Aldrich, SF, CA, USA) et incubées dans du milieu RPMI pendant 24 h pour obtenir des macrophages M0. Ensuite, la morphologie cellulaire avant et après induction a été observée par coloration de Wright. Les cellules THP-1 et les macrophages induits ont été remis en suspension dans 5 L de PBS, déposés sur la lame de verre, colorés par la solution de colorant de Wright et mélangés avec la solution tampon à 1:2. Colorées pendant 10 min et rincées à l'eau courante, les cellules ont été observées au microscope. De plus, les marqueurs macrophagiques M0 (CD68 et CD206) ont été mesurés par amplification en chaîne par polymérase quantitative à transcription inverse (RT-qPCR). Ensuite, les macrophages ont été induits dans les macrophages M1 par incubation avec 20 ng/mL d'interféron (IFN)-γ (#285-IF; R&D Systems, MN, USA) et 10 pg/mL de LPS (#8630; Sigma-Aldrich) pendant 18 h. Les marqueurs des macrophages M1 (IDO1 et IL-12 p35) ont été examinés par RT-qPCR [15].
Extraction des exosomes
Les exosomes ont été extraits par un kit de séparation d'exosomes (ExoEasy Maxi Kit, Qiagen, Hilden, Allemagne). Le surnageant de macrophages a été amassé dans un tube à centrifuger de 15 ml dans des conditions aseptiques et filtré par un film filtrant de 0,8 M. Le surnageant cellulaire de chaque groupe a été ajouté au tampon XBP (1 : 1) puis centrifugé avec une colonne de centrifugation par affinité membranaire exoEasy à 500g . Les cellules ont été complétées avec 10 ml de tampon XWP et centrifugées avec 3000-5000g . La colonne de centrifugation par affinité membranaire exoEasy a été hachurée avec 400 L de tampon d'élution XE et centrifugée à 500g . Le tampon d'élution a été transféré sur une colonne de centrifugation par affinité membranaire exoEasy et centrifugé à 500g . Le tampon d'élution a été stocké pendant 24 h à 4 °C puis utilisé pour l'identification. Le reste a été conservé à − 80 °C.
Observation TEM et analyse du suivi des nanoparticules (NTA)
Les exosomes obtenus ci-dessus ont été déposés dans le treillis de cuivre à membrane supportée par du carbone et additionnés de 2 % d'acide phosphotungstique. L'échantillon a été observé sous un MET, et l'image optimale a été collectée et analysée.
Les impuretés et particules en PBS ont été éliminées par un filtre microporeux 0,22 µM. Selon la densité de particules des exosomes, le PBS filtré a été dilué à la concentration appropriée et détecté à l'aide d'un détecteur de nanoparticules Nanosight NS300 (Malvern, Westborough, MA, USA).
Après identification, les exosomes dérivés de macrophages transfectés avec l'inhibiteur miR-326 et le contrôle négatif (NC) de l'inhibiteur miR-326, miR-326 mimic et miR-326 mimic NC ont été extraits par le kit de séparation d'exosomes (Invitrogen).
RT-qPCR
L'ARN total a été extrait par le réactif Trizol (Thermo Fisher) et la PCR en temps réel a été réalisée à l'aide du Master Mix PCR SYBR-Green (Roche) et du système de PCR en temps réel ABI 7500 (Life Technologies, Grand Island, NY, USA ). Les séquences d'amorces sont présentées dans le tableau 1. L'analyse quantitative a été réalisée en utilisant la méthode de 2 −△△Ct .
Analyse Western Blot
La protéine totale des cellules et des exosomes a été extraite. La concentration en protéines a été déterminée par un kit d'acide bicinchoninique (BCA) (Boster Biological Technology Co. Ltd., Wuhan, Hubei, Chine). La protéine a été ajoutée à un tampon d'échantillon et bouillie à 95 °C, et chaque puits a été chargé avec 30 µg. La protéine a été séparée par électrophorèse sur gel de polyacrylamide à 10 % (Boster Biological Technology) et électrotransférée sur une membrane en fluorure de polyvinylidène, qui a été suivie d'un scellement dans 5 % d'albumine de sérum bovin (BSA). La membrane a été hachurée avec un anticorps primaire contre CD63 (1 :1000, Developmental Studies Hybridoma Bank, University of Iowa, Ames, IA, USA), CD181 (1 :1000, R&D Systems), GAPDH (1 :2000, Jackson ImmunoResearch Laboratories, PA, USA) et avec l'anticorps secondaire (1 :500, Jackson ImmunoResearch Laboratories) marqué par la peroxydase de raifort. Les images ont été obtenues par le système d'imagerie à balayage par fluorescence infrarouge bicolore Odyssey, et les valeurs de gris des bandes ont été mesurées par le logiciel d'analyse d'images Quantity One.
Culture cellulaire et criblage
La lignée cellulaire hépatique normale HL-7702 et la lignée cellulaire HCC humaine BEL-7404, HepG2, SMMC-7721 et QGY-7703 ont été sélectionnées et cultivées dans Gibco RPMI Media 1640 avec 10 % de sérum bovin foetal (FBS), de la pénicilline (100 U/mL ) et la streptomycine (100 mg/mL). L'expression de MiR-326 a été détectée par RT-qPCR et les lignées cellulaires appropriées ont été criblées.
Étiquetage des exosomes et absorption des exosomes
Les exosomes ont été remis en suspension avec 250 L de diluant C et doucement triturés en évitant d'endommager la membrane de l'exosome. Le colorant PKH67 (1 L, Sigma-Aldrich) a été ajouté à 250 L de Diluant C pour atteindre 500 L et incubé. La solution a été additionnée de 500 L de BSA 1% et incubée à 37°C pendant 1 min. Les exosomes ont été obtenus par centrifugation à 120 000g 4°C pendant 2h. Les exosomes marqués au PKH67 ont été obtenus par centrifugation à 120 000g 4°C pendant 2h. Les exosomes ont été remis en suspension avec 6 ml de milieu RPMI-1640 en évitant la lumière. Ensuite, les exosomes marqués ont été co-cultivés avec des cellules HCC pendant 12 h. Après cela, le milieu de culture a été retiré et lavé avec du PBS pendant 3 fois, 5 min/temps, et les exosomes marqués par fluorescence qui n'étaient pas absorbés en interne par les cellules HCC ont été soigneusement lavés. Les exosomes ont été fixés avec du paraformaldéhyde à 4 % et teints avec du 4'-6-diamidino-2-phénylindole. Après scellement, la distribution de fluorescence a été observée par un microscope confocal laser.
Regroupement et traitement des cellules
Les cellules HepG2 et les cellules SMMC-7721 ont été ensemencées dans la plaque à 12 puits à 0,5–1 × 10 6 cellules/puits. Avec 50 à 60 % de confluence, les cellules ont été transfectées avec de la Lipofectamine 2000 (Invitrogen, Carlsbad, CA). Les cellules HepG2 ont été réparties dans le groupe miR-326-mimic (transfecté avec miR-326 mimic) et le groupe NC-mimic (transfecté avec miR-326 mimic NC). Les cellules SMMC-7721 ont été assignées au groupe inhibiteur miR-326 (transfecté avec l'inhibiteur miR-326) et au groupe inhibiteur NC (transfecté avec l'inhibiteur NC miR-326). miR-326-mimic, miR-326-inhibiteur et leurs NC ont été mélangés avec de la Lipofectamine 2000 pour la transfection. Les cellules HepG2 et les cellules SMMC-7721 sans aucun traitement ont été définies comme groupe blanc. miR-326-mimic, miR-326-inhibitor et leur NC ont été conçus et composés par Guangzhou RibBio Co., Ltd. (Guangzhou, Chine) (tableau 1).
Co-culture d'exosomes dérivés de macrophages M1 avec des cellules HCC
La concentration en protéines de la suspension d'exosomes dérivés des macrophages M1 a été détectée par la méthode BCA, et le volume de suspension d'exosomes correspondant avec 50 g de protéine a été calculé. Les cellules HepG2 et les cellules SMMC-7721 ont été ensemencées dans une plaque de 12 puits à 1 × 10 5 cellules/mL par puits. Les cellules HepG2 ont été réparties en 4 groupes :groupe témoin (cellules HepG2 non co-cultivées avec des exosomes), groupe exosomes (Exo) (cellules HepG2 co-cultivées avec des exosomes dérivés de macrophages M1), groupe Exo-miR-326-mimic (HepG2 cellules co-cultivées avec des exosomes dérivés de macrophages M1 transfectés avec miR-326 mimic), groupe Exo-NC-mimic (cellules HepG2 co-cultivées avec des exosomes dérivés de macrophages M1 transfectés avec miR-326 mimic NC). Les cellules SMMC-7721 ont également été réparties en 4 groupes :groupe blanc (cellules SMMC-7721 non co-cultivées avec des exosomes), groupe Exo (cellules SMMC-7721 co-cultivées avec des exosomes dérivés de macrophages M1), Exo-miR-326- groupe inhibiteur (cellules SMMC-7721 co-cultivées avec des exosomes dérivés de macrophages M1 transfectés avec un inhibiteur miR-326), groupe inhibiteur Exo-NC (cellules SMMC-7721 co-cultivées avec des exosomes dérivés de macrophages M1 transfectés avec miR- 326 inhibiteur NC).
Dosage du bromure de 3-(4,5-diméthylthiazol-2-yl)-2, 5-diphényltétrazolium (MTT)
Les cellules ont été détachées à la trypsine et ensemencées sur plaque 96 puits avec une densité cellulaire de 4 × 10 4 cellules par puits. Le milieu de culture a été abandonné après 12, 24, 36, 48, 60 h de culture respectivement. Incubées avec 500 L de solution de MTT à 0,5 g/L, les cellules ont été ajoutées à 200 L de solution de diméthylsulfoxyde, triturées et écloses. Les valeurs de densité optique (DO, 490 nm) ont été mesurées par un lecteur de microplaques.
Test de formation de colonie
Cultivées pendant 24 h et détachées à la trypsine, les cellules ont été ensemencées dans une petite boîte de 35 mm avec 300 cellules par boîte. La solution était remplacée tous les 3 jours. Après 10 j de culture, les cellules ont été fixées à 40 g/L −1 paraformaldéhyde et teint avec 1 g/L −1 solution de cristal violet et séchée. Le nombre de colonies (plus de 50 cellules) a été calculé au microscope.
Test de Transwell
Cellules (1 × 10 5 ) ont été suspendus avec 200 L de milieu de culture vierge. Les expériences ont été menées conformément aux instructions de la chambre Transwell (Corning Glass Works, Corning, N.Y., USA) (le matrigel était nécessaire pour l'expérience d'invasion, mais pas pour l'expérience de migration). Du RPIM 1640 (10 % FBS, 600 L) a été ajouté dans la chambre inférieure. Les chambres supérieure et inférieure étaient séparées par une membrane Transwell pré-enduite de matrigel (BD Biosciences, Franklin Lakes, NJ, USA). Cultivée pendant 24 h, la chambre a été fixée avec de l'alcool à 95 %. Après coloration avec une solution de cristal violet, les cellules ont été observées dans cinq champs visuels au microscope.
Cytométrie en flux
Cycle cellulaire :les cellules ont été détachées par la trypsine. Cellules (1 × 10 6 ) ont été mis en suspension avec 0,5 ml de PBS et triturés en une seule suspension. Mélangées avec 4,5 ml d'éthanol à 70% pré-refroidi sur glace, les cellules ont été centrifugées à 3000g , rincé avec 5 mL de PBS et centrifugé à nouveau à 3000g . Ensuite, les cellules ont été mises en suspension avec 1 ml de solution de coloration PI/Triton X-100 (20 pg de PI/0,1 % de Triton X-100) contenant 0,2 mg de RnaseA. Le cycle cellulaire a été détecté par cytométrie en flux.
Apoptose cellulaire :Cellules trypsinisées (1 × 10 6 ) ont été mis en suspension avec 1 mL de PBS, trituré et centrifugé à 3000g . Les cellules ont été rincées tour à tour avec du tampon d'incubation (10 mmol/L Hepes/NaOH, pH 7,4, 140 mmol/L NaCl, 5 mmol/L CaCl2 ) et centrifugé à 3000g . Ensuite, les cellules ont été incubées avec 100 L de solution de marquage (FITC-Annexin V et PI ont été ajoutés au tampon d'incubation pour atteindre 1 g/mL), centrifugés à 3000g , lavé une fois avec du tampon d'incubation et éclos avec une solution fluorescente (SA-FLOUS). L'apoptose cellulaire a été détectée par cytométrie en flux. La longueur d'onde de la cytométrie en flux était de 488 nm, et la fluorescence FITC a été détectée par filtre à bande à 515 nm, tandis que PI avec une longueur d'onde supérieure à 560 nm. Les résultats ont été analysés automatiquement par ordinateur.
Xénogreffe tumorale chez des souris nues
Quarante souris (College of Animal Science, Jilin University, Jilin, Chine) âgées de 4 à 6 semaines ont été réparties au hasard dans 8 groupes, avec 5 souris dans chaque groupe. Les souris ont été nourries dans un laboratoire animalier spécifique sans agent pathogène pendant 1 semaine, et la nourriture, le coussin et la bouteille d'eau ont été remplacés à temps. L'état de santé des souris doit être observé tous les jours. Une semaine plus tard, les cellules HCC ont été préparées en suspension cellulaire et injectées par voie sous-cutanée dans le cou et le dos à 0,1 ml de suspension cellulaire (1 × 10 6 ). La croissance de la tumeur a été observée après 3 à 5 jours. Les souris nudes ont été pesées tous les 4 jours et le volume tumoral a été mesuré. Les souris nude ont été euthanasiées 20 jours après l'injection.
Analyse statistique
Toutes les données ont été interprétées par le logiciel SPSS 17.0 (SPSS Statistics, Chicago, IL, USA). Les données de mesure ont été indiquées en tant que moyenne ± écart type. Les comparaisons entre deux groupes ont été formulées par le t -test, tandis que les comparaisons entre plusieurs groupes ont été évaluées par une analyse de variance à un facteur (ANOVA). P value < 0.05 était indicatif d'une différence statistiquement significative.
Résultats
Identification du macrophage M1 et des exosomes
La coloration de Wright a été utilisée pour observer la morphologie des cellules THP-1 induites par le PMA. Il a été illustré que le volume de cellules THP-1 avant l'induction était faible et la proportion de caryoplasme était plus élevée; la morphologie des cellules après induction était irrégulière, le volume est devenu plus grand et la proportion de caryoplasme a diminué; le cytoplasme était plus riche et de couleur bleu clair, avec des particules abondantes et quelques vacuoles; le noyau était rouge violacé et souvent incliné d'un côté, montrant que les cellules avaient les caractéristiques morphologiques typiques des macrophages (Fig. 1a).
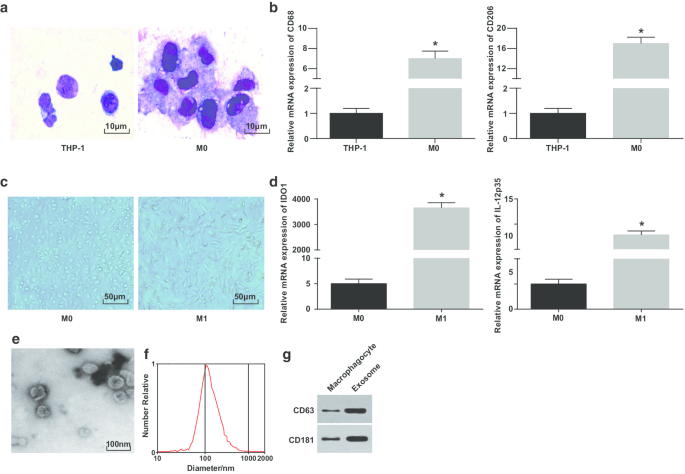
Identification du macrophage M1 et des exosomes. un Coloration de Wright pour l'observation de la morphologie des cellules THP-1 avant et après traitement au PMA. b Expression de CD68 et CD206 dans les cellules THP-1 et les cellules THP-1 traitées au PMA détectées par RT-qPCR ; c morphologie des macrophages et des macrophages M1. d Expression de IDO1 et IL-12 p35 dans les macrophages M0 et les macrophages M0 traités au LPS et INF-γ détectés par RT-qPCR. e MET pour l'observation des exosomes. f Détection de la distribution granulométrique des exosomes par NTA. g Bandes protéiques de CD63 et CD181. Dans le panneau b, *P <0,05 par rapport aux cellules THP-1 ; Dans le panneau d, *P < 0,05 par rapport aux macrophages M0. Les données de mesure ont été représentées sous forme de moyenne ± écart-type (N = 3), les comparaisons entre deux groupes ont été réalisées par test t
Pour vérifier davantage l'induction réussie des macrophages, l'expression de CD68 et CD206 avant et après l'induction a été testée par RT-qPCR. Il s'est avéré que l'expression de CD68 et CD206 était élevée depuis l'induction du PMA, indiquant que le PMA a induit avec succès les cellules THP-1 dans les macrophages M0 (Fig. 1b). Ensuite, les macrophages M0 ont été polarisés en macrophages M1 par induction de LPS et d'INF-γ. La morphologie et l'expression du marqueur de surface des macrophages de type M1 IDO1 et IL-12 p35 des macrophages ont été observées et testées. Les macrophages M0 présentaient des morphologies adhérentes diverses et irrégulières, de forme ronde, elliptique ou fusiforme. La morphologie des macrophages stimulés par l'IFN-γ a été démontrée avec plus de pseudopodes et de protubérances et de fusiformes (Fig. 1c). La RT-qPCR a montré (Fig. 1d) qu'après traitement au LPS et INF-γ, les macrophages M1 ont montré une augmentation de leurs marqueurs (IDO1 et IL-12p35).
Par la suite, les exosomes dérivés des macrophages ont été observés par le MET. Il a été révélé que les exosomes dérivés des macrophages étaient riches et de forme ronde ou ovale, avec une structure membraneuse, une taille uniforme et moins de polluants (Fig. 1e). NTA a montré que les exosomes avec un pic centralisé de courbe MODE et linéaire lisse avaient un diamètre plus concentré et moins d'impuretés (Fig. 1f). L'analyse par transfert Western a rapporté que par rapport aux macrophages, l'expression des protéines marqueurs spécifiques CD63 et CD181 s'est intensifiée dans les exosomes dérivés des macrophages (Fig. 1g). Ces résultats indiquent que nous avons réussi à induire les monocytes à se différencier en macrophages et à les polariser en macrophages M1.
Les exosomes dérivés des macrophages M1 délivrent miR-326 aux cellules HCC et affectent l'expression de miR-326 dans les cellules HCC
Pour vérifier si les exosomes dérivés des macrophages M1 transportaient miR-326 vers les cellules HCC, des cellules HepG2 et SMMC-7721 ont été co-cultivées avec des exosomes. Il a pu être reconnu qu'un grand nombre d'exosomes ont été assimilés par les cellules HepG2 et SMMC-7721 lors d'une transfection de 4 h sous le microscope à fluorescence (Fig. 2a, b).
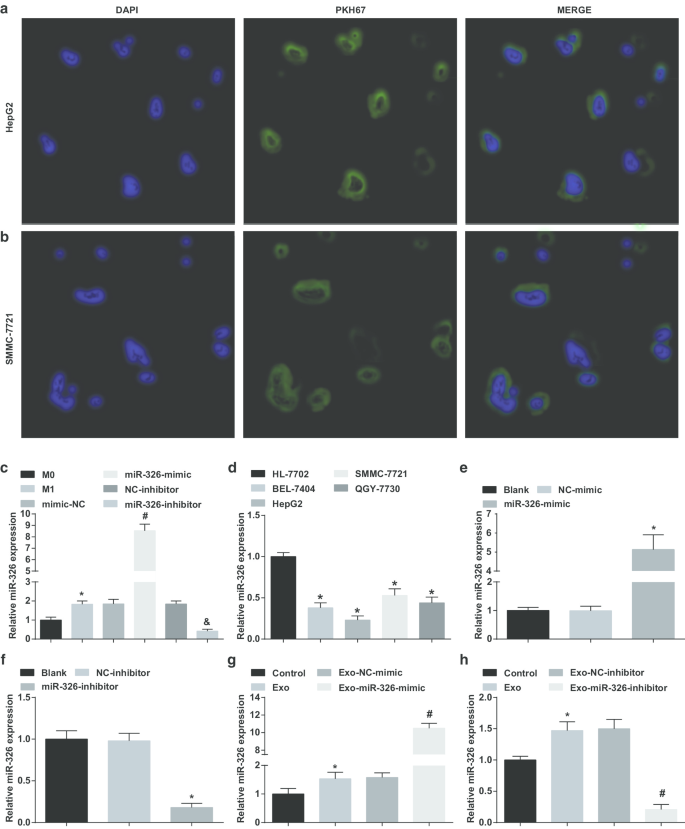
Les exosomes dérivés des macrophages M1 délivrent miR-326 aux cellules HCC et affectent l'expression de miR-326 dans les cellules HCC. un Absorption des exosomes dérivés des macrophages M1 par les cellules HepG2. b Absorption des exosomes dérivés des macrophages M1 par les cellules SMMC-7721. c Comparaison de l'expression de miR-326 des exosomes de macrophages dans chaque groupe avant et après l'induction d'INF-γ et de LPS. d Expression de miR-326 dans les lignées cellulaires HCC (BEL-7404, HepG2, SMMC-7721, QGY-7703) et la lignée cellulaire d'hépatocytes humains normaux HL-7702 détectés par RT-qPCR. e La RT-qPCR a détecté l'effet de miR-326 mimic sur l'expression de miR-326 dans les cellules HepG2. f La RT-qPCR a détecté l'effet de l'inhibiteur de miR-326 sur l'expression de miR-326 dans les cellules SMMC-7721. g La RT-qPCR a détecté les effets des exosomes dérivés des macrophages M1 transfectés miR-326 sur l'expression de miR-326 dans les cellules HepG2. h La RT-qPCR a détecté les effets des exosomes dérivés des macrophages M1 transfectés par un inhibiteur de miR-326 sur l'expression de miR-326 dans les cellules SMMC-7721. Dans le panneau c, *P <0,05 versus le macrophage M0; Dans le panneau d, *P <0,05 par rapport aux cellules HL-7702 ; Dans le panneau e, *P <0,05 par rapport au groupe NC-mimic ; Dans le panneau f, *P <0,05 versus le groupe NC-inhibiteur ; Dans le panneau g, *P <0,05 versus le groupe témoin, #P <0,05 versus le groupe Exo-NC-mimic ; Dans le panneau h, *P <0,05 versus le groupe témoin, #P <0,05 versus le groupe Exo-NC-inhibiteur. Les données de mesure ont été représentées sous forme de moyenne ± écart-type (N = 3), les comparaisons entre plusieurs groupes ont été effectuées par une analyse de variance à un facteur
Par la suite, miR-326-mimic/inhibitor a été transfecté dans des macrophages et vérifié l'expression de miR-326 dans leurs exosomes avant et après la transfection. Il a été montré que par rapport aux exosomes des macrophages M0, l'expression de miR-326 était augmentée dans les exosomes dérivés des macrophages M1. Une expression élevée de miR-326 a été observée dans les exosomes dérivés des macrophages M1 avec un traitement miR-326-mimic. Expression de MiR-326 réduite dans les exosomes dérivés des macrophages M1 avec un traitement par inhibiteur de miR-326 (Fig. 2c).
Ensuite, l'expression de miR-326 dans des lignées cellulaires HCC a été testée. Comme cela s'est manifesté, l'expression de miR-326 a diminué dans les cellules BEL-7404, HepG2, SMMC-7721 et QGY-7703 par rapport aux cellules HL-7702, parmi lesquelles les cellules HepG2 se sont manifestées avec l'expression la plus faible, tandis que les cellules SMMC-7721 avec l'expression la plus élevée (Fig. 2d).
Ensuite, miR-326-mimic et miR-326-inhibitor ont été transfectés dans des cellules HepG2 et SSMC-7721, respectivement, et ont examiné leurs effets sur l'expression de miR-326. MiR-326 imitent l'expression élevée de miR-326 dans les cellules HepG2, tandis que l'inhibiteur de miR-326 a réduit l'expression de miR-326 dans les cellules SSMC-7721 (Fig. 2e, f).
Ensuite, les exosomes des macrophages M1 transfectés avec miR-326-mimic et miR-326-inhibiteur ont été co-cultivés avec des cellules HepG2 et SSMC-7721, respectivement. Il a été mis en évidence que la co-culture avec des exosomes augmentait l'expression de miR-326 dans les cellules HCC, les exosomes dérivés des macrophages M1 transfectés miR-326 ont encore augmenté l'expression de miR-326 dans les cellules HepG2, tandis que les macrophages M1 transfectés par l'inhibiteur miR-326 -Les exosomes dérivés ont dégradé l'expression de miR-326 dans les cellules SSMC-7721 (Fig. 2g, h). Il est suggéré que les exosomes dérivés des macrophages M1 délivrent miR-326 aux cellules HCC et affectent l'expression de miR-326 dans les cellules HCC.
Le miR-326 exosomal dérivé de macrophages M1 réduit la prolifération cellulaire et la capacité de formation de colonies dans les cellules HCC
En explorant l'effet du miR-326 exosomal sur la prolifération des cellules HCC, des tests de formation de MTT et de colonies ont été effectués pour examiner la prolifération des cellules HCC. Il a été suggéré que dans les cellules HepG2, la restauration de miR-326 altère la prolifération cellulaire et la capacité de formation de colonies (Fig. 3a, e, f). Les exosomes dérivés des macrophages M1 ont empêché les cellules HepG2 de proliférer et de former des colonies. MiR-326 imitant les exosomes dérivés des macrophages M1 transfectés a encore altéré la capacité de prolifération cellulaire et de formation de colonies (Fig. 3c, i, j).
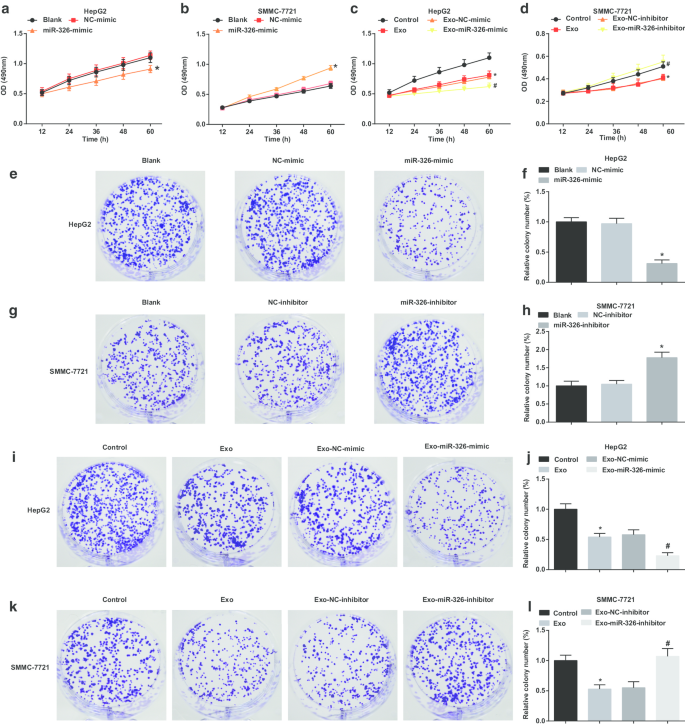
M1 macrophage exosomal miR-326 inhibe la prolifération cellulaire et la formation de colonies dans les cellules HCC. un Le test MTT a détecté l'effet de la transfection de miR-326 mimic sur la prolifération des cellules HepG2. b Le test MTT a détecté l'effet de la transfection de l'inhibiteur de miR-326 sur la prolifération des cellules SMMC-7721. c Le test MTT a détecté l'effet de la co-culture avec des exosomes dérivés de macrophages M1 transfectés miR-326 sur la prolifération des cellules HepG2. d Le test MTT a détecté l'effet de la co-culture avec des exosomes dérivés de macrophages M1 transfectés par un inhibiteur miR-326 sur la prolifération des cellules SMMC-7721. e Le test de formation de colonies a détecté l'effet de la transfection de miR-326 mimic sur la capacité de formation de colonies de cellules HepG2. f Nombres de colonies de cellules HepG2. g Le test de formation de colonies a détecté l'effet de la transfection de l'inhibiteur de miR-326 sur la capacité de formation de colonies des cellules SMMC-7721. h Nombres de colonies de cellules SMMC-7721. je Le test de formation de colonies a détecté l'effet de la co-culture avec des exosomes dérivés de macrophages M1 transfectés miR-326 sur la capacité de formation de colonies de cellules HepG2. j Nombre de colonies de cellules HepG2 traitées avec des exosomes. k Le test de formation de colonies a détecté l'effet de la co-culture d'exosomes dérivés de macrophages M1 transfectés par un inhibiteur de miR-326 sur la capacité de formation de colonies de cellules SMMC-7721. l Nombres de colonies de cellules SMMC-7721 traitées avec des exosomes. Dans les panneaux a et f, *P <0,05 par rapport au groupe NC-mimic ; Dans les panneaux b et h, *P <0,05 versus le groupe NC-inhibiteur ; Dans les panneaux c et j, *P <0,05 versus le groupe témoin, #P <0,05 versus le groupe Exo-NC-mimic ; Dans les panneaux d et l, *P <0,05 versus le groupe témoin, #P <0,05 versus le groupe Exo-NC-inhibiteur. Les données de mesure ont été représentées sous forme de moyenne ± écart-type (N = 3), les comparaisons entre plusieurs groupes ont été effectuées par une analyse de variance à un facteur
Dans les cellules SMMC-7721, le knockdown de miR-326 a amélioré la prolifération cellulaire et la capacité de formation de colonies (Fig. 3b, g, h). Dans les cellules SMMC-7721 traitées avec des exosomes dérivés des macrophages M1, la prolifération cellulaire et la capacité de formation de colonies ont diminué. Les exosomes dérivés des macrophages M1 transfectés par l'inhibiteur MiR-326 ont davantage encouragé la prolifération cellulaire et la capacité de formation de colonies (Fig. 3d, k, l). Il est suggéré que miR-326 dérivé d'exosomes dérivés de macrophages M1 empêche la prolifération des cellules HCC.
Le miR-326 exosomal dérivé des macrophages M1 supprime la migration et l'invasion des cellules HCC
Ensuite, l'effet de miR-326 exosomal sur l'invasion et la migration des cellules HCC a été examiné. Il a été démontré que dans les cellules HepG2, la restauration de miR-326 limitait l'invasion et la migration (Fig. 4a–c). Les exosomes dérivés des macrophages M1 ont perturbé les cellules HepG2 pour envahir et migrer. L'invasion et la migration se sont encore dégradées lorsque des cellules HepG2 ont été co-cultivées avec des exosomes dérivés de macrophages M1 transfectés miR-326 (Fig. 4g-i).
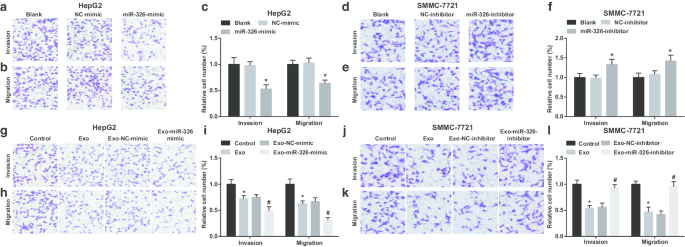
M1 macrophage exosomal miR-326 inhibe la migration et l'invasion des cellules HCC. un Le test Transwell a détecté l'effet de la transfection de miR-326 sur l'invasion des cellules HepG2. b Le test Transwell a détecté l'effet de la transfection de miR-326 imitant la migration des cellules HepG2. c Le nombre d'invasion et de migration des cellules HepG2. d Le test Transwell a détecté l'effet de la transfection de l'inhibiteur de miR-326 sur l'invasion des cellules SMMC-7721. e Le test Transwell a détecté l'effet de la transfection de l'inhibiteur de miR-326 sur la migration des cellules SMMC-7721. f Le nombre d'invasion et de migration des cellules SMMC-7721. g Le test Transwell a détecté l'effet de la co-culture avec des exosomes dérivés de macrophages M1 transfectés miR-326 sur l'invasion des cellules HepG2. h Le test Transwell a détecté l'effet de la co-culture avec des exosomes dérivés de macrophages M1 transfectés miR-326 sur la migration des cellules HepG2. je Le nombre d'invasion et de migration de cellules HepG2 co-cultivées avec des exosomes dérivés de macrophages M1. j Le test Transwell a détecté l'effet de la co-culture avec des exosomes dérivés de macrophages M1 transfectés par un inhibiteur miR-326 sur l'invasion des cellules SMMC-7721. k Le test Transwell a détecté l'effet de la co-culture avec des exosomes dérivés de macrophages M1 transfectés par un inhibiteur miR-326 sur la migration des cellules SMMC-7721. l Le nombre d'invasion et de migration de cellules SMMC-7721 co-cultivées avec des exosomes dérivés de macrophages M1. Dans le panneau c, *P <0,05 par rapport au groupe NC-mimic ; Dans le panneau f, *P <0,05 versus le groupe NC-inhibiteur ; Dans le panneau i, *P <0,05 versus le groupe témoin, #P <0,05 versus le groupe Exo-NC-mimic ; Dans le panneau l, *P <0,05 versus le groupe témoin, #P <0,05 versus le groupe Exo-NC-inhibiteur. Les données de mesure ont été représentées sous forme de moyenne ± écart-type (N = 3), les comparaisons entre plusieurs groupes ont été effectuées par une analyse de variance à un facteur
MiR-326 knockdown a entraîné des améliorations dans l'invasion et la migration des cellules SMMC-7721 (Fig. 4d-f). Lorsqu'elles sont traitées avec des exosomes dérivés des macrophages M1, les cellules SMMC-7721 ont présenté une diminution de l'invasion et de la migration. However, SMMC-7721 cell invasion and migration were boosted upon co-culture with miR-326-inhibitor-transfected M1 macrophages-derived exosomes (Fig. 4j–l). It is implied that miR-326 derived from M1 macrophage exosomes impedes the invasion and migration of HCC cells.
M1 Macrophage-Derived Exosomal miR-326 Promotes Apoptosis of HCC Cells
When examining the effect of exosomal miR-326 on the cell cycle and apoptosis of HCC cells, PI single staining and Annexin V-FITC/PI double staining were applied. It was illustrated that miR-326 overexpression increased cells arrested at G0/G1 phase, reduced cells arrested at S and G2/M phases and raised apoptosis in HepG2 cells (Fig. 5a–d). Co-culturing with M1 macrophages-derived exosomes increased cells arrested at G0/G1 phase, reduced cells arrested at S and G2/M phases and raised cell apoptosis of HepG2 cells. Co-cultivation with exosomes from M1 macrophages transfected with miR-326-mimic further enhanced these effects (Fig. 5i–l).
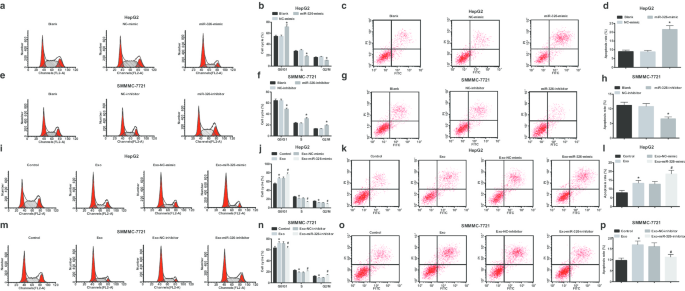
M1 macrophage exosomal miR-326 promotes apoptosis of HCC cells. un , b Flow cytometry detected the effect of transfection of miR-326 mimic on HepG2 cell cycle; c , d flow cytometry detected the effect of transfection of miR-326 mimic on HepG2 cell apoptosis; e , f flow cytometry detected the effect of transfection of miR-326 inhibitor on SMMC-7721 cell cycle; g , h flow cytometry detected the effect of transfection of miR-326 inhibitor on SMMC-7721 cell apoptosis; je , j flow cytometry detected the effects of co-culture of with miR-326 mimic-transfected M1 macrophage-derived exosomes on HepG2 cell cycle; k , l flow cytometry detected the effect of co-culture with miR-326 mimic-transfected M1 macrophage-derived exosomes on HepG2 cell apoptosis; m , n flow cytometry detected the effect of co-culture with miR-326 inhibitor-transfected M1 macrophage-derived exosomes on SMMC-7721 cell cycle; o , p , flow cytometry detected the effect of co-culture with miR-326 inhibitor-transfected M1 macrophage-derived exosomes on SMMC-7721 cell apoptosis. In panel b and d, *P <0.05 versus the NC-mimic group; In panel f and h, *P <0.05 versus the NC-inhibitor group; In panel j and l, *P <0.05 versus the control group, #P <0.05 versus the Exo-NC-mimic group; In panel n and p, *P <0.05 versus the controlgroup, #P <0.05 versus the Exo-NC-inhibitor group. Measurement data were depicted as mean ± standard deviation (N = 3), comparisons among multiple groups were conducted by one-way analysis of variance
In SMMC-7721 cells, miR-326 down-regulation reduced cells arrested at G0/G1 phase, elevated cells arrested at S and G2/M phases, and declined cell apoptosis (Fig. 5e–h). Untransfected M1 macrophages-derived exosomes increased cells arrested at G0/G1 phase, reduced cells arrested at S and G2/M phases, and heightened cell apoptosis. MiR-326-inhibitor-transfected M1 macrophages-derived exosomes degraded cells arrested at G0/G1 phase, elevated cells arrested at S and G2/M phases, and decreased cell apoptosis (Fig. 5m–p). Briefly, it is summarized that miR-326 derived from M1 macrophage exosomes arrests cell cycle in G0/G1 phase and induces cell apoptosis in HCC.
M1 Macrophage-Derived Exosomal miR-326 Declines CD206 and NF-κB Expression in HCC Cells
Next, the potential mechanism of miR-326 derived from M1 macrophage exosomes in the biological progress of HCC cells was explored. NF-κB is the key link between inflammation and cancer. Many regulatory proteins and miRNAs could inhibit the excessively activated NF-κB signaling to suppress cancer [16]. Such a beneficial effect may include the polarization of M2 macrophages into M1 macrophages. CD206 and NF-κB expression in HepG2 and SMMC-7721 cells was tested by RT-qPCR. It was suggested that miR-326 restoration decreased CD206 and NF-κB expression in HepG2 cells, while miR-326 knockdown enhanced CD206 and NF-κB expression in SMMC-7721 cells (Fig. 6a, b). Moreover, co-culture with M1 macrophage exosomes significantly reduced CD206 and NF-κB expression in HepG2 cells, while co-culture with M1 macrophage exosomes-overexpressing miR-326 further decreased CD206 and NF-κB expression. Treated with untransfected M1 macrophages-derived exosomes, CD206 and NF-κB expression was decreased in SMMC-7721 cells. Co-cultured with miR-326-inhibitor-transfected M1 macrophages-derived exosomes, SMMC-7721 cells were featured by heightened CD206 and NF-κB expression (Fig. 6c, d). It was concluded that miR-326 from M1 macrophage exosomes played a tumor suppressor by inhibiting NF-κB in HCC cells.
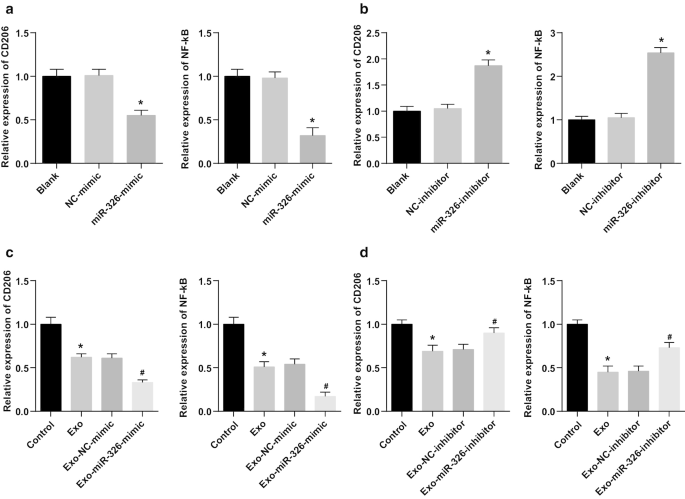
M1 macrophage exosomal miR-326 declines CD206 and NF-κB expression in HCC cells. un RT-qPCR detected the effect of transfection of miR-326 mimic on the expression of CD206 and NF-κB in HepG2 cells; b RT-qPCR detected the effect of transfection of miR-326 inhibitor on the expression of CD206 and NF-κB in SMMC-7721 cells. c RT-qPCR detected the effect of co-culture with miR-326 mimic-transfected M1 macrophage-derived exosomes on the expression of CD206 and NF-κB in HepG2 cells; d RT-qPCR detected the effect of co-culture with miR-326 inhibitor-transfected M1 macrophage-derived exosomes on the expression of CD206 and NF-κB in SMMC-7721 cells. In panel a, *P <0.05 versus the NC-mimic group; In panel b, *P <0.05 versus the NC-inhibitor group; In panel c, *P <0.05 versus the control group, #P <0.05 versus the Exo-NC-mimic group; In panel d, *P <0.05 versus the control group, #P <0.05 versus the Exo-NC-inhibitor group. Measurement data were depicted as mean ± standard deviation (N = 3), comparisons among multiple groups were conducted by one-way analysis of variance
miR-326 from M1 Macrophage Exosomes Inhibits HCC Tumor Growth In Vivo
Finally, the in vivo results were validated through tumor xenografts. As displayed, miR-326 overexpression decreased volume and weight of tumors in HepG2 cells (Fig. 7a–c). In mice transplanted with HepG2 cells co-cultured with exosomes, the treatment with M1 macrophage exosomes significantly reduced the tumor volume and weight of HepG2 cells, while treatment with M1 macrophages-overexpressing miR-326 further reduced the tumor volume and weight of HepG2 cells (Fig. 7g–i).
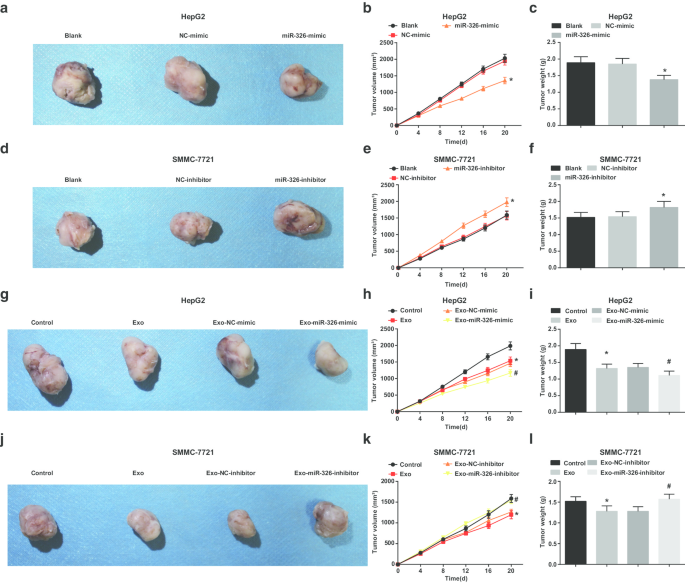
M1 macrophage exosomal miR-326 reduces the volume and weight of HCC tumor in vivo. un –c The effect of transfection of miR-326 mimic on the tumor volume and tumor of nude mice xenografted with HepG2 cells. d –f The effect of transfection of miR-326 inhibitor on the tumor volume and tumors of nude mice xenografted with SMMC-7721 cells. g –je The effect of co-culture with miR-326 mimic-transfected M1 macrophage-derived exosomes on the tumor volume and tumor of nude mice xenografted with HepG2 cells. j –l The effect of co-culture with miR-326 inhibitor-transfected M1 macrophage-derived exosomes on the tumor volume and tumor of nude mice xenografted with SMMC-7721 cells. In panel b and c, *P <0.05 versus the NC-mimic group; In panel e and f, *P <0.05 versus the NC-inhibitor group; In panel h and i, *P <0.05 versus the control group, #P <0.05 versus the Exo-NC-mimic group; In panel k and l, *P <0.05 versus the control group, #P <0.05 versus the Exo-NC-inhibitor group. Measurement data were depicted as mean ± standard deviation (n = 5), comparisons among multiple groups were conducted by one-way analysis of variance
In SMMC-7721 cells, miR-326 suppression increased volume and weight of tumors (Fig. 7d–f). Untransfected M1 macrophages-derived exosomes obstructed tumor growth in volume and weight of SMMC-7721 cells. Co-cultured with miR-326 inhibitor-transfected M1 macrophages-derived exosomes, SMMC-7721 cells were injected into mice and caused elevations in tumor volume and weight (Fig. 7j–l). Summarily, miR-326 derived from M1 macrophage exosomes depressed tumor growth of HCC in vivo.
Discussion
HCC is a common cancer that is characterized with high morbidity and mortality, difficult early diagnosis and treatment, poor prognosis and 5-year survival rate [17]. Recently, a study has highlighted that the lowly expressed lncRNA cox-2 declines the ability of M1 macrophages to suppress HCC cell growth, invasiveness, angiogenesis migration and promote apoptosis [18]. Hu et al. have discussed that miR-326 is obviously degraded in HCC tissues and cell lines, while down-regulated miR-326 is connected to the TNM stage, lymph node metastasis and differentiation of HCC patients [14]. It is customarily considered that HCC cells-derived exosomes can form a fertile environment to facilitate HCC cells growth, invasiveness and metastasis as well as development of drug resistance [19]. The current study was designed to explore the mechanism of exosomal miR-326 in regulating invasion and migration of HCC cells.
Our results indicated that miR-326 expression decreased in HCC cells but increased in exosomes. A recent study has pointed out that miR-326 expression is declined in HCC tissues [20]. Another study has presented miR-326 expression is notably reduced in HCC cell lines and tissues and its down-regulation predicts a poor prognosis in HCC [13]. It is reported that miR-326 acts a tumor-suppression role and is greatly depressed in HCC cells [21]. All these aforementioned evidences are in line with our findings. A study has purported that in comparison with the controls, miR-326 expression is raised in Tconv-derived exosomes which is observed in relapsing–remitting multiple sclerosis patients [11].
Other results emerge from our data that highly expressed exosomal miR-326 reduced cell proliferation, colony formation, migration and invasion as well as facilitated apoptosis of HCC cells in vitro and reduced the volume and weight of HCC tumor in vivo. It has been suggested previously that HCC cell growth can be suppressed via overexpression of miR-326, and HCC cell migration and invasion ability are markedly attenuated through elevating miR-326 [21]. It is reported that the up-regulated miR-326 expression suppresses HCC cell growth and invasiveness as well as stimulates cell apoptosis in vitro [14]. Besides that, a prior study has verified that overexpression of miR-326 declined tumor growth in vivo [13]. A study has revealed that ectopic expressed miR-326 markedly attenuates cell growth, and suppresses cellular migration and invasiveness in non-small cell lung cancer cell lines [22]. Moreover, it is found that miR-326 decreases profibrotic genes like MMP-9, implying its repressive function in cancer cell proliferation [23]. Also, it is presented that miR-326 represses Bcl-2 protein expression and elevates Bax expression so as to affect the apoptosis [24]. Similar to our findings, there are some miRNAs interacting with exosomes to play a role in HCC development. It is displayed that highly expressed exosomal miR-638 can repress the proliferation of HCC cells, involving the potential impact on carcinogenesis [25]. Another study also proves that HCC cells-derived exosomal miR-451a suppresses tumor angiogenesis via disrupting endothelial functions as apoptosis, tube formation, migration and permeability [26]. A prior research generally confirms that when treated with the overexpression of miR-744 exosomes, the proliferation of HCC cells is dramatically suppressed [27].
Conclusion
To briefly conclude, our study provides evidence that M1 macrophage-derived exosomal miR-326 suppresses proliferation, migration and invasion as well as advances apoptosis of HCC cells, supplying a new insight in a novel target therapy for HCC. Due to the limited sample size and limited known researches, the exact mechanism of miR-326 is not fully elucidated, and therefore, further large-scale studies are required to illustrate the underlying mechanism.
Abréviations
- HCC:
-
Hepatocellular carcinoma
- miRNAs:
-
MicroRNAs
- FBS :
-
Sérum fœtal bovin
- M/CSF:
-
Macrophage colony-stimulating factor
- PBS :
-
Solution saline tamponnée au phosphate
- DMEM :
-
Dulbecco’s modified eagle medium
- PPAR:
-
Peroxisome proliferator-activated receptor
- TEM :
-
Microscope électronique à transmission
- NTA:
-
Nanoparticle tracking analysis
- NC :
-
Contrôle négatif
- RT-qPCR :
-
Réaction en chaîne par polymérase quantitative de transcription inverse
- BCA:
-
Bicinchoninic acid
- BSA :
-
Sérum albumine bovine
- Exo:
-
Exosomes
- ANOVA :
-
Analyse unidirectionnelle de la variance
Nanomatériaux
- Contrôle complet de la polarisation térahertz avec bande passante élargie via des métasurfaces diélectriques
- Étude in vitro de l'influence des nanoparticules Au sur les lignées cellulaires HT29 et SPEV
- Effet de l'électrode ITO pulvérisée à angle oblique dans les structures de cellules solaires en pérovskite MAPbI3
- Fabrication et caractérisation d'un nouveau support de catalyseur anodique en nanofibre de carbone composite Tio2 pour pile à combustible au méthanol direct via la méthode d'électrofilage
- Amélioration de l'efficacité antitumorale et de la pharmacocinétique de la bufaline via les liposomes pégylés
- Réglage du mode de mouvement Slide-Roll des nanotubes de carbone via des groupes hydroxyle
- Évaluation de l'activité du cytochrome P450 3A4 inhibée par les nanoparticules d'or et des mécanismes moléculaires sous-jacents à sa toxicité cellulaire dans la lignée cellulaire de carcinome …
- De minuscules nanoparticules de fluorure de terres rares activent la croissance des cellules tumorales via des interactions polaires électriques
- Mécanisme de défaillance de commutation dans une cellule de métallisation programmable à base de peroxyde de zinc



