Réadaptation de médicaments
Inhibition de la protéase principale du SRAS-CoV-2
Le développement d'un nouveau médicament est un processus long et coûteux. Aujourd'hui, alors que le monde est confronté à une pandémie, il est urgent d'identifier rapidement des médicaments qui arrêtent la prolifération du virus. La réorientation des médicaments offre une alternative intéressante à ce long processus en essayant d'identifier si un médicament connu pour être sans danger pour l'homme pourrait être utilisé pour traiter de nouvelles maladies.
Bien que l'utilisation individuelle de ces médicaments repositionnés puisse éventuellement ne pas entraîner d'avantage clinique significatif, une combinaison prudente de médicaments ciblant plusieurs protéines cruciales pour la réplication et la prolifération du virus pourrait être très efficace, comme ce fut le cas avec le VIH dans les années 1990. La question urgente est quelle combinaison serait la plus efficace ?
Ici, nous essayons de comprendre la structure du site actif de la protéase SARS-CoV-2, en la comparant aux structures existantes de la protéase SARS-CoV complexée avec des inhibiteurs micromolaires, afin de mieux comprendre les interactions clés nécessaires pour créer un bon inhibiteur. pour la protéase SARS-CoV-2.
Nous avons ensuite mené une expérience de dépistage virtuel en utilisant une bibliothèque de médicaments approuvés par la FDA pour voir si certains d'entre eux devraient se lier à la protéase. Nous avons examiné comment ils devraient se lier à la protéase du SRAS-CoV-2 et pourraient donc être utilisés dans une thérapie combinée.
Protéines SARS-CoV-2
Le génome du SRAS-CoV-2 de patients malades a été rapidement isolé et séquencé, fournissant les séquences de cibles protéiques possibles. Ces protéines partagent une similitude de séquence élevée avec les protéines du SRAS-CoV et les groupes de recherche ont initialement commencé à construire des modèles d'homologie. Maintenant, nous voyons de plus en plus de ces structures dérivées expérimentalement (rayons X et cryo-EM) devenir disponibles.
L'une des cibles médicamenteuses les mieux caractérisées parmi les coronavirus est la principale protéase :Mpro, également appelée protéase 3CL. 1 Avec les protéases de type papaïne, cette enzyme est essentielle pour traiter les polyprotéines qui sont traduites à partir de l'ARN viral. 2 Il clive le squelette d'acides aminés au niveau de 11 sites de clivage sur la grande polyprotéine 1ab (Fig. 1).
L'inhibition de l'activité de cette enzyme bloquerait la réplication virale. Étant donné qu'aucune protéase humaine avec une spécificité de clivage similaire n'est connue, les inhibiteurs de cette cible sont beaucoup moins susceptibles d'être toxiques et de provoquer des effets secondaires.
Conception de médicaments basée sur la structure pour la réorientation des médicaments :protéase principale
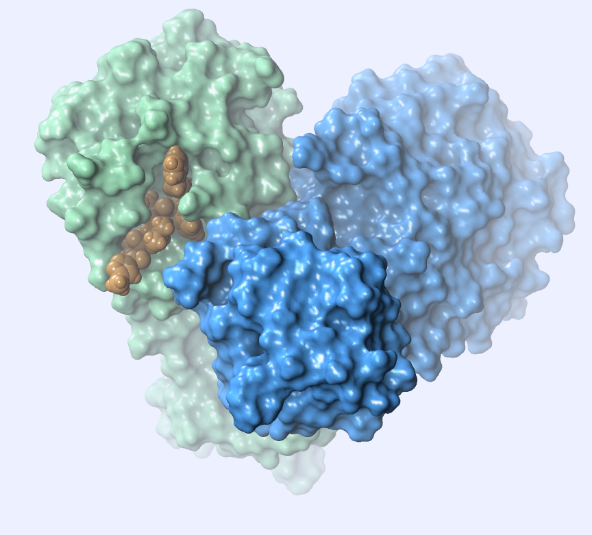
Cette protéase principale est un homodimère (Fig. 2) et chaque sous-unité contient une dyade catalytique His41/Cys145.
Le SARS-CoV-2 partage 96,1 % d'identité et 99 % de similarité avec la protéase du SARS-CoV. (Fig. 3) Il n'y a qu'une seule différence d'acides aminés dans le site actif :le résidu 46 dans le SARS-CoV-2 est une sérine au lieu d'une alanine dans le SARS-CoV.
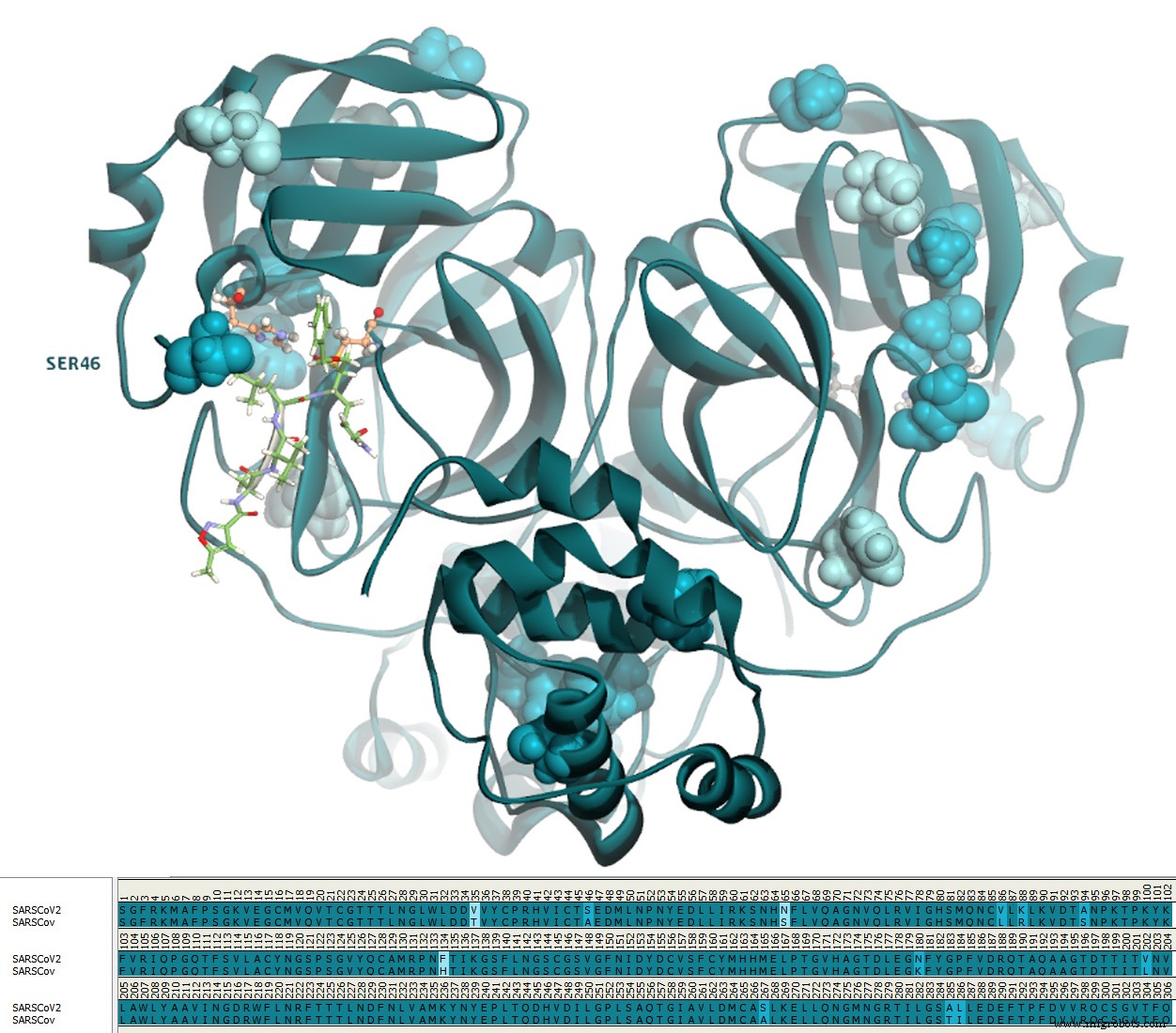
Il existe de nombreuses structures de SARS-CoV Mpro complexées avec des ligands disponibles au PDB et les ligands qui ont une affinité de liaison micromolaire sont tous des ligands covalents.
Les sites actifs de la protéase principale sont hautement conservés parmi les coronavirus et sont généralement composés de quatre sites (S1′, S1, S2 et S4) 3 (Fig.4).
Dans le cas des structures PDB 2ZU4 et 2GX4 pour lesquelles les ligands ont respectivement des valeurs de Ki de 0,038 µM et 0,053 µM, le thiol de la cystéine 145 dans le site S1' contacte les inhibiteurs par une liaison covalente. Par rapport à d'autres inhibiteurs avec une affinité plus faible, cela semble être important pour une affinité plus élevée.
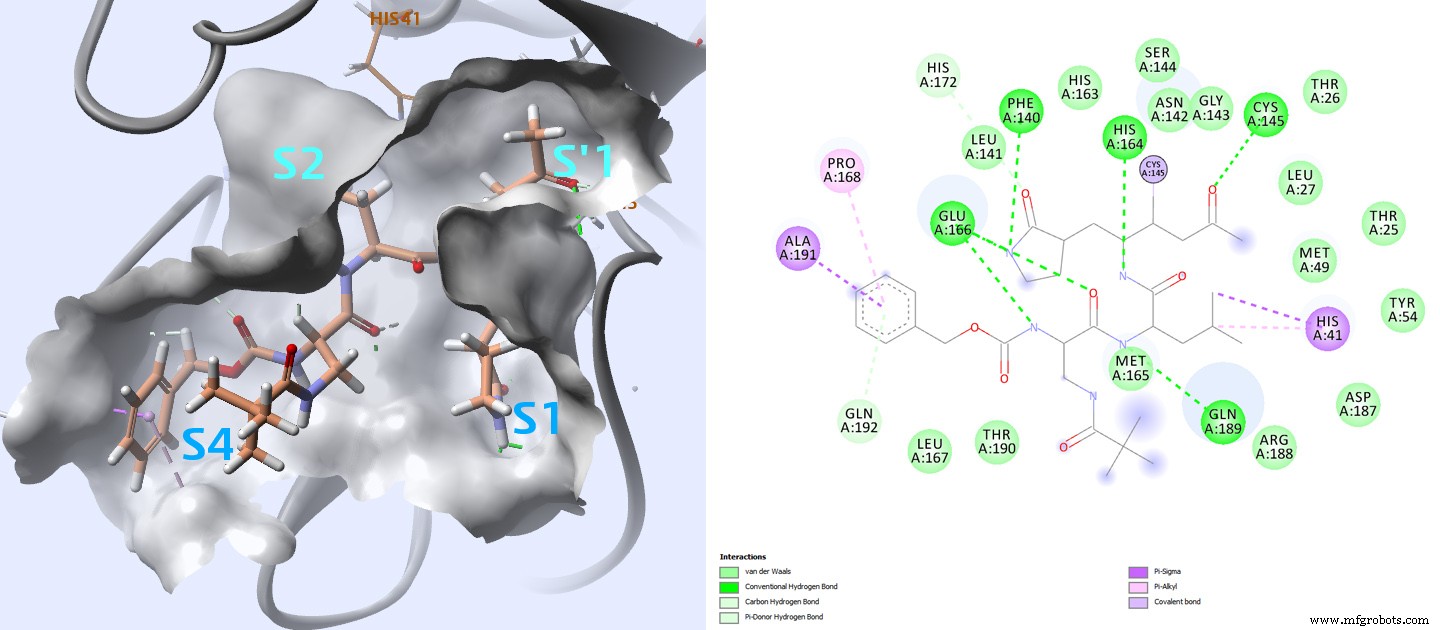
Un examen attentif de la façon dont tous ces ligands interagissent avec cette protéase peut fournir des informations sur les interactions clés à surveiller lors de l'analyse des résultats d'amarrage.
Dépistage virtuel
Tirer parti d'une structure cristalline haute résolution du dimère de protéase SARS-COV-2 en complexe avec un inhibiteur de type peptidique lié de manière covalente N3 publié en février (6LU7) 4 , nous avons utilisé cette structure pour mener une expérience de criblage virtuel. L'inhibiteur N3 qui est co-cristallisé se lie dans la poche de liaison au substrat dans une conformation étendue. (Illustration 5)
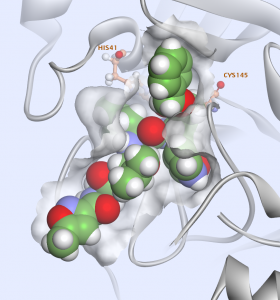
Nous avons commencé avec une bibliothèque contenant des médicaments approuvés par la FDA avec 2 684 composés. Nous avons retenu des composés de poids moléculaire inférieur à 800 kDa et effectué un amarrage avec GOLD du CCDC. Plus tard, nous avons calculé les énergies libres de liaison avec le solvant implicite CHARMM et GBMV. Pour chaque pose, nous avons d'abord effectué une minimisation du ligand in situ avec une sphère de 14 de diamètre pour la flexibilité des résidus, et estimé l'entropie du ligand lors du calcul de l'énergie libre de liaison. La plupart des composés amarrés sont des médicaments protéases connus contre le VHC et le VIH. (Illustration 6)
De nombreux composés établissent des contacts dans les 4 sous-sites :S1, S'1, S2, S4 et certains avec HIS 41 et CYS 145, en particulier les inhibiteurs de la protéase du VIH.
Le composé le mieux noté est la Troxérutine, qui est un flavonoïde.
- Il a été démontré que les flavonoïdes inhibent certaines protéases 5 et récemment IC50 les valeurs ont été calculées à partir des courbes d'inhibition dose-dépendantes de l'herbacétine, de la rhoïfoline et de la pectolinarine sur le SRAS-CoV. Les valeurs mesurées étaient respectivement de 33,17, 27,45 et 37,78 μM.
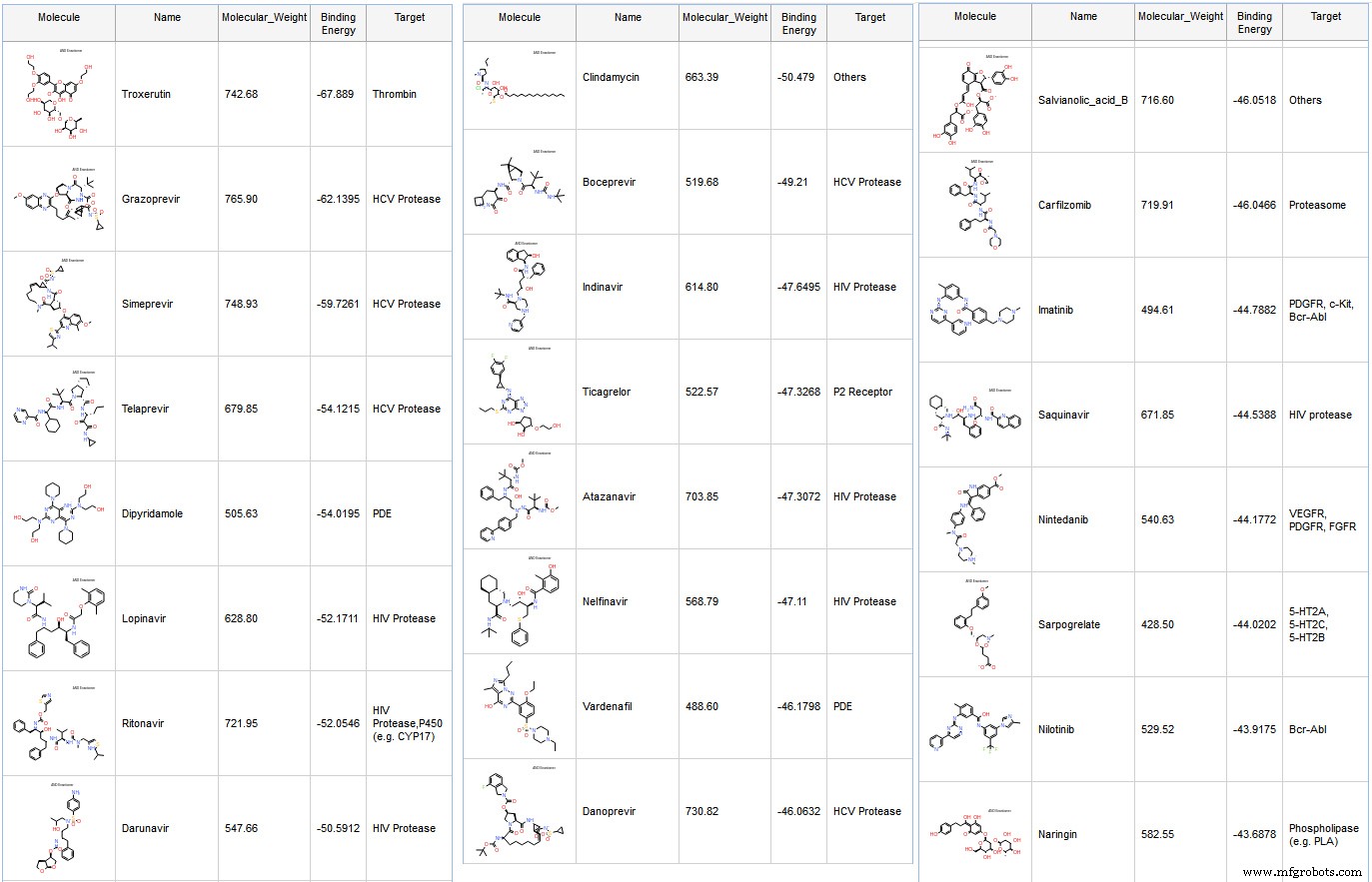
Le dipyridamole (Fig. 7), qui est le cinquième composé classé, a été mentionné récemment dans une prépublication Effets thérapeutiques du dipyridamole sur les patients atteints de COVID-19 présentant un dysfonctionnement de la coagulation, et il a été démontré qu'il supprime la réplication du HCoV-19 à une CE50 de 100 nM in vitro.
Figure 7 :Rendu 3D et carte d'interaction 2D du Dipyridamole dans la protéase principale. Des interactions avec les résidus catalytiques sont présentes, et avec certains des résidus mentionnés précédemment pour les inhibiteurs du SRAS-CoV.
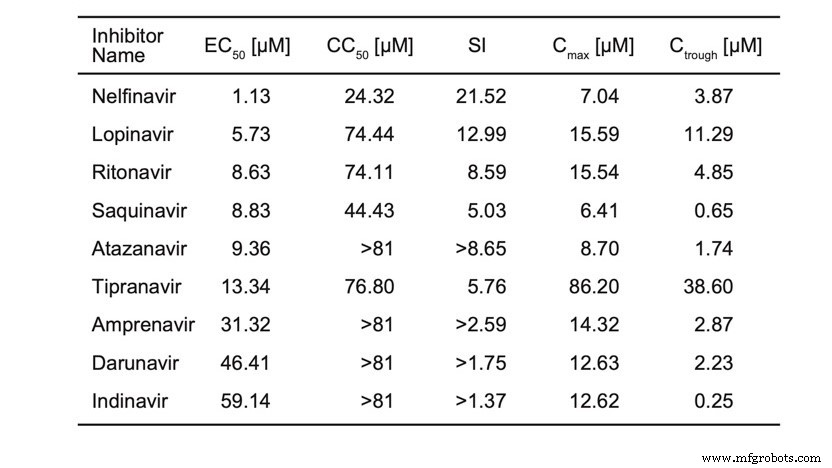
Une prépublication récente a rapporté des tests cellulaires de plusieurs inhibiteurs de la protéase du VIH :« Le nelfinavir inhibe la réplication du coronavirus 2 du syndrome respiratoire aigu sévère in vitro ."
Nous rapportons le tableau de la préimpression (tableau 1). Ce ne sont pas des IC50, mais des EC50, donc cela ne dit pas dans quelle mesure ces molécules se lient à la protéase. Cependant, cela montre que tous ces composés inhibent la réplication du SRAS-CoV-2 et que certains sont plus actifs que d'autres.
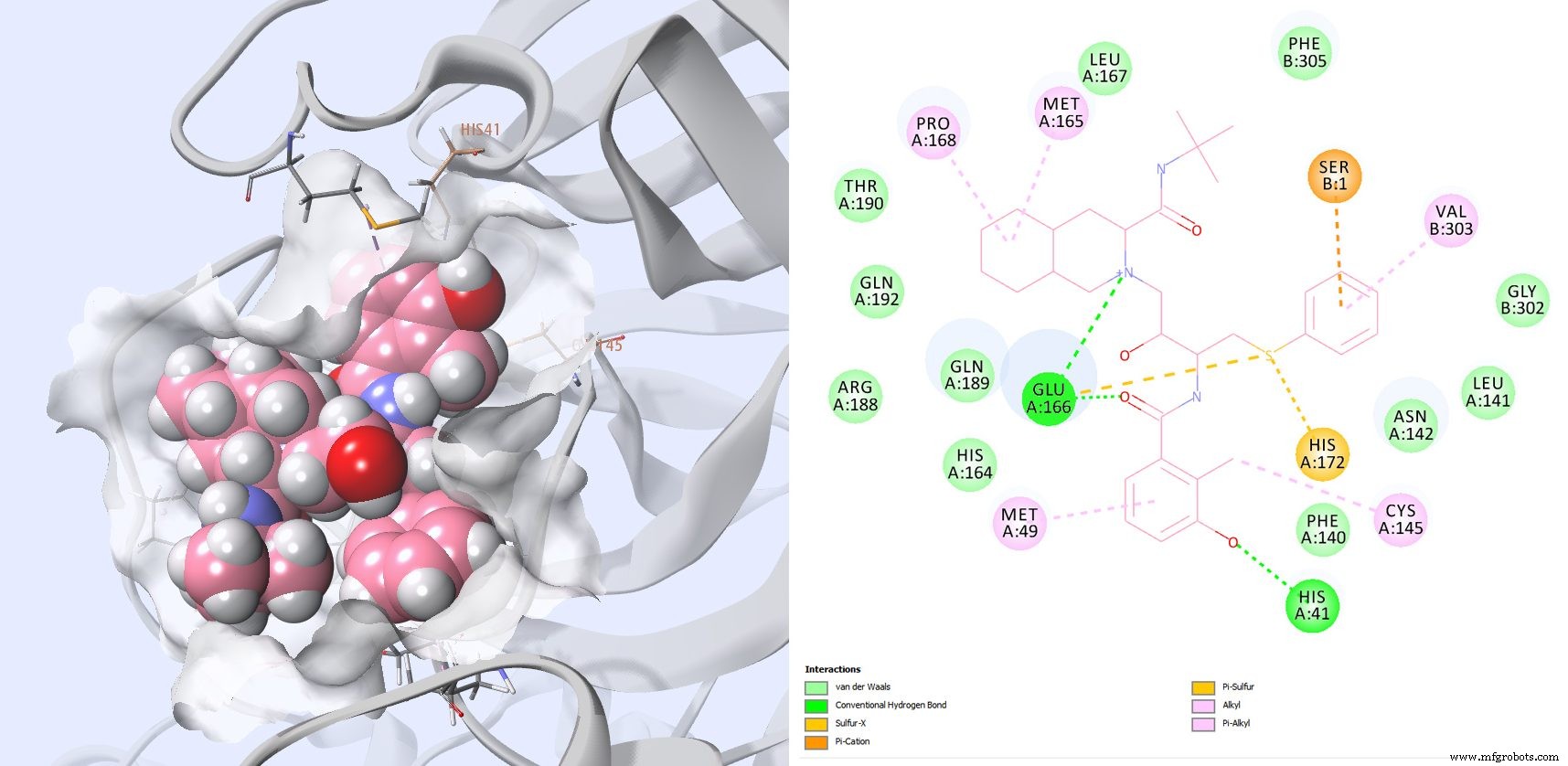
Le ritonavir et le lopinavir interagissent avec de nombreux résidus précédemment observés dans d'autres composés ciblant la protéase du SRAS-CoV (Fig. 8 et 9). Bien que plusieurs essais cliniques soient en cours avec une combinaison de ces deux molécules, il n'y a malheureusement pas de données IC50 disponibles pour le Ritonavir ou le Lopinavir sur le SARS-CoV-2.
Figure 8 :Pose la mieux notée pour le Ritonavir :et diagramme d'interaction 2D montrant que le Ritonavir interagit avec les deux résidus catalytiques :CYS145 et HIS41, mais aussi GLU166, PRO168 et GLN189 comme on le voit avec d'autres inhibiteurs connus du SARS-CoV.
Figure 9 :Pose la mieux notée pour le Lopinavir :et diagramme d'interaction 2D montrant que le Lopinavir interagit avec les deux résidus catalytiques :CYS145 et HIS41, mais aussi GLU166, PRO168 et GLN189 comme on le voit avec d'autres inhibiteurs connus du SRAS-CoV.
Le nelfinavir (Fig. 10) a une affinité de liaison prédite élevée dans nos calculs et a également été caractérisé contre COVID19 dans une prépublication récente :« Nelfinavir is active against SARS-CoV-2 in Vero E6 cells. »
Une deuxième prépublication montre que l'atazanavir est actif sur les cellules infectées par le SRAS-CoV-2 :« L'atazanavir inhibe la réplication du SARS-CoV-2 et la production de cytokines pro-inflammatoires. »
Une structure PDB 6W63 qui contient un nouvel inhibiteur du SARS-CoV-2 a été publiée après le début de ce travail (Fig. 11). Il n'y a aucune information publiée sur l'affinité de liaison de ce composé au SARS-CoV-2 Mpro. Les chaînes latérales dans le site de liaison sont presque dans la même orientation que pour 6LU7. Nous avons ancré nos composés et les avons réévalués avec les approches MMGBSA comme précédemment et avons trouvé des résultats très similaires.
Figure 11 :structure PDB 6W63 :protéase principale du SARS-CoV-2 en complexe avec l'inhibiteur X77.
Perspectives
Le site de liaison de la protéase du SARS-CoV-2 est très grand, contient quatre sous-sites et peut accueillir de nombreux ligands différents avec une affinité de liaison modérée. Comme expliqué ici, le criblage virtuel est un outil utile pour identifier les inhibiteurs possibles et a pu nous donner une explication structurelle sur la façon dont les inhibiteurs possibles pourraient interagir avec la protéase.
Cependant, toutes ces hypothèses nécessitent une confirmation par des preuves expérimentales, comme la mesure de l'IC50, pour construire des modèles plus robustes.
Ici, nous n'avons pas effectué d'amarrage covalent, mais comme les composés les plus actifs contre la protéase du SRAS-CoV étaient covalents, cela pourrait être une exigence d'inhibiteurs puissants. Les inhibiteurs covalents présentent également des avantages par rapport aux inhibiteurs réversibles, tels qu'une forte affinité cible et une durée de vie prolongée chez les patients.
Pour qu'un médicament soit actif chez un patient, nous devons considérer d'autres aspects pharmacologiques tels que la pharmacocinétique. Il sera nécessaire d'évaluer la capacité de ces médicaments à atteindre les concentrations plasmatiques et pulmonaires cibles après un dosage approuvé chez l'homme.
Il n'entre pas dans le cadre de ce travail de commenter d'autres recherches de conception basées sur la structure, car cela ne serait pas opportun pour l'épidémie actuelle. Notre objectif était d'évaluer des thérapies facilement utilisables pour des médicaments candidats potentiels contre COVID19.
Nous tenons à remercier le Cambridge Crystallographic Data Center (CCDC) pour la permission d'utiliser GOLD pour le criblage virtuel dans ce travail. GOLD a fait ses preuves dans le criblage virtuel, l'optimisation des pistes et l'identification du mode de liaison correct des molécules actives. Une interface vers GOLD est disponible dans Discovery Studio.
FORMATION EN LIGNE PIPELINE PILOTE
La modélisation et la simulation proposent de nouvelles perspectives pour le SARS-CoV-2
Produits biologiques
- La Chine va établir une liaison en matière de brevets
- La chaîne d'approvisionnement en médicaments aux États-Unis en crise :solutions aux pénuries
- COVID-19 a révélé des failles fatales dans la chaîne d'approvisionnement des médicaments aux États-Unis
- Comment la blockchain pourrait aider à lutter contre les médicaments contrefaits
- Application de la blockchain et de l'apprentissage automatique aux chaînes d'approvisionnement en médicaments
- Test tout-en-un pour la surveillance du COVID-19
- Un nouvel effet photonique pourrait accélérer le développement de médicaments
- Smartwatch suit les niveaux de médicaments
- 5 Ws du capteur RapidPlex SARS-CoV-2



