La modélisation et la simulation proposent de nouvelles perspectives pour le SARS-CoV-2
Dans notre blog précédent, nous avons discuté de l'utilisation d'outils de modélisation prédictive pour construire des structures initiales au niveau atomique de cibles médicamenteuses potentielles (par exemple, des protéines) et pour affiner des régions qui n'étaient pas déterminables expérimentalement (Voir la vidéo ). Ces outils incluent l'ajout d'atomes d'hydrogène et de boucles flexibles qui ne sont parfois pas résolvables expérimentalement. Nous avons examiné cela dans le contexte de la cryomicroscopie électronique (cryo-EM) de la protéine de pointe SARS-CoV-2 (S) récemment publiée dans la revue Science (DOI :10.1126/science.abb2507).
Dans ce blog, nous détaillerons comment la modélisation moléculaire et la simulation de modèles structurels raffinés tels que la protéine SARS-CoV-2 S peuvent aider à générer de nouvelles hypothèses pour la découverte et la conception de thérapies putatives pour traiter le COVID-19.
La liaison aux médicaments dépend des changements structurels
Dans les systèmes vivants, les protéines existent naturellement en tant qu'entités dynamiques. Leur dynamique prédétermine souvent leur fonction. Le physicien Richard Feynman a dit un jour :
« Si nous devions nommer l'hypothèse la plus puissante de toutes, qui conduit indéfiniment à essayer de comprendre la vie, c'est que toutes les choses sont faites d'atomes et que tout ce que font les êtres vivants peut être compris en termes des tremblements et des remuements des atomes. 1
La protéine SARS-CoV-2 S ne fait pas exception au principe de Feynman. Avant d'entrer dans les cellules humaines, la protéine S se lie à un récepteur appelé enzyme de conversion de l'angiotensine 2 (ACE2). 2 Le domaine de liaison au récepteur (RBD) fait partie de la protéine S qui se lie à l'ACE2 (Figure 1A ). Le RBD peut exister dans au moins deux états conformationnels primaires appelés états haut (accessible au récepteur) et bas (inaccessible au récepteur) (Figure 1B ). Lorsque le RBD est à l'état haut, la protéine S est plus « ouverte » pour faciliter la liaison de l'ACE2. 2 Des études ont suggéré que le duvet, l'état inaccessible aux récepteurs, est plus stable. 2 Cela implique que des thérapies putatives telles que de petites molécules organiques capables de se lier au RBD pourraient stabiliser le RBD à l'état down et empêcher le virus d'interagir avec l'ACE2 ; ainsi, empêchant COVID-19 d'infecter les gens.
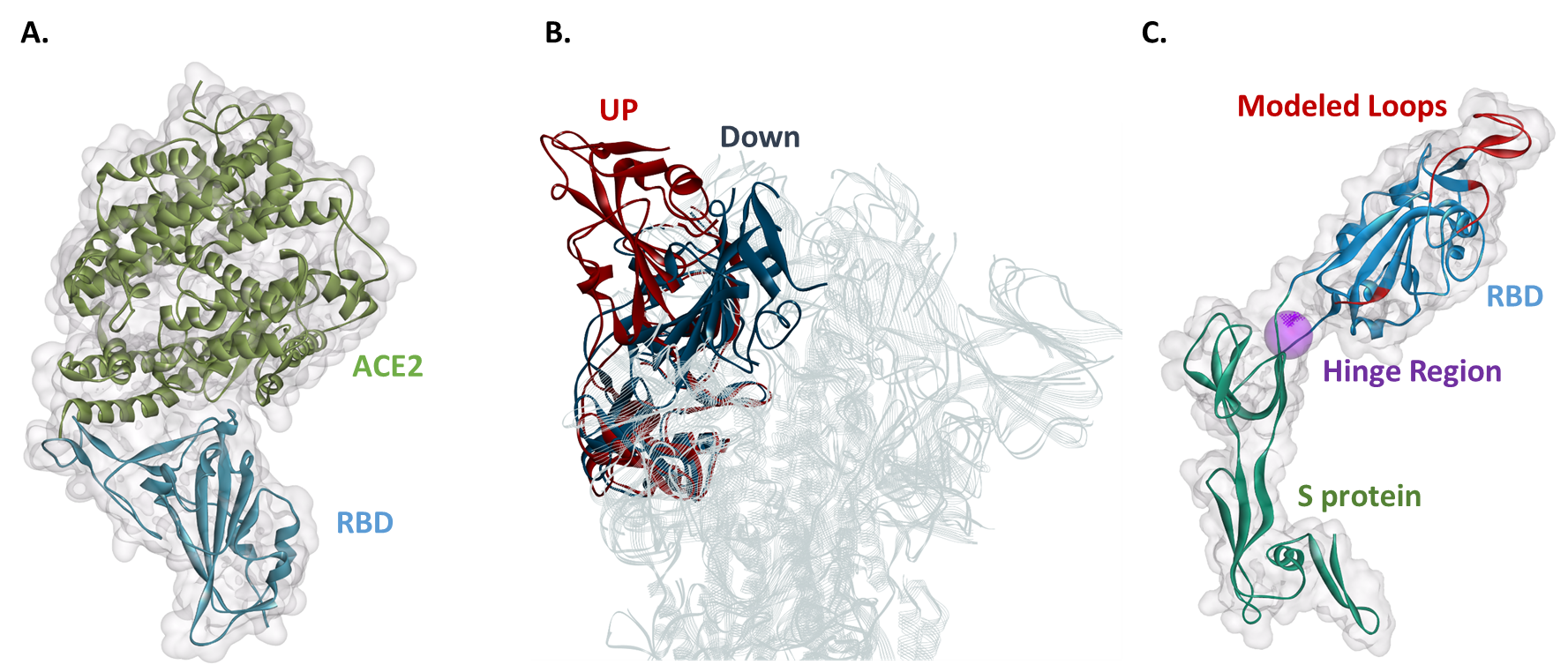
Le RBD de la protéine S est comme une charnière sur une porte
Un linker flexible relie le RBD à la protéine S restante. La flexibilité de l'éditeur de liens permet à RBD de passer de l'état bas à l'état haut grâce à un mouvement de flexion de la charnière (Figure 1B ). À partir de la protéine SARS-CoV-2 S, nous avons extrait le RBD avec le lieur flexible associé et le domaine adjacent (Figure 1C ). Nous avons utilisé la structure RBD de la protéine S à l'état haut (PDB 6VYB), mais il manque à cette structure trois boucles potentiellement importantes pour la liaison de l'ACE2 (Figure 1C ). En conséquence, nous avons dû construire un modèle d'homologie du RBD avec le linker flexible en utilisant la structure cryo-EM à partir de laquelle la troncature a été faite à l'origine (PDB 6VYB) avec un modèle supplémentaire. Le modèle supplémentaire était la structure cristalline du RBD seul en complexe avec ACE2 (PDB 6M17) (Figure 1A ).
La structure RBD contient des boucles dont un de plus de 20 résidus d'acides aminés, qui ne sont pas présents dans la structure à l'état ouvert (PDB 6VYB). Deux de ces boucles forment l'interaction avec ACE2; ainsi, la modélisation d'homologie est nécessaire pour comprendre les interactions de la protéine Spike. Nous pouvons ensuite affecter des atomes d'hydrogène dans des états de protonation appropriés pour imiter des conditions physiologiques telles que le pH.
Nous pourrions ensuite effectuer une simulation de dynamique moléculaire (MD) pour simuler la transition conformationnelle et/ou prédire les sites de liaison possibles où de petites molécules putatives peuvent se lier pour perturber l'interaction de la protéine S avec ACE2. Lorsque nous avons prédit les sites de liaison possibles à l'aide de BIOVIA Discovery Studio pour notre modèle d'homologie du RBD avec le lieur flexible, nous avons identifié un site de liaison situé à la charnière du lieur flexible (Figure 1C ). Nous avons qualifié cette région de charnière et notons qu'elle pourrait mériter une enquête plus approfondie pour la découverte de thérapeutiques putatives. Si une petite molécule devait se lier à la région charnière, elle pourrait éventuellement verrouiller le RBD dans un état bas et ainsi empêcher la liaison de l'ACE2.
Enquêtes complémentaires
Des recherches approfondies, y compris des prédictions informatiques et des expériences biologiques, pourraient clarifier davantage l'utilité de la région charnière. Des exemples de prédictions informatiques peuvent inclure l'analyse en mode normal (NMA) et/ou la simulation MD. 3 Par exemple, une longue échelle de temps, environ des centaines de nanosecondes, de simulation MD pourrait permettre aux scientifiques d'échantillonner plusieurs conformations de la région charnière.
D'un autre côté, une NMA pourrait fournir une estimation grossière et rapide des transitions conformationnelles. 3 Les conformations spécifiques de la simulation MD et/ou de la NMA sont des points de départ pour le criblage virtuel à haut débit de bases de données potentielles de petites molécules. Les scientifiques pourraient amarrer et marquer chaque petite molécule pour toutes les conformations. Ils pourraient ensuite classer toutes les poses résultantes et soumettre les meilleurs résultats pour validation expérimentale. Des études ont montré que cette méthode de découverte informatique de médicaments, souvent appelée criblage virtuel basé sur un ensemble, améliore les chances d'identifier des candidats-médicaments. 4 La méthode reflète également la réalité selon laquelle la liaison au médicament dépend de changements structurels dans la protéine, comme indiqué ci-dessus. Nous pensons que les résultats préliminaires identifiés ici sont intéressants et méritent d'être approfondis.
Deuxièmement, nous aimerions noter que le modèle structurel raffiné de la protéine S pourrait être utilisé comme cible pour l'immunothérapie. 5 Les scientifiques pourraient concevoir des anticorps monoclonaux qui se lient aux protéines du SRAS CoV-2 S sur la base des connaissances préalables du site de liaison ACE2. Ils pourraient alors effectuer in silico études d'affinité-maturation pour améliorer la spécificité de liaison. 6
En tant que soutien actif de la communauté scientifique qui collabore aujourd'hui aux solutions COVID-19, BIOVIA Dassault Systèmes développe BIOVIA Discovery Studio. Cet environnement éprouvé de modélisation et de simulation des sciences de la vie rassemble plus de 30 ans de recherche évaluée par des pairs et de classe mondiale in silico technique. Le logiciel fournit aux scientifiques un ensemble complet d'outils à utiliser depuis l'identification des cibles jusqu'à l'optimisation des pistes, y compris des outils pour la conception et l'analyse de produits biologiques, les simulations classiques, la conception basée sur la structure et les fragments, le criblage virtuel de ligands, ainsi que l'ADME et la prédiction de la toxicité.
Dans le cadre de la responsabilité sociale d'entreprise de Dassault Systèmes, BIOVIA a le plaisir d'offrir aux groupes de recherche universitaires qualifiés impliqués dans des études liées au SRAS-CoV-2 une licence gratuite de six mois pour BIOVIA Discovery Studio pour les aider dans la recherche de candidats-médicaments thérapeutiques rapides, sûrs et efficaces contre le virus SARS-CoV-2. Si vous êtes un chercheur universitaire dans ce domaine, veuillez demander une licence de logiciel et télécharger. Cette offre est valable jusqu'au 30 juin 2020.
Produits biologiques
- Pixus :nouvelles façades épaisses et robustes pour cartes embarquées
- DSM et Nedcam vont développer de nouvelles applications pour l'impression 3D grand format
- Teradyne prévoit un nouveau hub de cobots pour les sociétés de portefeuille UR et MiR
- PLASTICS publie une nouvelle norme de sécurité pour la robotique et le moulage par injection
- Une nouvelle feuille de route pour les chaînes d'approvisionnement en pétrole et en gaz
- Coaching pour le développement durable :mise en œuvre et maintien de nouveaux processus et changements
- B&R dévoile un nouvel outil de simulation pour le développement de jumeaux numériques
- ABB fournit la planification de l'automatisation et de l'électrification d'une nouvelle mine en Suède
- La 5G et Edge soulèvent de nouveaux défis en matière de cybersécurité pour 2021



