Contrôle de la translocation d'ADN via des nanopores à l'état solide
Résumé
Par rapport au statut des bio-nanopores, plusieurs défis doivent encore être surmontés avant que les nanopores à l'état solide puissent être appliqués dans le séquençage commercial de l'ADN. Une faible résolution spatiale et une faible résolution temporelle sont les deux défis majeurs. En raison des restrictions sur la longueur des nanopores et des propriétés de surface des nanopores à l'état solide, il est encore possible d'améliorer la résolution spatiale. Pendant ce temps, la translocation de l'ADN est trop rapide sous une force électrique, ce qui entraîne l'acquisition de quelques points de données valides. La résolution temporelle des nanopores à l'état solide pourrait ainsi être améliorée si la vitesse de translocation de l'ADN est bien contrôlée. Dans cette mini-revue, nous résumons brièvement les méthodes d'amélioration de la résolution spatiale et nous nous concentrons sur les méthodes contrôlables pour promouvoir la résolution de la détection des nanopores. De plus, nous offrons une perspective sur le développement du séquençage de l'ADN par les nanopores.
Introduction
Au cours des dernières décennies, de nombreux progrès ont été réalisés dans l'application du séquençage de l'ADN pour lire les séquences de bases dans le génome [1, 2]. Pour développer des médicaments personnalisés, les chercheurs ont recherché une méthode de séquençage de l'ADN plus rapide et moins chère, où les médicaments cibles et les traitements médicaux peuvent être appliqués spécifiquement à un individu [3, 4]. Parce que la technologie des nanopores a été utilisée dans la détection de l'ADN [5], elle a été considérée comme une méthode efficace pour le séquençage de nouvelle génération [6, 7]. La technologie Nanopore est une plate-forme prometteuse pour identifier des détecteurs à molécule unique hautement sensibles pour l'ADN [5] ou l'ARN [8]. Dans le schéma de détection de base, une chambre électrochimique est séparée en deux réservoirs (compartiments cis et trans) par une fine membrane avec un nanopore [9], qui relie la solution conductrice et l'analyte dans la chambre électrochimique. En appliquant une tension à travers la membrane, les ions électrolytiques traversent le nanopore à l'état solide et forment un courant de pore, qui est mesuré à l'aide d'une configuration de patch clamp avec l'électronique ultra-sensible associée. Lorsqu'une molécule ou un complexe moléculaire traverse le nanopore, l'analyte peut exclure certains ions du volume défini par le nanopore, qui peuvent être détectés en surveillant de brefs changements dans le courant. À partir du temps de séjour (temps de séjour) dans le nanopore et de la signature d'amplitude actuelle, des informations sur les molécules peuvent être obtenues. La résolution spatiale du séquençage des nanopores est déterminée par les dimensions du nanopore, ce qui suggère qu'il peut être utilisé comme un seul capteur pour les petits objets moléculaires résultant en une signature de courant détectable. De plus, les capteurs à nanopores sont faciles à intégrer dans des dispositifs de laboratoire sur puce hautement portables et à miniaturiser [10].
Des progrès significatifs ont été réalisés dans le séquençage de l'ADN par des nanopores, tels que les nanopores à l'état solide [2, 11] et les nanopores protéiques [2, 7]. Le séquençage de l'ADN par des nanopores de protéines a été réalisé [7]. Cependant, les systèmes de nanopores de protéines ont des limites pour l'étude des molécules biologiques. Il y a moins de contraintes avec les nanopores à l'état solide par rapport aux nanopores biologiques/protéiques. Ces difficultés peuvent être surmontées par des nanopores à l'état solide. Par rapport aux nanopores de protéines, ils sont fonctionnels sur des plages de températures, de tensions et de conditions de solvant plus larges et peuvent être réglés en diamètre avec une précision inférieure au nanomètre. Ils sont prometteurs pour une application dans la technologie de nouvelle génération pour le séquençage de l'ADN.
De nombreux nanopores à l'état solide de différents matériaux et structures ont été fabriqués pour les capteurs d'ADN. Cependant, le séquençage de l'ADN n'est pas réalisé par des nanopores à l'état solide. Pour les capteurs à nanopores à l'état solide, il existe un besoin de surmonter deux obstacles majeurs, concernant leur résolution spatiale et leur résolution temporelle, avant leur application commerciale au séquençage de l'ADN. La difficulté concernant la résolution spatiale est que le nanopore à l'état solide peut distinguer le minuscule espacement entre deux nucléotides voisins afin d'obtenir une résolution à base unique. L'obstacle de la résolution temporelle est que la translocation de l'ADN est trop rapide sous une force électrique, ce qui entraîne l'acquisition de très peu de points de données valides par les patch clamps existants ou d'autres systèmes d'acquisition de signaux. Cette mini-revue présente un aperçu des différentes méthodes pour améliorer la résolution spatiale et la résolution temporelle de la détection d'ADN nanopore à l'état solide. Cette mini-revue se concentre également davantage sur les méthodes de ralentissement de la translocation de l'ADN à travers les nanopores à l'état solide.
Résolution spatiale
En 2001, des nanopores à l'état solide de nitrure de silicium ont été signalés pour la première fois par Li et al. [12]. Divers nanopores à l'état solide ont été démontrés pour la détection moléculaire de l'ADN, tels que l'oxyde de silicium [13], le silicium [14], Al2 O3 [15], et HfO2 [16]. Alors que les nanopores à l'état solide peuvent finalement être robustes aux conditions chimiques et mécaniques, ils ont certaines limites, telles qu'une faible résolution spatiale. En raison de l'épaisseur des matériaux, des dizaines de bases peuvent traverser les nanopores à l'état solide à la fois. Actuellement, le nanopore de nitrure de silicium le plus fin est de 3 nm, ce qui ne distingue toujours pas les quatre types de base [17].
Fait intéressant, l'épaisseur d'une seule couche de matériau bidimensionnel (2D) est d'environ 3,0 à 11,0 , ce qui est comparable à l'espacement entre deux nucléotides voisins le long de l'ADN ss (3,2 à 5,2 ) [18]. Membranes bidimensionnelles, telles que le graphène (3.4 Å [19]), MoS2 (6,5 Å [18]), WS2 (7 Å [20]) et h-BN (11 Å [21]), ont été démontrés pour détecter les translocations d'ADN [21,22,23] en raison de leur rapport signal/bruit et de leur résolution spatiale élevés. Il ressort clairement de leur résolution spatiale que ces matériaux peuvent être utilisés pour la détection d'ADN. En outre, des techniques de croissance reproductibles et des procédures de transfert à grande échelle permettent de fabriquer à grande échelle des pores sub-nanométriques dans des membranes 2D.
Le graphène est une feuille atomiquement mince d'atomes de carbone disposés en un réseau en nid d'abeilles bidimensionnel [24]. Les chercheurs ont démontré que des molécules d'ADN uniques en solution peuvent être détectées et caractérisées avec des nanopores de graphène [22, 25]. Cependant, il existe de fortes interactions hydrophobes entre les nucléotides de l'ADN et le graphène, et l'ADN se bouchera et collera gravement aux nanopores de graphène, ce qui aurait un impact marqué sur la vitesse de translocation [26]. Modifié avec les groupes hydrophiles [26] ou enrobé, un matériau hydrophile [25] sur le graphène pourrait améliorer l'hydrophilie du graphène et éviter que l'ADN ne colle à sa surface. Malheureusement, soit la modification avec des groupes hydrophiles, soit le revêtement avec des matériaux hydrophiles augmenteront l'épaisseur du film en suspension, entraînant une augmentation de l'épaisseur des nanopores, diminuant ainsi la résolution spatiale du graphène.
Le dichalcogénure de métal de transition en couches est un autre matériau 2D, qui comprend le MoS2 [18, 27] et WS2 [20]. Un rapport signal/bruit élevé (SNR> 10) et une amélioration de cinq fois du signal de courant ionique ont été détectés lors de la translocation de l'ADN via le MoS2 membrane nanopore [18]. Pendant ce temps, MoS2 est hydrophile, donc aucun traitement de surface spécial n'est nécessaire pour éviter l'interaction hydrophobe entre l'ADN et sa surface. L'autre matériel, WS2 a une bande interdite directe de 2,1 eV [28], et son émission de photoluminescence (PL) est plus forte que celle du MoS2 bien étudié [29]. Danda et al. [20] a fabriqué un WS2 nanopore et a démontré l'obtention d'une taille de nanopore contrôlée atomiquement à l'aide de courtes impulsions lumineuses, ce qui peut avoir des effets positifs sur les nanopores à l'état solide pour la détection de l'ADN.
En outre, Liu et al. [21] ont rapporté la première expérience de translocation d'ADN à travers des nanopores h-BN. Semblable au graphène, le h-BN a une faible hydrophilie, ce qui entraînera le blocage des nanopores par l'ADN. Par la suite, Zhou et al. [23] ont amélioré avec succès l'hydrophilie des nanopores h-BN en tirant parti de l'antioxydation et de l'intégrité du matériau h-BN après traitement UV-ozone. La nature isolante du h-BN peut présenter une durabilité et des propriétés isolantes plus remarquables dans une solution à haute résistance ionique que dans le graphène. Il s'agit d'un candidat compétitif pour obtenir une résolution à base unique sur des structures nanoporeuses ultrafines.
L'utilisation de matériaux bidimensionnels peut potentiellement augmenter la résolution spatiale du dispositif pour obtenir une résolution à un seul nucléotide. Bien que des expériences de détection d'ADN pour plusieurs matériaux bidimensionnels aient été rapportées, personne n'a signalé la réalisation d'un séquençage d'ADN nanopore à l'état solide. La résolution temporelle du séquençage des nanopores est également un défi.
Résolution temporelle
La vitesse de translocation de l'ADN à travers un nanopore à l'état solide est très rapide, jusqu'à 0,01–1 µs/base [30], ce qui conduit à très peu de données efficaces collectées par les patch clamps commerciaux. En tant que tel, il n'est pas possible de distinguer chaque base sur la base du signal de courant de blocage. Les vitesses de translocation de l'ADN des nanopores à l'état solide de matériaux 2D, tels que le graphène, MoS2 , WS2 , et h-BN, sont illustrés à la figure 1. Idéalement, la vitesse de translocation de l'ADN dans un nanopore devrait être de 1 à 100 bp/ms pour permettre un enregistrement satisfaisant du signal de chaque nucléotide [32].
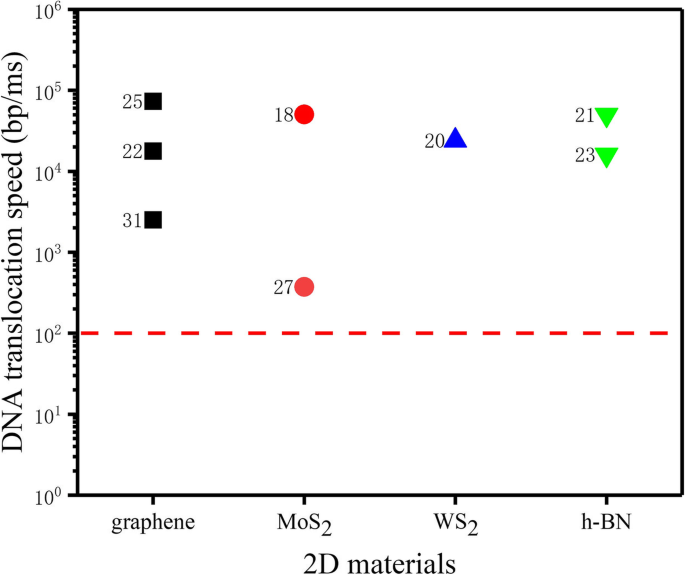
Vitesse de translocation de l'ADN du nanopore à l'état solide 2D [18, 20, 21, 22, 23, 25, 27, 31]. La ligne rouge en pointillé indique un taux de translocation d'ADN de 100 bp/ms
Dans ce contexte, ralentir la vitesse de translocation de l'ADN est un objectif important poursuivi par de nombreux chercheurs. Diverses méthodes ont été développées pour ralentir la translocation d'ADN afin d'améliorer la résolution temporelle de la détection des nanopores à l'état solide. La méthode habituelle consiste à modifier l'influence de facteurs expérimentaux tels que la température [33], la viscosité de l'électrolyte [27], la tension de commande [34], la concentration en ions [35] et la densité de charge de surface d'un nanopore [36] en modifiant l'ADN translocation à travers elle. Wanunu et al. [33] se sont concentrés sur le ralentissement de la translocation de l'ADNdb à travers les nanopores à l'état solide en modifiant la température, la tension et la longueur de l'ADN. De plus, Feng et al. [27] ont montré qu'un système de gradient de viscosité, basé sur des liquides ioniques à température ambiante, peut être utilisé pour contrôler la dynamique de la translocation de l'ADN via MoS2 nanopores, et a démontré que la viscosité élevée des liquides ioniques à température ambiante fournit des vitesses de translocation optimales d'un seul nucléotide de 373 bp/ms.
Bien que de nombreuses approches aient le potentiel de réduire la vitesse de translocation de l'ADN et de faciliter la détection du courant ionique, elles ne peuvent toujours pas répondre aux exigences du séquençage de l'ADN. Par conséquent, il est nécessaire de développer des méthodes plus radicales pour contrôler le passage de l'ADN à travers les nanopores. Ici, nous discutons des méthodes de contrôle de la structure des nanopores et du mouvement quantitatif de l'ADN pour améliorer la résolution temporelle.
Système à deux nanopores
Les chercheurs ont utilisé des systèmes à deux nanopores pour manipuler les translocations de molécules d'ADN, qui peuvent détecter de manière contrôlée la même molécule plusieurs fois. Deux nanopores empilés, séparés par un compartiment de cavité micrométrique [37] (Fig. 2a), peuvent piéger les molécules d'ADN pendant un certain temps et les libérer de manière contrôlée. La dynamique des molécules d'ADN peut être déduite des signaux des deux pores par analyse de corrélation, qui fournit une preuve électrique directe de la translocation. De plus, en raison des barrières entropiques du système de nanopores à deux couches, le mouvement brownien des molécules d'ADN peut être confiné, ce qui peut améliorer la précision du séquençage de l'ADN avec les nanopores. Par rapport aux techniques de détection de molécule unique, les molécules d'ADN peuvent être mesurées plusieurs fois dans des systèmes de nanopores à deux couches en organisant en série plusieurs pores au lieu de les faire passer dans un seul pore [39].
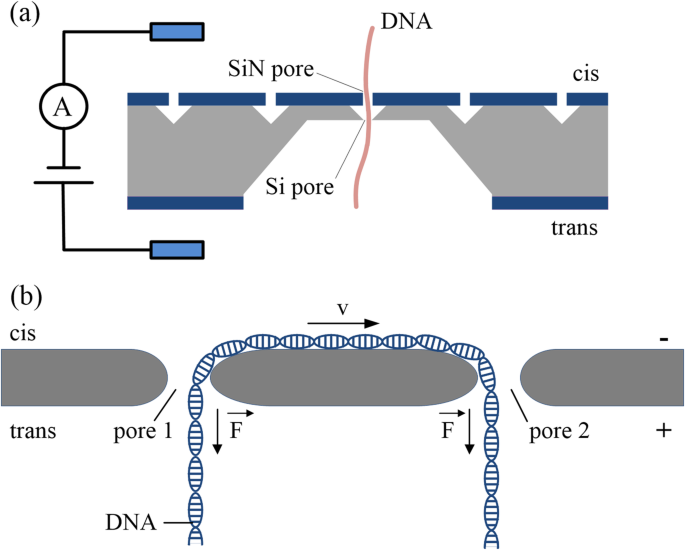
un Schéma du système de nanopores à deux couches utilisé pour la détection d'ADN nanopore [37]. b Schéma du système à double nanopore utilisé pour la détection d'ADN nanopore [38]
Les systèmes à double nanopore fournissent une autre méthode pour contrôler le transport moléculaire et relier efficacement les molécules entre deux pores; ils constituent une approche mécaniste sans marqueur pour la manipulation de l'ADN [40]. Dans un système à double nanopore, il y a deux nanopores adressables indépendamment et adjacents dans la même membrane à l'état solide. Lors du passage électrophorétique d'une molécule d'ADN à travers l'un des nanopores, une seule molécule d'ADN peut être capturée dans les deux pores, conduisant à un « tir à la corde » entre les deux nanopores [38] (Fig. 2b). Par conséquent, des forces sont appliquées aux différentes extrémités d'une molécule d'ADN, ce qui ralentit et même arrête complètement son mouvement. Le système à double nanopore ouvre une nouvelle voie au piégeage mécanique de l'ADN dans les nanopores à l'état solide, et c'est une technique prometteuse pour mesurer une large gamme de biomolécules avec les avantages d'être sans marqueur et d'avoir un signal élevé. rapport bruit/bruit et faible coût. Il peut efficacement confiner et piéger les molécules d'ADN pour ralentir la translocation de l'ADN et peut également être utilisé pour étudier la physique de ce bras de fer à l'échelle nanométrique sur l'ADN [41].
Piège Optique Nanopore
Les nanopores de piège optique permettent aux pinces optiques de piéger des particules de moins de dizaines de nanomètres. Il permet le piégeage optique de protéines [42], de fragments d'ADN [43] et d'autres biomolécules [44], ainsi que de petits virus [45]. La théorie de base derrière les nanopores des pièges optiques est un piège optique à contre-action auto-induit [46]. Le faisceau laser focalisé sur la région du réseau de nanopores formera un champ lumineux local à haute densité de puissance au bord de la couche métallique dans le trou. Lorsqu'une particule se déplace entre le champ lumineux local, elle peut provoquer un changement important dans la transmission optique locale, qui à son tour produira une grande force optique et diélectrique sur la particule. Une structure à double nano-trous est utilisée pour briser le goulot d'étranglement de taille des particules capturées. Mohammed et al. [47] ont démontré l'utilisation potentielle de nanopores de piège optique avec de la silice de 20 nm et des nanoparticules d'Au. La forme en haltère du double nanotrou a été fraisée dans le film d'Au, et un nanopore de 25 nm a été percé à travers le Six en suspension. Ny membrane au milieu du double nanotrou, comme le montre la figure 3a. Force électrophorétique entraînant des nanoparticules à travers le nanopore, lors du passage à travers le bord du trou, la force plasmonique auto-induite d'action de retour existant entre les extrémités du double nanotrou s'oppose à la force électrophorétique, réduisant la vitesse des nanoparticules. Les résultats ont montré que le piégeage optique a prolongé le temps de translocation électrophorétique de quatre ordres de grandeur. Kim et al. [43] ont réalisé la capture optique d'ADN plasmidique et d'ADN lambda en utilisant les structures nanoplasmoniques de nanopores uniques. La technologie a des applications potentielles pour la détection d'ADN et plusieurs champs lumineux locaux peuvent être configurés pour réaliser une détection parallèle. Cependant, l'oscillation du courant ionique à haute fréquence peut affecter les résultats de détection de courant de l'ADN. Cela peut être dû à la compétition entre les forces d'action inverse électrophorétique et auto-induite, faisant flotter les nanoparticules de haut en bas à travers l'embouchure du nanopore.
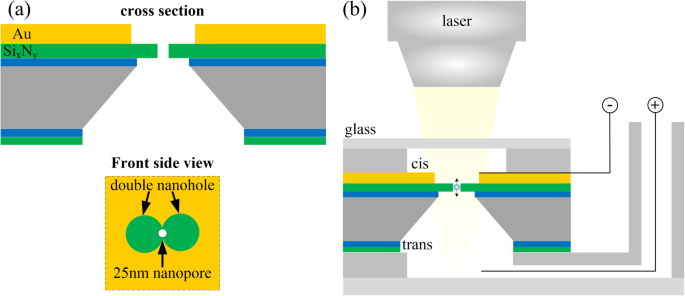
un Schéma de la puce à nanopores piège optique [47]. b Schéma du système de contre-action auto-induit
Pinces optiques
Les pincettes optiques peuvent être utilisées pour contrôler la translocation moléculaire à travers les nanopores et ont été couramment utilisées ces dernières années. En 2006, Keyser et al. [48] ont d'abord démontré un bras de fer moléculaire entre les forces électrophorétiques et mécaniques en appliquant des pincettes optiques pour contrôler la translocation de l'ADN par SiNx nanopores. Ce système agit comme un simple ressort de Hooke, et la force de tension sur le bourrelet peut être calculée selon la loi de Hooke :F pas =−k piège Z , où F pas est la force optique, k piège est la rigidité du piège le long de la direction de déplacement, et Z est la déformation linéaire du bourrelet [48]. La méthode de la pince à épiler optique, qui piège une perle de polystyrène attachée à l'ADN dans le croisement d'un faisceau laser focalisé, peut manipuler la perle de polystyrène attachée à l'ADN en trois dimensions et a une plage de sensibilité à la force pico-newton, comme le montre la figure 4a. . Pour piéger l'ADN en translocation à l'intérieur du nanopore, la force de tension a été réglée pour équilibrer la force du champ électrique (F el ) sur l'ADN. Par conséquent, la tension peut être utilisée à la fois pour réduire la vitesse de translocation de l'ADN et pour extraire la molécule d'ADN d'un nanopore [52]. Ce système permet un échantillonnage spatial simultané et des mesures de force à haute résolution d'acides nucléiques et de protéines, ce qui a permis de réaliser des progrès significatifs dans le séquençage de l'ADN. Cependant, la technique de la pince à épiler optique souffre de plusieurs difficultés fondamentales. Premièrement, il est difficile de passer à un grand nombre de nanopores. Deuxièmement, l'échauffement causé par le laser dans les pincettes optiques a un impact important sur le courant ionique à travers un nanopore et le niveau de bruit, nécessitant que la bille piégée optiquement soit à plusieurs micromètres du nanopore [53].
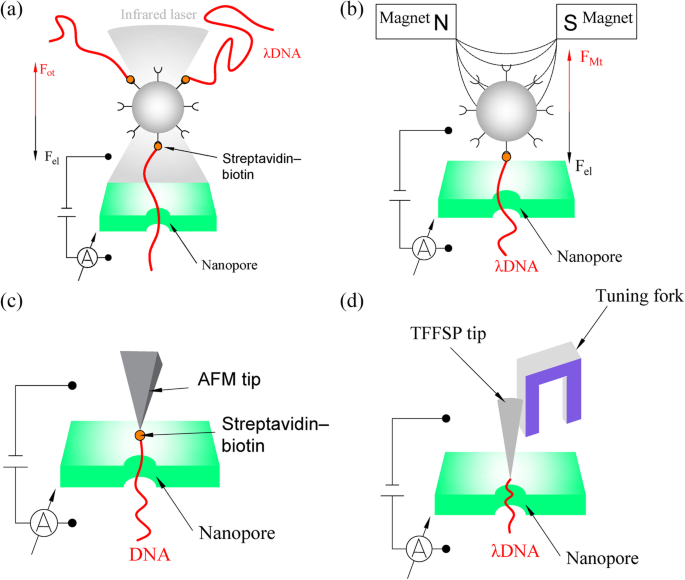
un Schéma de pinces optiques utilisées pour la détection d'ADN nanopore [48]. Lorsque le système de pincettes optiques est en équilibre, la force optique (Fot ) est égal à la force du champ électrique (Fel ). b Schéma des pincettes magnétiques utilisées pour la détection d'ADN nanopore [49]. Lorsque le système de pincettes magnétiques est en équilibre, la force magnétique (FMt ) est égal à la force du champ électrique (Fel ). c Schéma de l'AFM utilisé pour la détection d'ADN nanopore [50]. d Schéma du TFFS utilisé pour la détection d'ADN nanopore [51]
Pince à épiler magnétique
Les pincettes magnétiques offrent un autre moyen de contrôler la translocation de l'ADN par la force de tension, et il a été démontré que la technique de la pince magnétique est efficace pour ralentir la translocation de l'ADN [49]. Dans ce système, comme le montre la figure 4b, les molécules d'ADN peuvent être attachées à une bille magnétique de la taille d'un micromètre en utilisant une forte interaction or-thiol [54] ou streptavidine-biotine [49]. Ensuite, l'extrémité libre de l'ADN peut être capturée dans le nanopore par un champ électrique appliqué. Par la suite, deux aimants avec un petit espace peuvent être utilisés pour créer un gradient de champ magnétique. Cette technique peut équilibrer la force électrique sur l'ADN piégé pour réduire les vitesses de translocation et même l'électrophorèse inverse. Par rapport aux pinces optiques, les pinces magnétiques sont un candidat prometteur pour la spectroscopie de force massivement parallèle. Dans ce système, des centaines de billes et donc de molécules d'ADN peuvent être contrôlées simultanément dans des centaines de nanopores, ce qui est facilement extensible à de nombreux nanopores adressables. Cela peut accélérer le processus d'analyse par des ordres de grandeur. Cependant, par rapport aux pincettes optiques, un inconvénient évident de l'approche des pincettes magnétiques est le manque de contrôle tridimensionnel des molécules [55].
Sonde de détection de force
Bien que des progrès significatifs aient été réalisés dans le contrôle de la vitesse de translocation de l'ADN par des pincettes optiques et des pinces magnétiques, les méthodes de piégeage des billes ont un problème avec le mouvement brownien qui rend difficile le contrôle du mouvement de la bille avec une résolution inférieure à 10 nm [51] . Pour surmonter cela, l'AFM a été utilisé pour contrôler la vitesse de translocation de l'ADN [50], et il peut également mesurer simultanément la force et le courant de blocage. Dans une étude utilisant ce système, comme le montre la figure 4c, l'ADN a été attaché à la pointe d'une sonde AFM, puis il a été serré dans le support de sonde. En contrôlant le mouvement de la sonde, la translocation de l'ADN pourrait être contrôlée pour réduire sa vitesse et même inverser l'électrophorèse. De plus, en rétractant la pointe à une hauteur au-dessus de la surface correspondant à la longueur de la molécule, les mesures peuvent être répétées. Avec l'assistance de l'AFM, la détection de l'ADN a progressé dans la pratique et la théorie car la résolution de détection peut être considérablement améliorée par les signaux combinés des mesures de courant de blocage et de force AFM [56]. Cependant, il existe toujours un obstacle à l'application des techniques des nanopores dans la détection de l'ADN, à savoir l'apparition intermittente de fluctuations régulières de la force (et du courant) tous les 0,35-0,72 nm lorsqu'une molécule d'ADN glisse de manière relativement sans friction à travers un nanopore. Ces fluctuations sont attribuées à des nucléotides individuels se traduisant à travers le nanopore dans un mouvement de type tourniquet [50].
Des études ont démontré qu'un diapason, qui peut être utilisé comme capteur de détection de force, peut contrôler le passage de l'ADN à travers un nanopore à une vitesse inférieure au nanomètre [51, 57]. Dans une étude utilisant cet appareil intégré, comme le montre la figure 4d, une molécule d'ADN a été attachée à une pointe de sonde qui a été collée à une broche d'un diapason. Un système de nano-positionnement, qui possède une précision inférieure au nanomètre, a été utilisé pour maintenir le diapason [51]. La position de la pointe de la sonde peut être détectée par le capteur de force de rétroaction basé sur un diapason et contrôlée en manipulant le système de positionnement nanométrique. Cette vitesse de déplacement est 10 fois plus lente que celle de l'ADN manipulé par des pincettes optiques et 1000 fois plus lente que l'ADN passant librement à travers les nanopores à l'état solide [57]. Par rapport à l'AFM conventionnel, un diapason peut fournir un mouvement de balayage plus rapide et posséder une sensibilité à la force élevée lorsqu'il est immergé dans le liquide. En incorporant TFFS avec un nanopore, le courant ionique à travers un nanopore, la position de la pointe et l'amplitude de vibration de la pointe peuvent être mesurés simultanément pendant le passage d'une molécule d'ADN à travers le nanopore.
Sonde Si
Toutes les méthodes ci-dessus, à savoir les pinces magnétiques, les pinces optiques, l'AFM et le TFFS, doivent analyser la position des nanopores. Ils doivent localiser le nanopore dans la longueur effective de l'ADN pour s'assurer que l'ADN peut traverser le nanopore. L'adressage des nanopores est une partie importante de ces méthodes mais il est difficile [32] d'avoir connecté l'ADN à une grande surface d'une sonde en silicium (Fig. 5), plus grande que la surface de la puce, ce qui signifiait qu'ils pouvaient facilement insérer l'ADN immobilisé dans un nanopore sans rechercher l'emplacement du nanopore dans la membrane. La faisabilité de l'utilisation d'une sonde Si immobilisée par ADN et d'un contrôleur de position pour contrôler le mouvement de l'ADN dans et hors des nanopores a été démontrée. La difficulté de cette méthode est que la sonde Si est immergée dans la solution et reliée à l'ADN par le couplage peptidique. La densité de l'ADN à la surface de la sonde est difficile à contrôler, de sorte que plusieurs ADN traversant un nanopore en même temps sont susceptibles de se produire, affectant le courant de détection.
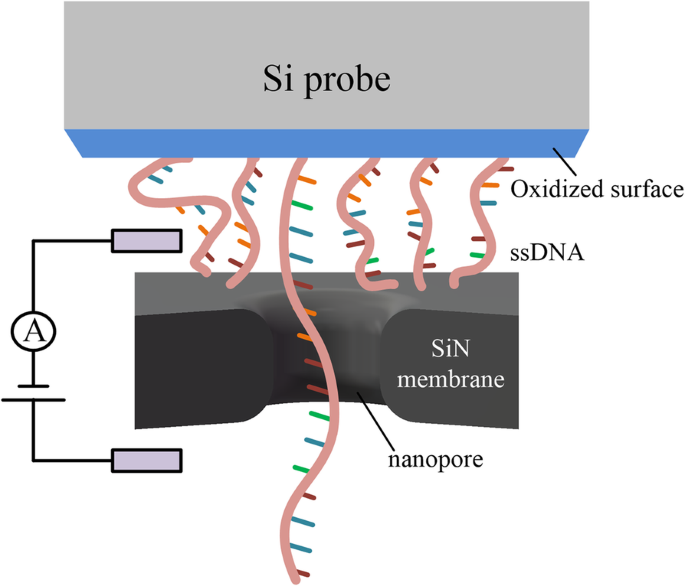
Schéma du système de sonde Si immobilisé par ADN utilisé pour la détection d'ADN nanopore [32]
Les pinces optiques, les pinces magnétiques, la microscopie à force atomique (AFM) et la détection de force basée sur le diapason (TFFS) peuvent détecter les forces et la position réelles de la molécule dans le nanopore, ce qui promet de contrôler le passage de l'ADN à travers les nanopores avec une vitesse appropriée. La difficulté d'adressage des nanopores est évitée en utilisant une sonde Si. De plus, l'utilisation d'un système à deux nanopores est une méthode réalisable pour contrôler et ralentir le passage de l'ADN à travers les nanopores. De plus, un nanopore de piège optique a un potentiel pour la détection d'ADN à l'avenir. Ici, nous avons résumé la vitesse de translocation de l'ADN d'un nanopore à l'état solide intégré à certaines méthodes de contrôle de l'ADN, telles qu'un système à deux nanopores, un piège optique nanopore, des pincettes optiques, des pincettes magnétiques, AFM, TFFS et sonde Si (tableau 1) .
Conclusion
Matériaux 2D monocouches, tels que le graphène, MoS2 , WS2 , et h-BN, sont probablement les matériaux les plus minces réalisables car ils sont aussi épais que l'espacement entre les nucléotides. Par rapport aux membranes à nanopores à semi-conducteur traditionnelles, les membranes 2D monocouches sont idéales pour les dispositifs à nanopores car elles présentent un rapport signal/bruit de courant ionique élevé et des régions de détection relativement grandes. Ils sont potentiellement éligibles pour réaliser le séquençage de l'ADN en combinant avec des pinces optiques, des pinces magnétiques, AFM, TFFS, une sonde Si, un système à deux nanopores ou un nanopore de piège optique. Cependant, avec ces techniques, plusieurs défis sont apparus, qui doivent être résolus avant la commercialisation du séquençage de l'ADN nanopore. Le premier d'entre eux se produit lorsque les billes ou les pointes de sonde sont proches d'un nanopore, lorsqu'il est plus difficile de discriminer les nucléotides d'ADN avec des signaux de courant ionique. Des poignées moléculaires ou d'autres molécules plus longues doivent être utilisées pour ajouter la longueur d'un brin d'ADN qui peut compenser l'effet sur le signal de courant provoqué par les billes ou les pointes. Deuxièmement, les réseaux de nanopores devraient être utilisés pour réaliser une détection parallèle et à haut débit, mais la technologie de détection parallèle n'est actuellement pas assez mature. Troisièmement, selon les méthodes de fabrication actuelles, il est difficile de fabriquer un système à deux nanopores et un système de nanopores à piège optique avec une précision et une reproductibilité élevées, ce qui est très important pour la détection d'ADN nanopore. Un faisceau d'ions hélium peut être la technologie clé pour résoudre ce problème [11, 58]. Ainsi, nous nous attendons à ce que le séquençage des nanopores de l'ADN continue d'être un objectif de recherche et puisse être intégré à davantage d'idées nouvelles et d'approches innovantes pour obtenir de faibles taux d'erreur, un enregistrement parallèle rapide et élevé et de longues longueurs de lecture allant jusqu'à 100 kilobases.
Disponibilité des données et des matériaux
Toutes les données générées ou analysées au cours de cette étude sont incluses dans cet article publié.
Abréviations
- 2D :
-
Deux dimensions
- 3D :
-
Matériaux tridimensionnels
- TMD :
-
Dichalcogénure de métal de transition en couches
- SNR :
-
Rapport signal/bruit
- PL :
-
Photoluminescence
- SIBA :
-
Contre-action auto-induite
- AFM :
-
Microscopie à force atomique
- TFFS :
-
Détection de force basée sur le diapason
Nanomatériaux
- Relais statiques
- Synthèse d'ADN
- Amazon Alexa Contrôle d'un Chromecast
- Conduire à travers l'écoute
- Blog :Détection de gènes par microarray
- Les scientifiques d'IBM mesurent le transfert de chaleur via des atomes uniques
- Sécuriser l'IoT par la tromperie
- Placage de trou
- Capteur de dioxyde de carbone à semi-conducteurs



